-
蛋白质的翻译后修饰(post-translational modification,PTM)是指在蛋白质翻译完成后,一些特殊的酶(修饰酶)通过共价键将特定的功能基团或分子添加到蛋白质的一个或多个氨基酸残基上的过程[1]。蛋白质的翻译后修饰会改变蛋白质的结构和功能,从而在个体发育、免疫反应、细胞代谢调控等生物过程中起到重要调控作用[2-3]。真核生物中已发现的蛋白质翻译后修饰类型多达400余种,不同类型的蛋白质翻译后修饰之间存在交互作用(cross-talk),即在同一蛋白质上相同或不同的位点上发生多种翻译后修饰,共同调控复杂的生物过程[4-5]。如Sun等[6]的研究发现,嗜水气单胞菌亚种(Aeromonas hydrophila)中的赖氨酸乙酰化和琥珀酰化可以发生在相同的赖氨酸残基上,并在关键的生物学通路(如碳代谢和TCA循环)中共同发挥调控作用。
蛋白质的乳酰化修饰发现于2019年,是一种由乳酸作为前体,在修饰酶的作用下,乳酰基团被连接到赖氨酸残基上的修饰过程。虽然发现的时间短,但大量的研究已表明,乳酰化修饰在癌症、个体发育等重大疾病和细胞过程中起重要作用,参与了碳氮代谢和能量代谢等生物过程的调控[7-10]。蛋白质的乳酰化修饰发生在化学修饰种类最多、最丰富的蛋白质翻译后修饰位点——赖氨酸残基上[11],意味着其与其他蛋白质翻译后修饰类型之间可能存在竞争修饰位点的交互作用。其中,乙酰化修饰的前体——乙酰辅酶A,与乳酸共同来源于糖酵解过程;研究表明,乳酰化修饰和乙酰化修饰存在共用一套修饰酶(乙酰化酶)和去修饰酶(去乙酰化酶)的现象。因此,推测乳酰化和乙酰化之间可能存在更紧密的交互作用[12-13]。
蛋白质后翻译修饰与其底物浓度息息相关,决定细胞内乳酸和乙酰辅酶A浓度的酶可能在乳酰化和乙酰化修饰中起重要作用。乳酸脱氢酶(lactate dehydrogenase,LDH)催化了丙酮酸与乳酸之间的可逆转化过程,能够决定丙酮酸的去向,进而决定了乳酸和乙酸辅酶A的浓度,从而影响胞内蛋白质乳酰化和乙酰化修饰水平。本研究前期的研究发现,在海洋模式生物三角褐指藻(Phaeodactylum tricornutum Bohlin)中,存在蛋白质乳酰化修饰的现象,但其乳酸脱氢酶——ptLDHA上并没有发生乳酰化修饰[14];而Chen等[15]的研究表明,ptLDHA上存在乙酰化修饰位点(K220),但ptLDHA的乙酰化修饰对其酶活的影响仍未清楚。有研究表明,人类和细菌的乳酸脱氢酶上均存在乙酰化修饰位点,二者乳酸脱氢酶的乙酰化修饰均能够调节酶的活性,从而调节胞内乳酸浓度[16-17];因此推测,ptLDHA的乙酰化修饰可能也能够通过酶活性的调节,从而在三角褐指藻乳酰化和乙酰化交互中起作用。
本研究以三角褐指藻cDNA为模板,克隆了ptLDHA基因序列,构建了原核表达载体pMBP-C-LDHA,转入BL21(DE3)菌株中进行原核表达,优化了诱导表达条件,并进行His标签融合蛋白的纯化和鉴定;为下一步构建其乙酰化修饰位点的定点突变;并探究修饰和去修饰状态对其酶活的影响奠定基础,有助于进一步揭示蛋白质翻译后修饰的交互作用在三角褐指藻代谢调控中的作用,对拓展藻类生物学研究及相关应用具有重要意义。
-
三角褐指藻取自于中国海洋大学微藻培养中心。培养基为人工海水f/2培养基[18],培养条件:光照强度为100 μmol·m−2·s−1,温度为22 ℃,光周期为12 h光照/12 h黑暗。
-
大肠杆菌(Escherichia coli)DH5α和大肠杆菌BL21(DE3)购于上海唯地生物技术有限公司(中国,上海);克隆载体pMD-19T购于宝日医生物技术有限公司(中国,北京);表达载体pMBP-C购于武汉淼灵生物科技有限公司(中国,武汉)。
-
植物RNA提取试剂盒、琼脂糖凝胶回收试剂盒、质粒DNA小量提取试剂盒均购于Omega Bio-Tek(美国,诺克罗斯);反转录试剂盒、DNA Maker DL2000、T4 DNA Ligase均购于南京诺唯赞生物科技有限公司(中国,南京);限制性内切酶BamH I、Nhe I购于赛默飞世尔科技公司(中国,上海);彩色预染蛋白maker购于武汉博士德生物工程有限公司(中国,武汉);Ni-IDA Agarose 6FF购于生工生物工程股份有限公司(中国,上海);LDHA多克隆抗体定制于金斯瑞生物科技股份有限公司(中国,南京);二抗HRP-conjugated Affinipure Goat Anti-Rabbit IgG 购于上海金畔生物科技有限公司(中国,上海);ECL化学发光试剂盒购于爱必信生物科技有限公司(中国,上海);其他化学药品均为国产分析纯。
-
提取三角褐指藻的总RNA,反转录为cDNA,以cDNA作为模板,用带BamH I、Nhe I酶切位点的引物LDHA-R和LDHA-F(表1)进行PCR扩增,获得目的基因DNA序列;PCR扩增程序如下:95 ℃预变性5 min,95 ℃变性15 s,56 ℃退火15 s,72 ℃延伸45 s,共35个循环,之后在72 ℃延伸7 min。将PCR产物进行琼脂糖凝胶电泳,并切胶回收目的基因片段。
表 1 本研究中使用的引物序列
Table 1. Primer sequences
引物名称
Primer引物序列(5′−3′)
Sequence (5′−3′)LDHA-F CGGGATCCATGGAGTTCACGACAGAG LDHA-R CGGCTAGCACTCTCTACAGCTGGGAT M13F GTAAAACGACGGCCAGT M13R CAGGAAACAGCTATGAC pMBP-C-LDHA-F TTGACAATTAATCATCGGCTCG pMBP-C-LDHA-R ACTGGCCGTCGTTTTAC 注:下划线分别代表BamH Ⅰ和Nhe Ⅰ引物的酶切位点。 Note:The underlined represent the restriction sites for BamH Ⅰ and Nhe Ⅰ in the primers. -
采用T4DNA Ligase将回收的目的基因片段与克隆载体pMD-19T进行连接,然后转化到大肠杆菌,具体步骤如下:取10 μL连接产物,加入大肠杆菌DH5ɑ感受态细胞中,混合混匀,冰浴 25 min,42 ℃热激45 s,冰浴2 min,加入700 μL的LB培养基,放入摇床中(37 ℃,180 r·min−1)培养1 h,随后离心(5 000 r·min−1)5 min,弃600 μL上清液;将剩余菌液混匀;取100 μL菌液,在含有100 mg·L−1氨苄青霉素的固体培养基上均匀涂布,在37 ℃恒温培养箱中过夜培养;挑取单菌落于含有100 mg·L−1氨苄青霉素的液体培养基中,利用M13F/M13R引物对单一菌落进行PCR验证(表1)。将PCR产物进行琼脂糖凝胶电泳,挑选条带大小符合预期的菌株,提取质粒送生工生物工程有限公司测序。经测序确认的质粒命名为pMD-19T-LDHA。
-
将测序确认的pMD-19T-LDHA质粒与原核表达载体pMBP-C分别以BamH I 和Nhe I 进行双酶切后,回收获得酶切后的目的基因片段和pMBP-C空载体。随后,利用T4 DNA Ligase将回收产物连接,构建重组表达质粒。将重组质粒转化至大肠杆菌DH5α感受态细胞中,随后取100 μL菌液,在100 mg·L−1氨苄青霉素的固体培养基上均匀涂布,挑选单一菌落,利用pMBP-C-LDHA-F/pMBP-C-LDHA-R引物对单一菌落PCR验证(表1)。将PCR产物进行琼脂糖凝胶电泳,挑选条带大小符合预期的菌株,提取质粒送生工生物工程有限公司测序。经测序确认的质粒命名为pMBP-C-LDHA。
-
将pMBP-C-LDHA质粒转化至大肠杆菌BL21(DE3),命名为pMBP-C-LDHA-BL21,将菌液均匀涂布在含有100 mg·L−1氨苄青霉素的固体培养基上,挑选单一菌落,进行PCR验证。挑选正确的菌落接种于含有100 mg·L−1氨苄青霉素的液体培养基中,37 ℃培养 12 h。随后,取 10 μL菌液接种至 5 mL 含氨苄青霉素的 LB 培养基中,在37 ℃摇床中培养,直至菌液OD600≈0.6。接着,在不同IPTG浓度和温度条件下诱导表达,具体条件见表2。诱导结束后,取1 mL菌液,12 000 r·min−1离心1 min,弃上清后加入200 μL无菌水,12 000 r·min−1离心 1 min,弃上清。随后,将菌体用 80 μL无菌水重悬,加入 20 μL 5×蛋白上样缓冲液混匀后,沸水浴 10 min。最后,进行SDS-PAGE电泳检测。
表 2 不同温度和IPTG浓度的诱导表达条件
Table 2. Inducing conditions with different temperatures and IPTG concentrations
温度/ ℃
Temperature诱导时间/h
Inducing timeIPTG浓度/(mmol·L−1)
IPTG concentration16 24 0、0.2、0.5、1.0 30 6 0、0.2、0.5、1.0 37 4 0、0.2、0.5、1.0 -
根据探索出的LDHA重组蛋白的诱导最佳条件,在IPTG浓度为0.2 mmol·L−1,16 ℃ 24 h条件下大量诱导LDHA重组蛋白的表达。将诱导后的菌液在4 ℃,8 000 r·min−1离心5 min,弃上清液,加入5 mL蛋白裂解液重悬沉淀,放入液氮中5 min,随后,将冷冻后的裂解液在4 ℃冰箱中过夜,使样品液自然融化。将融化后的样品,在冰水浴条件下进行超声波破碎,设置功率为180 w,震杆为2,开关时间为2 s与4 s,持续30 min。样品破碎后,在4 ℃条件下,以8 000 r·min−1离心30 min,收集上清液蛋白。随后,将离心后的包涵体蛋白加入1 mL缓冲液,混匀后在水平摇床上摇动4 h,充分裂解包涵体蛋白。接着,在4 ℃条件下,以8 000 r·min−1离心30 min,收集的上清即为包涵体蛋白。将收集的上清液中的可溶性蛋白和包涵体蛋白分别进行SDS-PAGE电泳检测。
-
LDHA重组蛋白主要存在于破碎菌体的上清液中,因此,用收集的上清液进行镍亲和层析柱的蛋白纯化。首先,用5倍柱体积的结合液洗涤柱子3次,然后将上清液上样,在4 ℃过夜;次日,依次使用浓度为0、20、30、150、250和500 mmol·L−1的咪唑洗脱液洗脱蛋白,每个浓度洗脱3次;收集不同咪唑浓度下洗脱的蛋白,进行SDS-PAGE分析,检测重组蛋白条带的位置,从而确定最佳的纯化条件。
-
纯化后的LDHA重组蛋白经SDS-PAGE凝胶电泳后,采用湿式转印法进行转膜,设置电压为100 V转印1.5 h,转膜完成后,将NC膜用5%(w/w)脱脂奶粉在室温下封闭1.5 h,1×Tris-盐酸缓冲液TBST重复洗涤5次,每次间隔5 min。洗膜结束后,将NC膜放入用5%(w/w)脱脂奶粉稀释以1:1 000稀释过的LDHA多克隆抗体,4 ℃孵育过夜。次日,用TBST重复洗涤5次,每次间隔5 min;加入用TBST 缓冲液以1:10 000稀释过的HRP标记绵羊抗兔抗体,室温孵育1 h,随后,用TBST洗涤5次,每次间隔5 min。最后,在NC膜上均匀滴加ECL化学发光液,在BIORAD成像系统中进行显影,观察并记录结果。
-
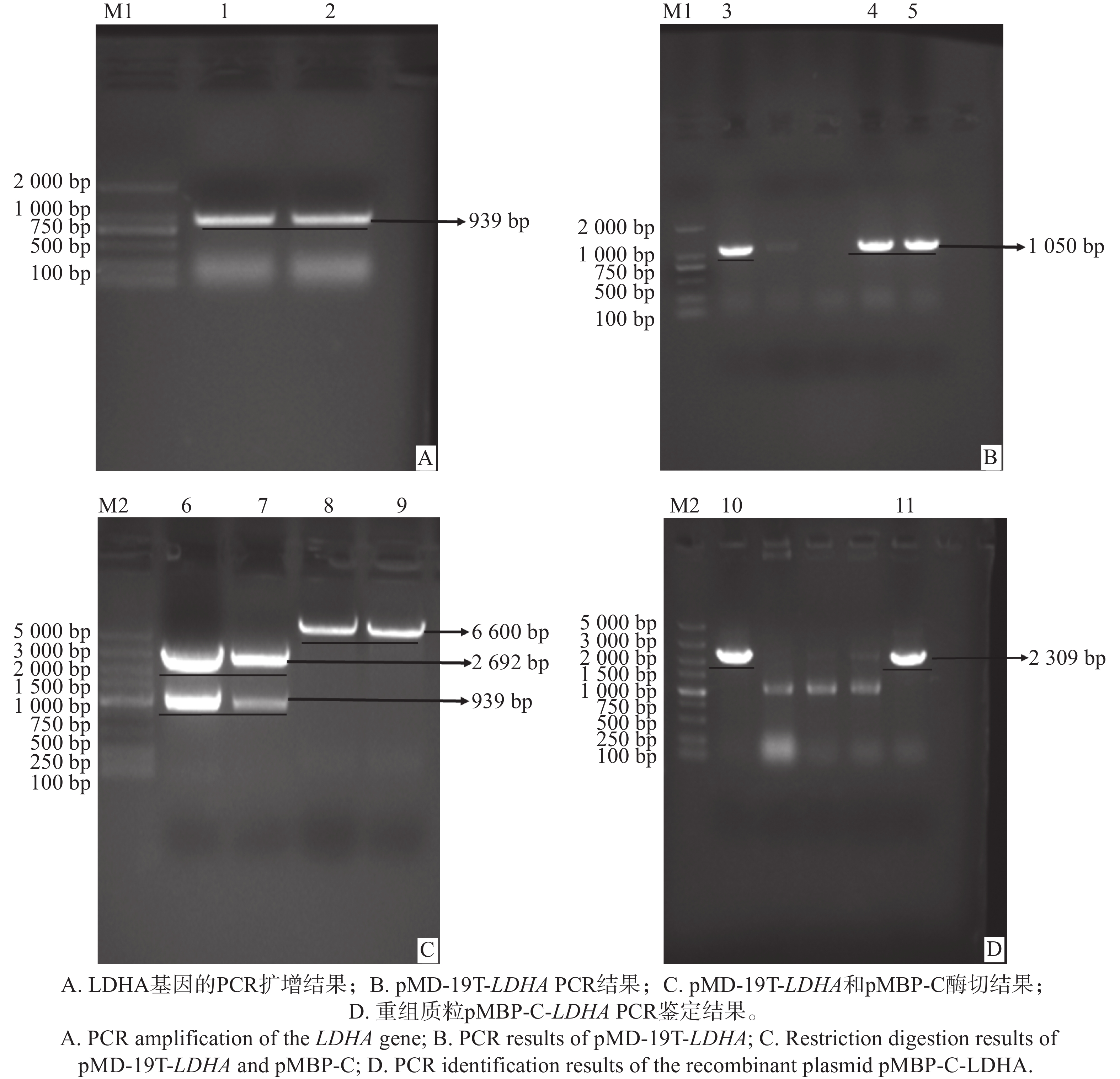
以三角褐指藻cDNA为模板进行PCR扩增,得到约939 bp大小的产物(图1-A),与目的基因片段的预期大小一致。该PCR产物克隆至pMD-19T载体后,转化至大肠杆菌DH5ɑ感受态细胞,电泳检测结果显示多个阳性克隆,呈现出约1 050 bp的条带,和预期片段大小一致,对筛选出的阳性克隆测序,结果与NCBI数据库(GenBank登录号:NW_002238036.1)中的报道序列基本一致(图1-B)。
-
通过酶切克隆质粒pMD-19T-LDHA和空载体pMBP-C,得到LDHA目的基因片段和pMBP-C载体的双酶切产物。电泳结果显示,在预期位置出现特异性条带,目的基因约为939 bp,pMD-19T空载体为2 692 bp,pMBP-C空载体为6 600 bp(图1-C),表明双酶切成功。将目的基因与pMBP-C双酶切产物连接,构建得到表达载体pMBP-C-LDHA,并转化至大肠杆菌DH5ɑ感受态细胞中。电泳检测结果显示在预期位置出现约2 309 bp的条带(图1-D),符合预期目标片段大小。挑选阳性克隆测序,结果显示,序列包含目的基因序列和质粒上部分序列,无移码突变,表明含有目的基因LDHA的表达质粒构建成功。
-
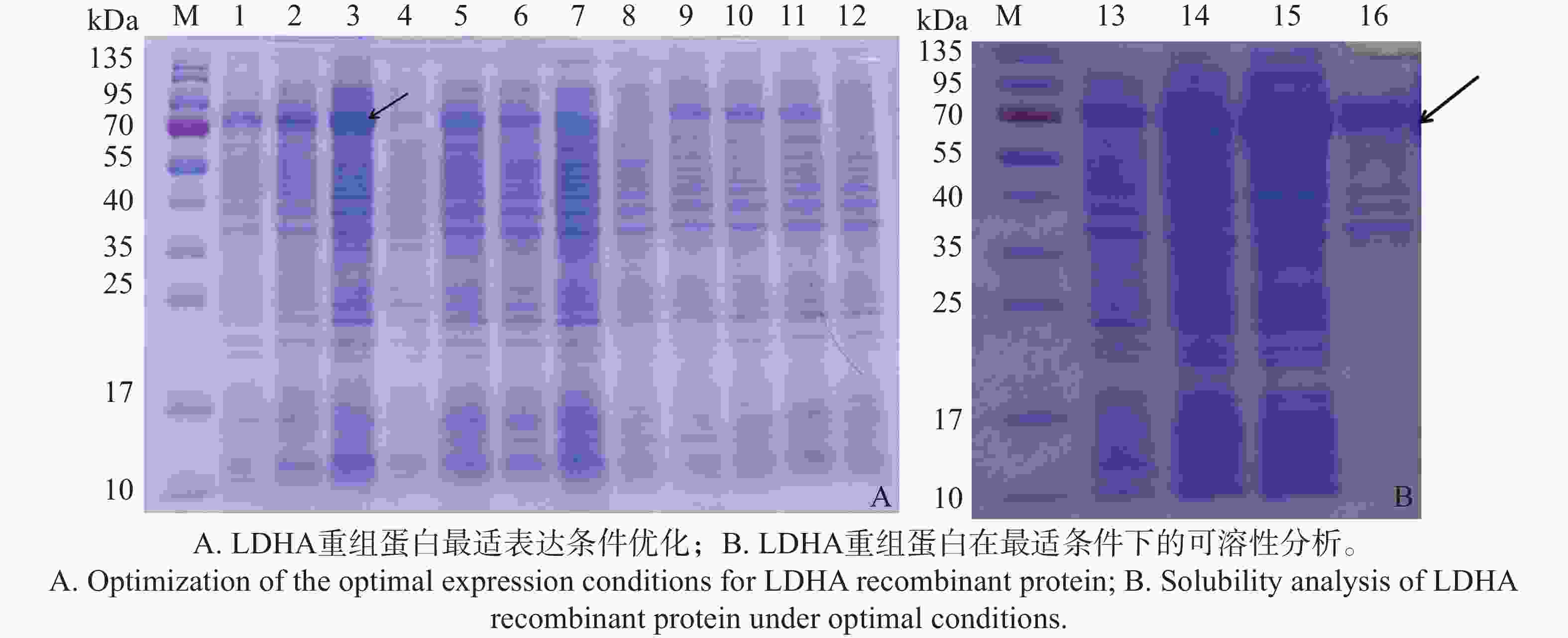
原核表达蛋白会受到诱导温度、诱导时间和IPTG浓度的影响。本实验培养至菌液OD600≈0.6时,在其中加入IPTG至终浓度分别为0.2、0.5和1.0 mmol·L−1,并在180 r·min−1,16、30和37 ℃下,分别培养24、8和4 h。结果显示,在16 ℃、IPTG为0.2 mmol·L−1下,诱导表达24 h时,表达量最高,为最佳表达条件(图2-A)。在此条件下,目标蛋白的表达量显著高于在其他温度(30、37 ℃)下的表达水平,且较低的IPTG浓度有助于避免高浓度IPTG对细胞生长的抑制作用,为后续蛋白的大规模表达与纯化实验提供了可靠保障,确保了目标蛋白的高效表达以及其良好的可纯化。
-
在最佳诱导条件下诱导菌液表达LDHA重组蛋白,并收集约100 mL菌体进行超声波破碎后,分别以上清液和沉淀制备样品,进行SDS-PAGE电泳分析。结果表明,在16 ℃、IPTG为0.2 mmol·L−1诱导表达24 h后,LDHA重组蛋白主要存在于上清液中(图2-B),进一步证实了在16 ℃、0.2 mmol·L−1 IPTG诱导下,LDHA重组蛋白的表达不仅具有较高的产量,而且大部分以溶解状态存在,为后续的蛋白纯化和功能研究提供了有利条件。
-
由于pMBP-C-LDHA诱导表达的蛋白带有组氨酸标签,将表达后的上清利用镍柱亲和层析进行纯化。SDS-PAGE结果(图3-A)显示,在使用咪唑浓度分别为150、250和500 mmol·L−1时,都能在约78 kDa处观察到明显的目的蛋白条带。其中,250 mmol·L−1浓度的咪唑洗脱获得的LDHA重组蛋白量较多且杂带较少,结果表明,250 mmol·L−1咪唑浓度能够有效地洗脱LDHA重组蛋白。此外,通过Western Blot检测,确认洗脱下来的蛋白条带与预期大小一致(图3-B)。综上所述,通过镍亲和层析和Western Blot验证,成功纯化得到了LDHA重组蛋白,为后续的功能研究和结构分析奠定了基础。
-
在蛋白质翻译后修饰(PTM)研究中,乙酰化和乳酰化修饰因其在细胞代谢调控中的关键作用而备受关注。乳酸脱氢酶(LDH)作为无氧糖酵解中的关键酶,负责催化乳酸和丙酮酸的相互转化,是维持细胞代谢平衡的重要调节因子。本研究的前期研究表明,尽管三角褐指藻中存在蛋白质乳酰化修饰的现象,但乳酸脱氢酶ptLDHA并未发生乳酰化修饰[14]。然而,已有研究指出,LDH在多种生物体内的乙酰化修饰位点能够显著调节其酶活性[6, 12],暗示乙酰化修饰在乳酸代谢中可能发挥着重要作用。因此,本研究推测,ptLDHA的乙酰化修饰可能通过影响其酶活性,在乳酰化与乙酰化修饰之间形成交互作用,从而进一步加深对三角褐指藻代谢调控机制的理解。
原核基因表达是研究蛋白质结构与功能的基础方法之一。本研究以三角褐指藻的cDNA为模板,成功克隆了LDHA基因,并分别构建了克隆载体pMD-19T和表达载体pMBP-C-LDHA。通过PCR检测转化的阳性克隆,验证了载体构建的成功,预期的LDHA基因片段大小为939 bp。然而,PCR结果也分别显示了约1 050 bp和2 309 bp的条带。这一现象可以通过引物设计和扩增机制进行解释。所使用的引物分别位于空质粒pMD-19T和pMBP-C的上下游区域,因此,扩增的产物为质粒上下游区域与LDHA基因片段的组合。具体而言,pMD-19T的上下游区域扩增片段大小为109 bp,加上LDHA基因的939 bp,总大小约为
1050 bp;而pMBP-C的上下游区域扩增片段大小为1367 bp,加上LDHA基因的942 bp,总大小为2 309 bp。上述结果进一步验证了克隆载体和表达载体均正确地包含了LDHA基因,为后续的原核表达和功能研究奠定了基础。重组蛋白的结构能够影响其原核表达的形式,从而影响下游的蛋白结构和功能的研究。赵蕾等[20]将猪TGFⅡR胞外域蛋白基因序列插入原核表达载体pET-32a(+)中,所得重组蛋白为变性蛋白,需经过复性处理才能恢复其功能。同样,刘晓玥等[21]通过构建原核表达载体pET-29b(+)-ZmERF061,分析了玉米抗病基因ZmERF061的表达特性,发现ZmERF061-His融合蛋白主要以包涵体形式存在,也需要复性处理才能恢复其功能。与此不同,pMBP-C载体含有MBP和His标签,MBP能够与目的蛋白融合表达,从而显著提高蛋白的稳定性和溶解性;His标签有助于后续的分离和纯化,便于进一步的酶活分析[19]。Chen等[15]为了研究三角褐指藻ptACSL1的乙酰化修饰对酶活性的影响,采用pMBP-C载体构建了ptACSL1的原核表达载体,显著提高了蛋白的溶解性与纯化效率。研究人员对ptACSL1中AMP结合域的乙酰化修饰位点K407和K425进行了定点突变,分别模拟乙酰化状态(K407Q,K425Q)和非乙酰化状态(K407R,K425R)。突变实验表明,K407和K425的突变均导致LACS活性下降,K407Q和K425Q的酶活性略高于K407R和K425R,提示赖氨酸乙酰化可能对酶活性具有正向调控作用。借鉴这一成功案例及其实验方法,本研究采用pMBP-C载体构建了三角褐指藻LDHA的原核表达载体,并通过融合MBP标签成功实现了LDHA蛋白的高效表达,在16 ℃的低温条件下,使用0.2 mmol·L−1的IPTG浓度并诱导24 h,可获得大量的可溶性重组蛋白。进一步的可溶性分析结果显示,LDHA重组蛋白主要以可溶性形式存在。这种表达模式不仅有助于维持蛋白的非变性状态,同时也为后续的纯化和功能研究提供了有利条件。通过镍柱亲和层析法对融合蛋白进行纯化,利用His标签实现高效分离,成功获得了高纯度的LDHA重组蛋白。Western blot验证进一步确认了所纯化蛋白的特异性和正确性,证明了LDHA重组蛋白成功表达和纯化。这一策略为后续深入研究ptLDHA的生物学功能及其乙酰化修饰对活性的调控机制奠定了坚实基础。
尽管本研究成功克隆并表达了LDHA基因,并验证了其重组蛋白表达和纯化的可行性,但目前的研究尚未深入探讨其翻译后修饰对酶活性的具体影响,且蛋白在体外和体内的功能一致性还未能得到证实。未来研究需重点关注定点突变实验,以进一步确认乙酰化修饰对ptLDHA酶活性的调控作用,并验证修饰与去修饰状态对其功能的影响。此外,还应进一步验证表达蛋白在体外和体内的功能一致性,并对蛋白质修饰状态进行深入确认。这将为全面理解ptLDHA的生物学功能提供关键支持,并揭示蛋白质翻译后修饰的交互作用在三角褐指藻代谢调控中的潜在作用,为拓展藻类生物学研究及相关应用提供重要理论依据。
通过低温诱导表达和镍柱亲和纯化策略,本研究高效表达并纯化了三角褐指藻的LDHA的重组蛋白,为下一步研究乙酰化对ptLDHA活性的影响,进一步研究ptLDHA在三角褐指藻蛋白质乳酰化和乙酰化修饰交互中可能的作用及其生物学功能奠定了基础。
Prokaryotic expression and purification identification of lactate dehydrogenase from Phaeodactylum tricornutum Bohlin
-
摘要: 为了探讨三角褐指藻(Phaeodactylum tricornutum Bohlin)中乳酸代谢的关键酶——乳酸脱氢酶(ptLDHA)上的乙酰化修饰位点及其在乳酰化和乙酰化交互作用中的潜在作用,以三角褐指藻cDNA为模板克隆了ptLDHA基因序列,并构建了原核表达载体pMBP-C-LDHA,随后将其转入大肠杆菌BL21(DE3)中进行诱导表达。经优化,在 16 ℃、0.2 mmol·L−1 IPTG 诱导 24 h 条件下,ptLDHA蛋白成功表达且主要为可溶性蛋白。利用His标签进行融合蛋白的纯化,并用ptLDHA多克隆抗体进行 Western blot 鉴定,在约 78 kDa处获得单一蛋白条带,证实纯化蛋白为ptLDHA。本研究成功实现了ptLDHA在原核细胞中的表达与纯化,为后续构建乙酰化修饰位点的定点突变并探究修饰与去修饰状态对酶活性的影响奠定了基础。Abstract: An attempt was made to investigate the expression of lactate dehydrogenase (ptLDHA), a key enzyme in lactate metabolism, and to explore the acetylation modification sites on ptLDHA and their potential role in the interplay between lactylation and acetylation modifications in Phaeodactylum tricornutum Bohlin. The ptLDHA gene sequence was first cloned from P. tricornutum Bohlin cDNA, and then a prokaryotic expression vector, pMBP-C-LDHA, was constructed. The recombinant plasmid was then transferred into Escherichia coli BL21 (DE3) for induced expression. Under optimized conditions (16 ℃, 0.2 mmol·L−1 IPTG for 24 hours), ptLDHA protein was successfully expressed, predominantly in a soluble form. The fusion protein was purified using His-tag affinity chromatography and identified by Western blot with ptLDHA polyclonal antibodies. A single protein band at approximately 78 kDa was observed, confirming that the purified protein was ptLDHA. The expression and purification of ptLDHA in prokaryotic cells was successfully established, laying foundations for subsequent site-directed mutagenesis of acetylation modification sites and for investigation of the effects of modification and demodification on enzyme activity.
-
Key words:
- Phaeodactylum tricornutum Bohlin /
- LDHA /
- prokaryotic expression /
- protein purification
-
图 2 LDHA重组蛋白表达优化与可溶性分析
注:M,彩色预染蛋白Marker;1)16 ℃,1.0 mmol·L−1 IPTG诱导24 h;2)16 ℃,0.5 mmol·L−1 IPTG诱导 24 h;3)16 ℃,0.2 mmol·L−1 IPTG诱导24 h;4)16 ℃,0 mmol·L−1 IPTG诱导 24 h;5)30 ℃,1.0 mmol·L−1 IPTG诱导8 h;6)30 ℃,0.5 mmol·L−1 IPTG诱导8 h;7)30 ℃,0.2 mmol·L−1 IPTG诱导8 h;8)30 ℃,0 mmol·L−1 IPTG诱导8 h; 9)37 ℃,1.0 mmol·L−1 IPTG诱导4 h;10)37 ℃,0.5 mmol·L−1 IPTG诱导4 h;11)37 ℃,0.2 mmol·L−1 IPTG诱导4 h;12)37 ℃,0 mmol·L−1 IPTG诱导 4 h;13)未诱导全蛋白;14)16 ℃,0.2 mmol·L−1 IPTG 24 h,全蛋白;15)16 ℃,0.2 mmol·L−1 IPTG,24 h,上清液;16)16 ℃,0.2 mmol·L−1 IPTG,24 h,沉淀。
Fig. 2 Expression optimization and solubility analysis of LDHA recombinant protein
Note: M, Protein Marker; 1) 16 ℃, 1 mmol·L−1 IPTG induction for 24 h; 2) 16 ℃, 0.5 mmol·L−1 IPTG induction for 24 h; 3) 16 ℃, 0.2 mmol·L−1 IPTG induction for 24 h; 4) 16 ℃, 0 mmol·L−1 IPTG induction for 24 h; 5) 30 ℃, 1 mmol·L−1 IPTG induction for 8 h; 6) 30 ℃, 0.5 mmol·L−1 IPTG induction for 8 h; 7) 30 ℃, 0.2 mmol·L−1 IPTG induction for 8 h; 8) 30 ℃, 0 mmol·L−1 IPTG induction for 8 h; 9) 37 ℃, 1.0 mmol·L−1 IPTG induction for 4 h; 10) 37 ℃, 0.5 mmol·L−1 IPTG induction for 4 h; 11) 37 ℃, 0.2 mmol·L−1 IPTG induction for 4 h; 12) 37 ℃, 0 mmol·L−1 IPTG induction for 4 h; 13) Samples were not induced; 14) 16 ℃, 0.2 mmol·L−1 IPTG induction for 24 h, Total Protein; 15) 16 ℃, 0.2 mmol·L−1 IPTG induction for 24 h, supernatant; 16) 16 ℃, 0.2 mmol·L−1 IPTG induction for 24 h, precipitate.
图 3 LDHA重组蛋白的纯化与Western Blot检测
注:M,彩色预染蛋白Marker;1)未过柱蛋白;2)0 mmol·L−1咪唑洗脱液;3)20 mmol·L−1咪唑洗脱液;4)30 mmol·L−1咪唑洗脱液;5)150 mmol·L−1咪唑洗脱液;6)250 mmol·L−1咪唑洗脱液;7)500 mmol·L−1咪唑洗脱液;8)纯化的LDHA蛋白与多克隆抗体的反应条带。
Fig. 3 Purification and Western Blot Detection of LDHA Recombinant Protein
Note: M. Color pre-stained protein marker; 1) Unbound protein; 2) 0 mmol·L−1 imidazole elution; 3) 20 mmol·L−1 imidazole elution; 4) 30 mmol·L−1zole elution; 5) 150 mmol·L−1 imidazole elution; 6) 250 mmol·L−1 imidazole elution; 7) 500 mmol·L−1 imidazole elution; 8) Purified LDHA protein reacted with polyclonal antibody.
表 1 本研究中使用的引物序列
Table 1 Primer sequences
引物名称
Primer引物序列(5′−3′)
Sequence (5′−3′)LDHA-F CGGGATCCATGGAGTTCACGACAGAG LDHA-R CGGCTAGCACTCTCTACAGCTGGGAT M13F GTAAAACGACGGCCAGT M13R CAGGAAACAGCTATGAC pMBP-C-LDHA-F TTGACAATTAATCATCGGCTCG pMBP-C-LDHA-R ACTGGCCGTCGTTTTAC 注:下划线分别代表BamH Ⅰ和Nhe Ⅰ引物的酶切位点。 Note:The underlined represent the restriction sites for BamH Ⅰ and Nhe Ⅰ in the primers. 表 2 不同温度和IPTG浓度的诱导表达条件
Table 2 Inducing conditions with different temperatures and IPTG concentrations
温度/ ℃
Temperature诱导时间/h
Inducing timeIPTG浓度/(mmol·L−1)
IPTG concentration16 24 0、0.2、0.5、1.0 30 6 0、0.2、0.5、1.0 37 4 0、0.2、0.5、1.0 -
[1] Mann M, Jensen O N. Proteomic analysis of post-translational modifications [J]. Nature Biotechnology, 2003, 21(3): 255−261. https://doi.org/10.1038/nbt0303-255 doi: 10.1038/nbt0303-255 [2] Olsen J V, Mann M. Status of large-scale analysis of post-translational modifications by mass spectrometry [J]. Molecular & Cellular Proteomics, 2013, 12(12): 3444−3452. [3] Khoury G A, Baliban R C, Floudas C A. Proteome-wide post-translational modification statistics: frequency analysis and curation of the Swiss-prot database [J]. Scientific Reports, 2011, 1: 90. https://doi.org/10.1038/srep00090 doi: 10.1038/srep00090 [4] Huang Y, Xu B, Zhou X, et al. Systematic characterization and prediction of post-translational modification cross-talk [J]. Molecular & Cellular Proteomics, 2015, 14(3): 761−770. [5] 陈艳梅. 蛋白质翻译后修饰之间的互作关系及其协同调控机理[J]. 生物技术通报, 2024, 40(2): 1−8. [6] Sun L, Yao Z, Guo Z, et al. Comprehensive analysis of the lysine acetylome in Aeromonas hydrophila reveals cross-talk between lysine acetylation and succinylation in LuxS [J]. Emerging Microbes & Infections, 2019, 8(1): 1229−1239. [7] Lund J, Aas V, Tingstad R H, et al. Utilization of lactic acid in human myotubes and interplay with glucose and fatty acid metabolism [J]. Scientific Reports, 2018, 8(1): 9814. https://doi.org/10.1038/s41598-018-28249-5 doi: 10.1038/s41598-018-28249-5 [8] Cai X, Ng C P, Jones O, et al. Lactate activates the mitochondrial electron transport chain independently of its metabolism [J]. Molecular Cell, 2023, 83(21): 3904−3920. https://doi.org/10.1016/j.molcel.2023.09.034 doi: 10.1016/j.molcel.2023.09.034 [9] Duan J, Li C, Zheng Y, et al. Characterization of exogenous lactate addition on the growth, photosynthetic performance, and biochemical composition of four bait microalgae strains [J]. Journal of Applied Microbiology, 2023, 134(11): lxad259. https://doi.org/10.1093/jambio/lxad259 doi: 10.1093/jambio/lxad259 [10] Zhang D, Tang Z, Huang H, et al. Metabolic regulation of gene expression by histone lactylation [J]. Nature, 2019, 574(7779): 575−580. https://doi.org/10.1038/s41586-019-1678-1 doi: 10.1038/s41586-019-1678-1 [11] Wang Z A, Cole P A. The chemical biology of reversible lysine post-translational modifications [J]. Cell Chemical Biology, 2020, 27(8): 953−969. https://doi.org/10.1016/j.chembiol.2020.07.002 doi: 10.1016/j.chembiol.2020.07.002 [12] Stacpoole P W, Dirain C O. The pyruvate dehydrogenase complex at the epigenetic crossroads of acetylation and lactylation [J]. Molecular Genetics and Metabolism, 2024, 143(1/2): 108540. [13] Yang Y, Luo N, Gong Z, et al. Lactate and lysine lactylation of histone regulate transcription in cancer [J]. Heliyon, 2024, 10(21): e38426. https://doi.org/10.1016/j.heliyon.2024.e38426 doi: 10.1016/j.heliyon.2024.e38426 [14] Huang A, Li Y, Duan J, et al. Metabolomic, proteomic and lactylated proteomic analyses indicate lactate plays important roles in maintaining energy and C: N homeostasis in Phaeodactylum tricornutum [J]. Biotechnology for Biofuels and Bioproducts, 2022, 15(1): 61. https://doi.org/10.1186/s13068-022-02152-8 doi: 10.1186/s13068-022-02152-8 [15] Chen Z, Luo L, Chen R, et al. Acetylome profiling reveals extensive lysine acetylation of the fatty acid metabolism pathway in the diatom Phaeodactylum tricornutum [J]. Molecular & Cellular Proteomics, 2018, 17(3): 399−412. [16] Zhao D, Zou S W, Liu Y, et al. Lysine-5 acetylation negatively regulates lactate dehydrogenase A and is decreased in pancreatic cancer [J]. Cancer Cell, 2013, 23(4): 464−476. https://doi.org/10.1016/j.ccr.2013.02.005 doi: 10.1016/j.ccr.2013.02.005 [17] Liu M, Huo M, Liu C, et al. Lysine acetylation of Escherichia coli lactate dehydrogenase regulates enzyme activity and lactate synthesis [J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 966062. https://doi.org/10.3389/fbioe.2022.966062 doi: 10.3389/fbioe.2022.966062 [18] Hayashi S I, Lin E C C. Purification and properties of glycerol kinase from Escherichia coli [J]. Journal of Biological Chemistry, 1967, 242(5): 1030−1035. https://doi.org/10.1016/S0021-9258(18)96228-9 doi: 10.1016/S0021-9258(18)96228-9 [19] Young C L, Britton Z T, Robinson A S. Recombinant protein expression and purification: a comprehensive review of affinity tags and microbial applications [J]. Biotechnology Journal, 2012, 7(5): 620−634. https://doi.org/10.1002/biot.201100155 doi: 10.1002/biot.201100155 [20] 赵蕾, 李辉, 覃水平, 等. 猪TGF-βⅡ型受体胞外域的表达及生物活性验证[J]. 江苏农业学报, 2024, 40(7): 1268−1275. https://doi.org/10.3969/j.issn.1000-4440.2024.07.013 doi: 10.3969/j.issn.1000-4440.2024.07.013 [21] 刘晓玥, 董庆文, 许赢, 等. 玉米ZmERF061基因原核表达载体构建及蛋白表达分析[J/OL]. 分子植物育种, (2024-03-21)[2024-12-13]. http://link.cnki.net/urlid/46.1068.s.20240320.1148.011. -




 下载:
下载: