-
水稻(Oryza sativa)在早期群体建立阶段的根系生长与邻株间互作,往往决定后续植株对营养资源的获取效率、竞争能力及最终产量[1-2]。在现代水稻生产体系中,为追求高产与管理效率,单一品种在大面积田块中的长期种植仍占主导地位。然而,这种高度同质化的种植模式在提高生产效率的同时,也显著降低了群体内部的遗传多样性,使稻田生态系统对病虫害侵染和环境波动的缓冲能力下降。大量研究表明,品种单一化是稻瘟病等重大病害周期性暴发的重要诱因之一,而通过提高群体内部的遗传异质性,可有效削弱病原菌传播效率、延缓抗性基因失效并降低对化学农药的依赖[3-4]。因此,品种多样化配置被认为是提升稻田生态稳定性和推动病虫害绿色防控的重要生态学途径之一[5-6]。
在实际农业生产中,同一种作物的不同品种常因播种混杂、补苗行为、品种更替以及群体生长差异等因素,在田间以空间接近甚至混合分布的方式共存。这种不同基因型在幼苗期“近距离共存”的格局,为品种间在地下尺度上发生直接互作提供了现实基础。与跨物种间作系统相比,同种作物不同品种之间的互作更加细微而复杂:由于其根系结构、营养需求和生理特性高度相似,竞争关系往往不完全由资源占据所决定,而更可能受根系信号、化学识别以及微弱生态位差异的调控。已有研究表明,同种内的基因型差异可显著影响作物对养分资源的获取与利用效率。例如,不同水稻基因型在氮等营养元素的吸收和利用能力上存在明显差异[7]。此外,植物根系之间的相互作用被认为可通过改变邻体的养分获取和根系生长策略而影响群体表现,这一过程可能与根系分泌物介导的化学信号和化感作用有关[8-9]。然而,目前的研究多集中于群体尺度的产量稳定性或竞争格局,对早期根系层面的互作机制关注不足。根系分泌物被认为是塑造植物地下互作关系的关键介质,其作为植物间化学信息传递的重要载体,可在无明显资源竞争的条件下直接影响邻株的根系结构、生理状态或代谢特征,从而改变竞争结果[10-11]。在跨物种体系中,化感作用及其化学生态基础已得到广泛证实,例如水稻对稗草的抑制作用,母育酚被鉴定为关键效应物质;向日葵、黑胡桃等植物通过释放萜类、酚酸类或醌类化合物调控邻近植物生长,已成为经典研究案例[12-14]。有研究指出,植物根系竞争并不完全由资源耗竭驱动,根系间可能存在通过干扰或信号作用形成的非对称竞争关系[15]。这一观点为理解同种作物不同基因型之间潜在的化学生态互作提供了理论基础。然而,这种同种内化感作用是否普遍存在,其调控是否具有明确方向性,以及其代谢基础为何,仍缺乏在可控条件下的系统解析。
更为关键的是,幼苗期是水稻群体结构和竞争格局建立的关键窗口期,根际互作在这一阶段可能产生持续性的“印记效应”,并在后续生育过程中影响植株优势建立、资源利用策略乃至群体稳定性[1]。同时,根系互作还可能通过改变根际化学环境与信号网络,间接影响病原微生物的定殖与扩散,从而为品种多样化在病害生态调控中的作用提供机制基础[16]。然而,迄今为止,鲜有研究在高度可控条件下,同时整合“品种间根系表型变化”与“根系分泌物代谢组变化”,以解析同种不同品种之间根际互作的化学生态本质。
因此,本研究聚焦于水稻品种间根系互作的化学基础,提出如下核心假设:在混种环境下,具有特殊代谢背景的水稻品种‘Kasalath’能够通过其特异的根系分泌物谱,对其他水稻品种的根系生长与代谢产生非对称调控效应,即在显著抑制竞争者根系发育的同时,维持自身相对稳定的生长与代谢状态。为验证该假设,本研究选取‘Kasalath’及另外3种水稻品种,在高度可控的水培条件下构建幼苗期配对混种体系,并整合根系表型测定与非靶向代谢组学分析,旨在明确混种条件下水稻幼苗根系形态的响应特征及其品种间互作关系、解析混种诱导的根系分泌物代谢重构特征和从代谢层面揭示‘Kasalath’与邻近品种根系生长互作的潜在机理。本研究可为理解水稻品种多样化配置的生态学基础、病虫害生态防控以及群体兼容型品种的选育提供理论依据。
-
本研究使用了4个由国际水稻研究所收集保存的水稻品种(种质),分别为‘Kasalath’、‘FL478’、‘IRGC
126961 ’和‘IRGC117406 ’ ,所有实验种子均为2024年收获于海南省东方市实验基地,为同一批次收获,并于实验前在相同条件下进行统一处理。 -
选取健康饱满的水稻种子,置于50℃恒温箱中处理48 h以打破休眠,使用5 %(w/w) NaClO对种子表面消毒20 min,用蒸馏水冲洗干净,浸种2 d,30℃催芽12 h。温室温度30℃,光周期为L光︰D暗=12 h︰12 h,相对湿度约70% ~ 80%。露白的种子手工播种于96孔去底的PCR板,每个品种播12粒,1粒1孔,将播完种子的PCR板置于配套水培盒中并在水培盒中注水至水面没过种子1 cm的位置为止,纯水培养3 d后换Yoshida营养液培养至两叶一心期。
采用完全随机区组设计,每个处理(包括单种对照及3种混种组合)设3个生物学重复,每板播种12粒种子,每个品种在单种处理中占一整板;混种处理采用1︰1的配对混种比例,即每个混种处理单元中,‘Kasalath’与伴生品种的植株数量相等。每个处理的3个重复均用于表型测量,并各取12株 幼苗的根系混合为一个样本进行后续的非靶向代谢组学分析。具体为:在每个96孔PCR板中,‘Kasalath’与相邻品种各占4排(每排2个品种交叉播种),每排12孔,共计各48株。单种处理:每个PCR板播单独品种水培,作为对照。混种处理(Co-culture):将‘Kasalath’分别与‘FL478’、‘IRGC 126961’和‘IRGC 117406’进行配对混种,每个PCR板交叉4排幼苗(4排‘Kasalath’+ 4排同伴品种)。所有处理在同一环境条件下同步培养。
-
待水稻幼苗生长至两叶一心期,用Yoshida营养液处理7天,之后将每个品种中长势均匀的幼苗依次平放在带有刻度的平板上并拍照,拍照时需注意相机位置在平板的正上方,高度以将整个平板放入取景框为准,并用三角架固定相机位置。所有品种依次拍照完成后,照片用Photoshop软件计算测量根面积,用Image J软件测量最大根长。
-
1)样本取样及前处理:在处理第10天,从 3个生物学重复 中各取 12株 幼苗的根系混合为一个样本,即每个处理共 3 个独立生物学样本(每个样本来源于12株混合组织)。拍照后把植物根系和叶片速冻后置于−80℃ 保存,后续经过真空冷冻干燥后,利用研磨仪(CT300,北京格瑞德曼)进行研磨,条件为30 Hz,1.5 min。随后称取100 mg的粉末,加入含有0.01 mg·L−1利多卡因内标的提取液(V甲醇︰V乙腈︰V纯水=1︰1︰1)。涡旋10 s后,超声10 min,重复3次。将样品在12 000 g转速下离心样品10 min,吸取上清,然后利用微孔滤膜(SCAA-104,孔径0.22 μm,上海安普)对样品进行过滤,将滤后的样品保存在进样瓶中准备用于LC-MS分析。2)非靶向代谢组检测条件及参数:非靶向代谢组检测使用Q Exactive Focus Orbitrap LC-MS/MS(Thermo Scientific, Waltham, MA, USA)。所用液相系统为UPLC系统(UltiMate
3000 ,Thermo Scientific, Waltham, MA, USA),HPLC分析条件包括:色谱柱为C18柱(Kinetex 2.6 µm Polar C18 100 Å,100*2.1 mm, Phenomenex);流动相,A相为超纯水(加入0.04% 乙酸),B相为甲醇(加入0.04% 乙酸);洗脱梯度为V纯水︰V甲醇=95︰5(0 min)→甲醇(11 min)→甲醇(15 min)→ V纯水︰V甲醇=95︰5(15.5 min)→15V纯水︰V甲醇=95︰5(15 min);流速为0.4 mL·min-1;柱温箱温度为40℃;进样量为2 μL。质谱检测采用以下设置进行数据采集:采用Full MS/dd-MS 2的扫描模式;离子扫描范围100~1 000 m/z;累积时间为0.01 s;鞘气流速40;辅助气流速12 ;喷雾电压3 000 V ;毛细管温度360℃;S-lens电压55.0 V;辅助器加热温度350℃;二级质谱扫描时,归一化碰撞能量20 V,40 V,60 V。原始检测数据采用Compound Discoverer(CD)3.3 软件进行处理,以获得所有检测化合物的质荷比(m/z)、保留时间和MS/MS2信息。 -
用MicrosoftExcel2020对原始数据进行简单处理,SPSS25.0进行方差分析和相关性分析;使用Origin2021 进行绘图,显著水平设为P<0.05或P<0.01。代谢组学数据在R环境下通过“Meta boanalyst R”包进行可视化与通路富集。差异代谢物的判定标准为 |log2 Fold Change|≥1且P<0.05(P值由t检验计算)。
-
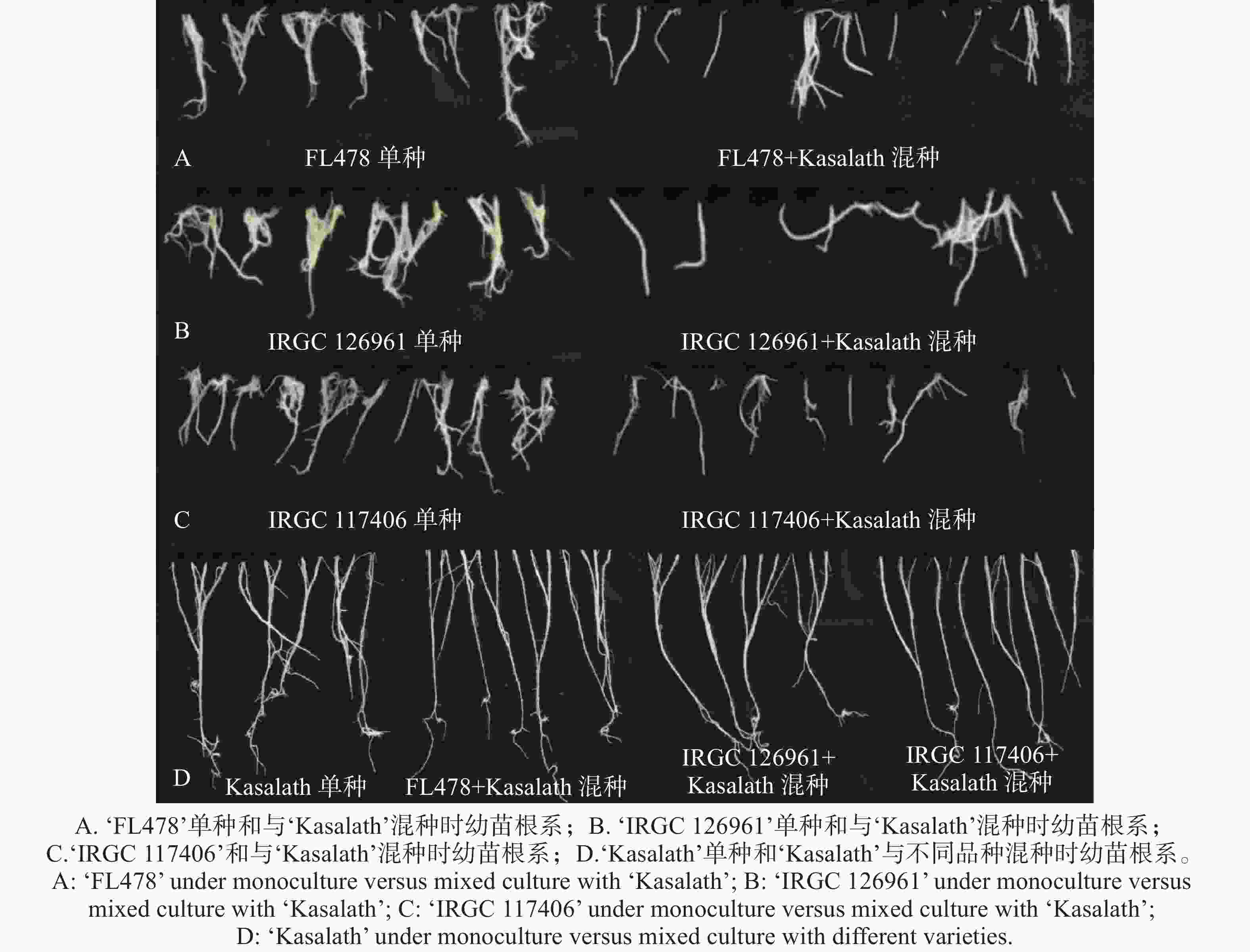
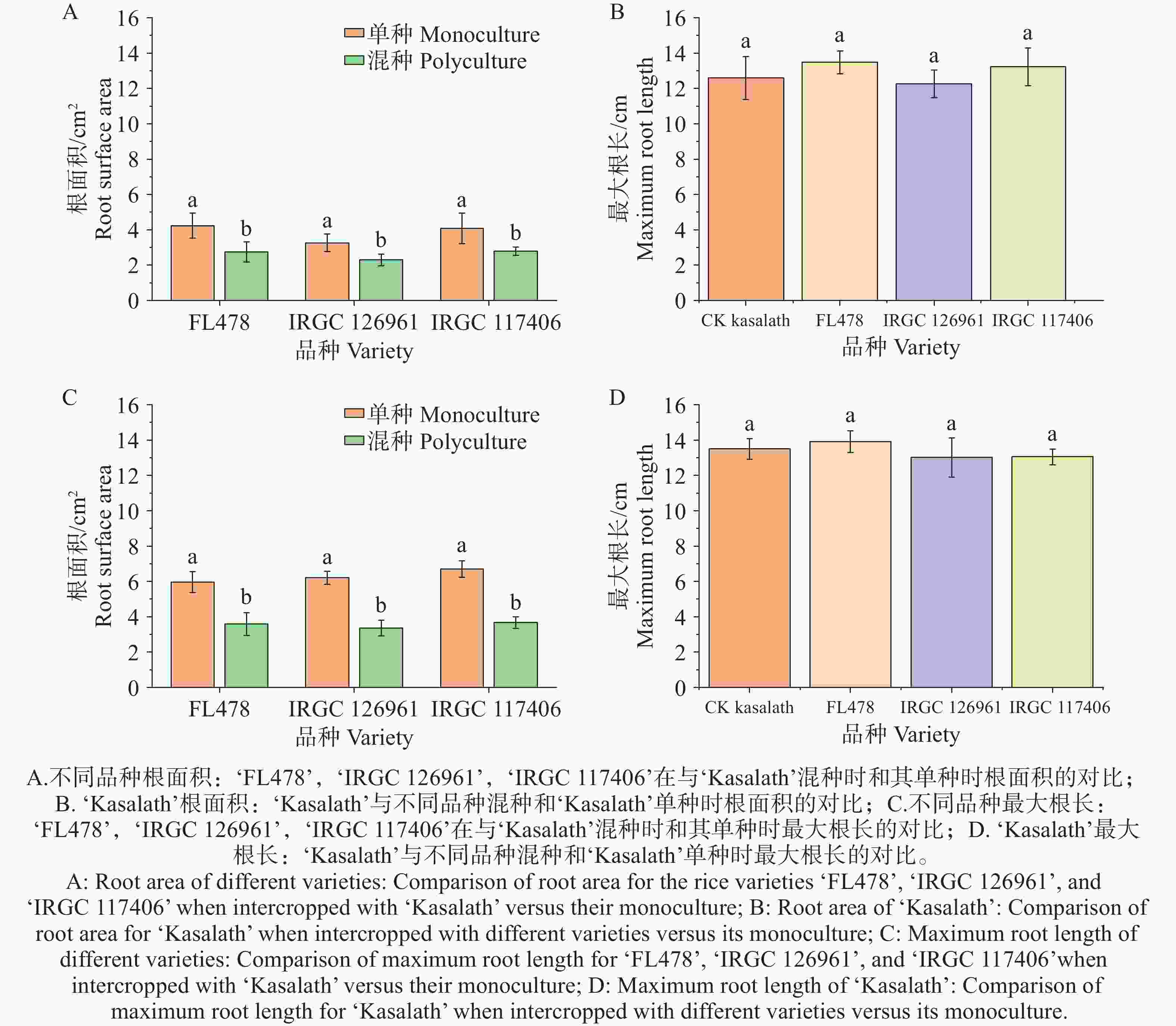
不同水稻品种单种与混种下的幼苗根系形态如图1所示。本研究比较了‘FL478’、‘IRGC
126961 ’和‘IRGC117406 ’在与‘Kasalath’混种条件下的根系生长表现。结果显示,与各自的单种处理相比,这3个品种在混种条件下的根面积和最大根长均显著下降。具体而言,‘FL478’、‘IRGC126961 ’和‘IRGC117406 ’的根面积分别下降39.8%、45.9%和45.2%;最大根长分别下降 35.24%、29.48% 和 31.54%,其中‘IRGC126961 ’的根系抑制最为显著(P < 0.05)(图2-A、C)。相比之下,‘Kasalath’的根系面积和最大根长在3种混种组合中与其自身单种对照相比均无显著变化(P > 0.05)(图2-B、D),表明其在混种条件下保持了稳定的根系生长。 -
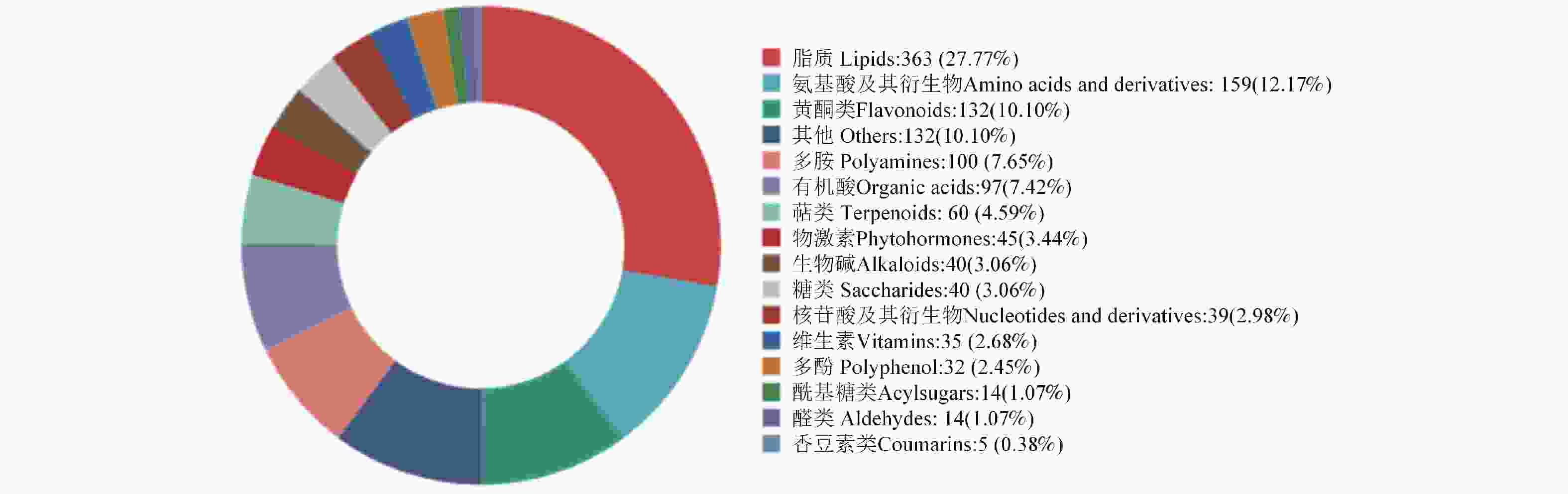
利用广泛靶向的代谢物检测技术,在水稻根系组织中共检测了1 307种代谢物的表达丰度,实验中所测定的水稻组织中的代谢物组为15个类别。代谢物分类的统计结果及各种物质的比例分布如下:脂质(Lipids)占比最大,为27.77%;氨基酸衍生物(Amino acid derivative)占12.17%;黄酮类(Flavonoid)占10.10%;有机酸(Organic acid)占7.42%;多胺(Polyamines)占7.65%;萜类化合物(Terpenoids)占4.59%;植物激素(Plant Hormones)占3.44%;生物碱(Alkaloids)占3.06%;糖类(Saccharides)占3.00%;核苷酸及其衍生物(Nucleotide derivatives)占2.98%;及维生素(Vitamins)占2.68%等。这些比例揭示了本研究样本中不同类别代谢物的组成特征(图3)。为系统解析混种互作的代谢基础,重点分析了各品种在混种处理与其自身单种对照间的差异代谢物。
-
代谢组学分析表明,3种水稻材料在与‘Kasalath’混种后,总体来看各比较组均检测到数量有限但具有方向性变化的显著差异代谢物(图4)。其中,‘FL478’在混种条件下仅呈现 7个上调和 1个下调代谢物(图4-A),其代谢响应变化幅度较小。相比之下,‘IRGC
126961 ’的代谢反应更为显著,在混种条件下共有5个上调和73个下调代谢物(图4-B),显示其代谢组受混种影响更强。对于‘IRGC117406 ’则检测到16个上调与7个下调代谢物(图4-C),表明其在混种条件下表现出相对均衡的代谢调控。三种材料均以下调代谢物为主,但下调幅度与数量在材料间存在明显差异,提示不同基因型对混种引发的代谢响应具有显著的遗传背景依赖性。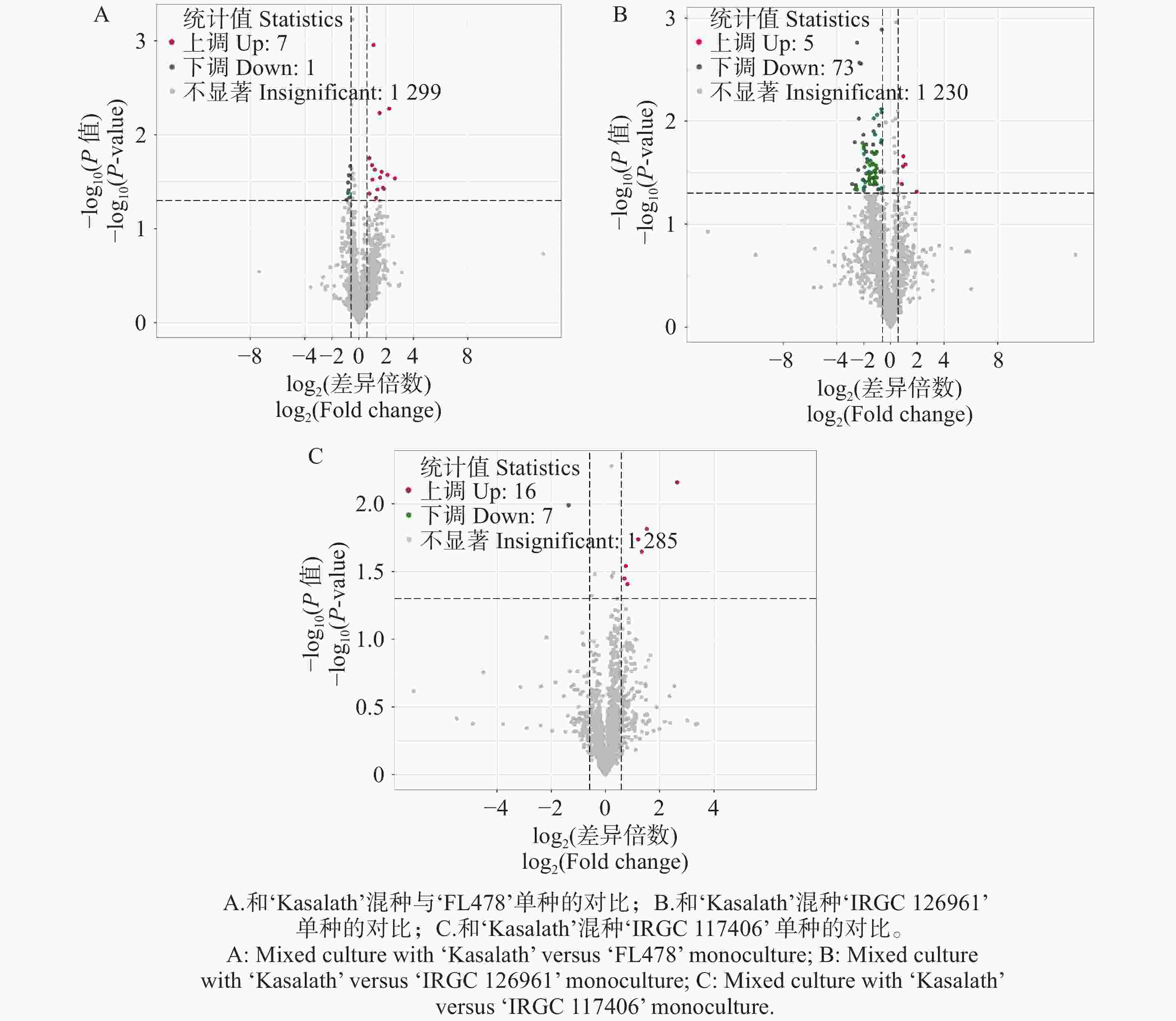
图 4 不同水稻品种混种条件下与单种时比较的差异代谢物
Figure 4. Differential metabolites in different rice varieties under mixed-culture versus monoculture conditions
代谢组学分析显示,‘FL478’在混种条件下的差异代谢物,其类别分布与单种时存在明显不同(图5-A)。本次鉴定到的差异代谢物以脂质为主(71.43%),其余则包括有机酸、多胺及其他类别代谢物(各占14.29%)。KEGG通路注释结果表明,这些差异代谢物主要富集于代谢相关通路,其中亚油酸代谢和不饱和脂肪酸生物合成通路均被显著注释(图5-B),提示‘FL478’在混种条件下与脂质代谢调控密切相关。此外,部分差异代谢物还涉及激素信号调控及离子稳态通路等多个生物过程。整体来看,‘FL478’在混种环境中的代谢响应主要表现为脂类代谢活动的改变,可能与其在混种条件下的生理适应策略密切相关。
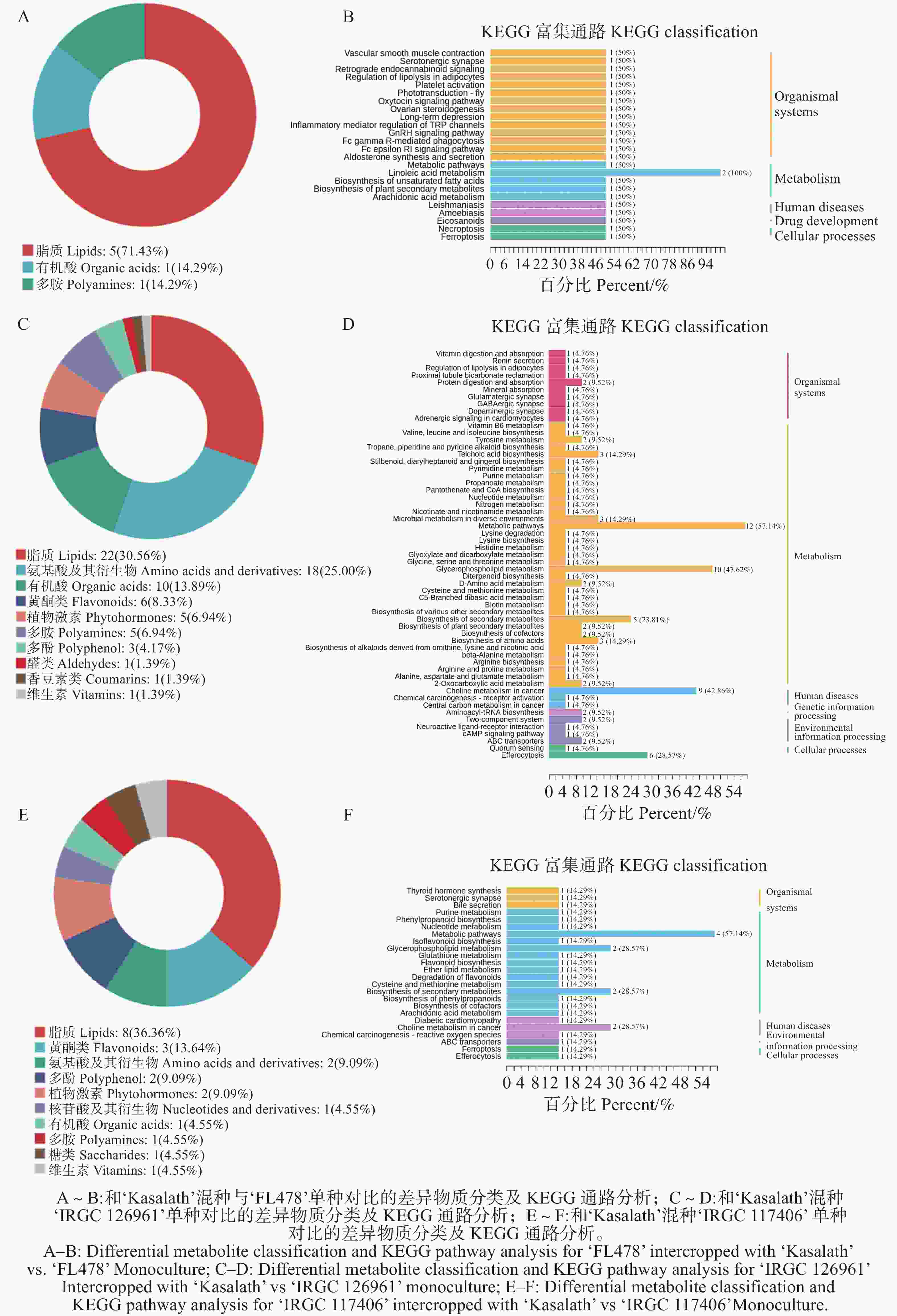
图 5 不同水稻品种混种条件下与单种时比较的差异物质分类及KEGG通路分析
Figure 5. Classification and KEGG pathway analysis of differential metabolites in different rice varieties under mixed-culture versus monoculture conditions
与单种相比,‘IRGC
126961 ’在混种条件下的差异代谢物分类存在明显不同(图5-C)。结果表明,差异代谢物的组成较为多样,主要包括脂质(30.56%)和氨基酸及其衍生物(25%),其次为有机酸(13.89%)、黄酮类(8.33%)、植物激素(6.94%)和多胺(6.94%),其余类别(如多酚类、醛类、内酯类及维生素等)所占比例较小。KEGG注释显示,这些差异代谢物广泛富集于多条代谢通路,尤其是氨基酸代谢、糖类代谢和能量相关代谢途径,如赖氨酸代谢、组氨酸代谢、胱氨酸与蛋氨酸代谢以及丙氨酸、天冬氨酸与谷氨酸代谢等(图5-D)。此外,部分代谢物还涉及生物碱生物合成、植物次生代谢物合成以及脂质代谢等过程。整体来看,‘IRGC126961 ’在混种条件下表现出更为广泛且显著的代谢重编程,特别是氨基酸及碳氮代谢途径的改变。与单种相比,‘IRGC
117406 ’在混种条件下的差异代谢物分类存在明显不同(图5-E)。代谢物分类结果显示,差异代谢物以脂质为主(36.36%),其次为黄酮类(13.64%)、氨基酸及其衍生物(9.09%)、多酚(9.09%)、植物激素(9.09%)和核苷酸及其衍生物(4.55%)。此外,有机酸、多胺、糖类和维生素等类别亦有少量分布(各占4.55%)。KEGG通路分析显示,差异代谢物主要富集于脂类和次生代谢相关通路,其中甘油磷脂代谢为占比最高的通路(图5-F),表明膜脂类组分的变化可能是‘IRGC117406 ’对混种条件的关键响应。此外,苯丙烷代谢、黄酮类生物合成、花生四烯酸代谢、植物激素生物合成以及半胱氨酸和蛋氨酸代谢等多条代谢通路亦被注释,涉及次生代谢调控、抗逆信号传导及氨基酸代谢等多个生物过程。整体而言,‘IRGC117406 ’在混种条件下的代谢变化特征以脂质和次生代谢途径的调控为主。 -
代谢组学分析表明,‘Kasalath’在与3种不同水稻材料混种时,相较于其单种条件,均能检测到一定数量的显著差异代谢物,但各组合间的代谢响应强度存在明显差异(图6)。与‘FL478’混种时,‘Kasalath’共检测到23个上调和13个下调的代谢物(图6-A),表明混种对其代谢状态产生较强影响。与‘IRGC
126961 ’混种时,差异代谢物数量略低,共鉴定19个上调和12个下调代谢物(图6-B),说明其代谢调节幅度相对中等。与‘IRGC117406 ’混种时,差异代谢物数量进一步减少,仅检测到15个上调和5个下调代谢物(图6-C),表明该混种组合对‘Kasalath’的代谢影响相对较弱。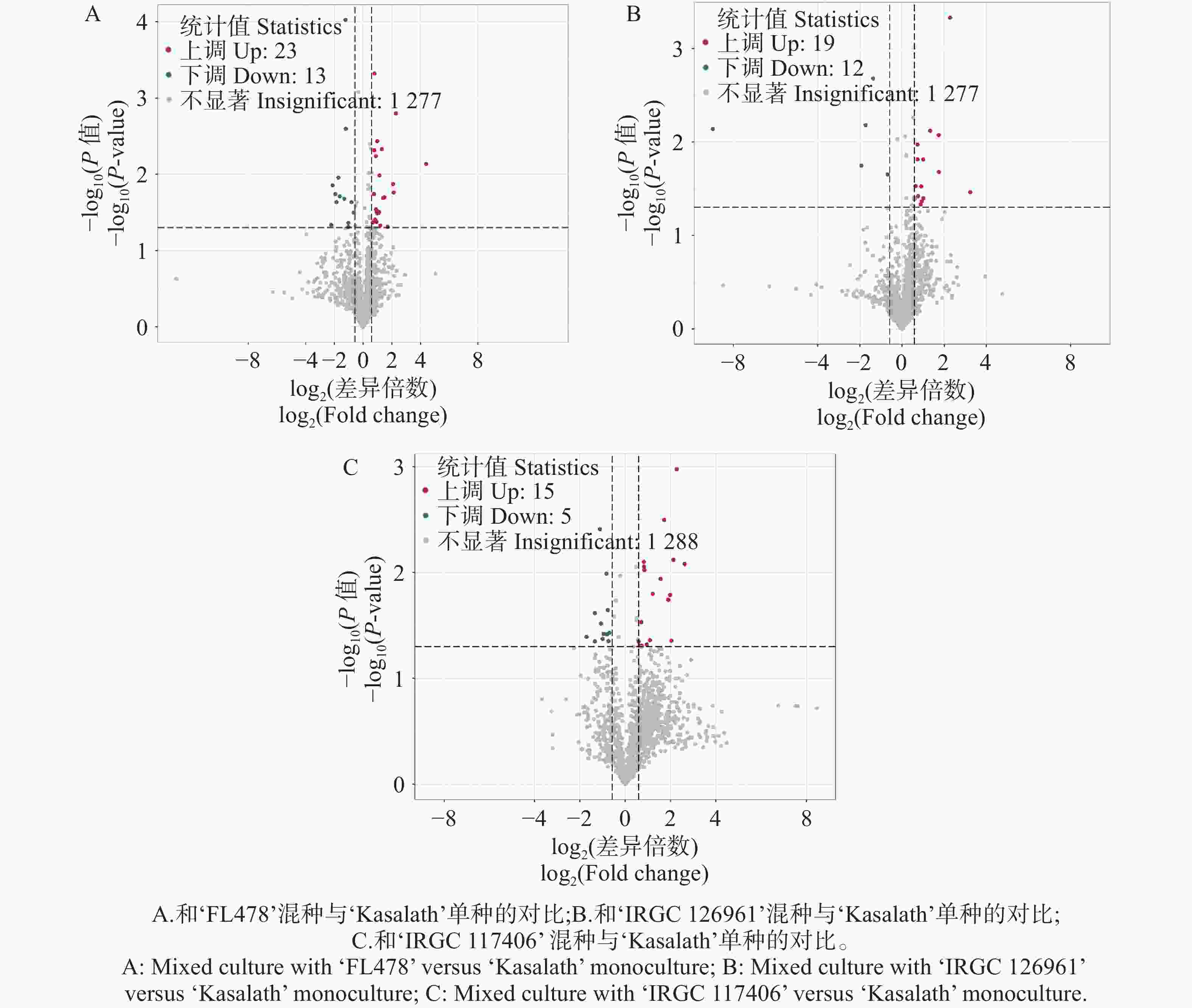
图 6 ‘Kasalath’在不同水稻品种混种与单种时的差异代谢物
Figure 6. Differential metabolites of ‘Kasalath’ under mixed-culture with different rice varieties versus monoculture
总体而言,‘Kasalath’在不同混种对象下均表现出方向性一致的代谢变化,以上调代谢物占比更高为特征,但其响应强度依赖于混种品种,其中与‘FL478’混种引发的代谢调控最为显著,而与‘IRGC
117406 ’混种的影响最小。这表明‘Kasalath’对混种环境的代谢响应具有明显的基因型差异,不同品种间的相互作用强度可能决定其代谢调控的幅度。代谢组学分析表明,‘Kasalath’在与‘FL478’混种条件下,其差异代谢物的类别分布相较于单种时存在显著不同(图7-A)。代谢物分类显示,差异代谢物来源多样,其中占比最高的类别为脂质(25%)、多胺(25%)和生物碱(10.71%),此外还包括黄酮类、核苷酸及衍生物、有机酸、植物激素、乙酰化产物、氨基酸衍生物以及糖类等。这些结果表明,‘Kasalath’在与‘FL478’混种时涉及较广泛的代谢调控网络。KEGG富集分析进一步揭示,差异代谢物主要与代谢类路径相关,包括嘌呤代谢、嘧啶代谢、核苷酸代谢、谷胱甘肽代谢及β-丙氨酸代谢等;同时还富集于苯丙烷类合成及ABC转运蛋白等路径(图7-B)。其中多条通路呈现较高占比,反映出混种显著影响‘Kasalath’在核苷酸代谢、抗氧化调控及底物运输等方面的代谢状态。
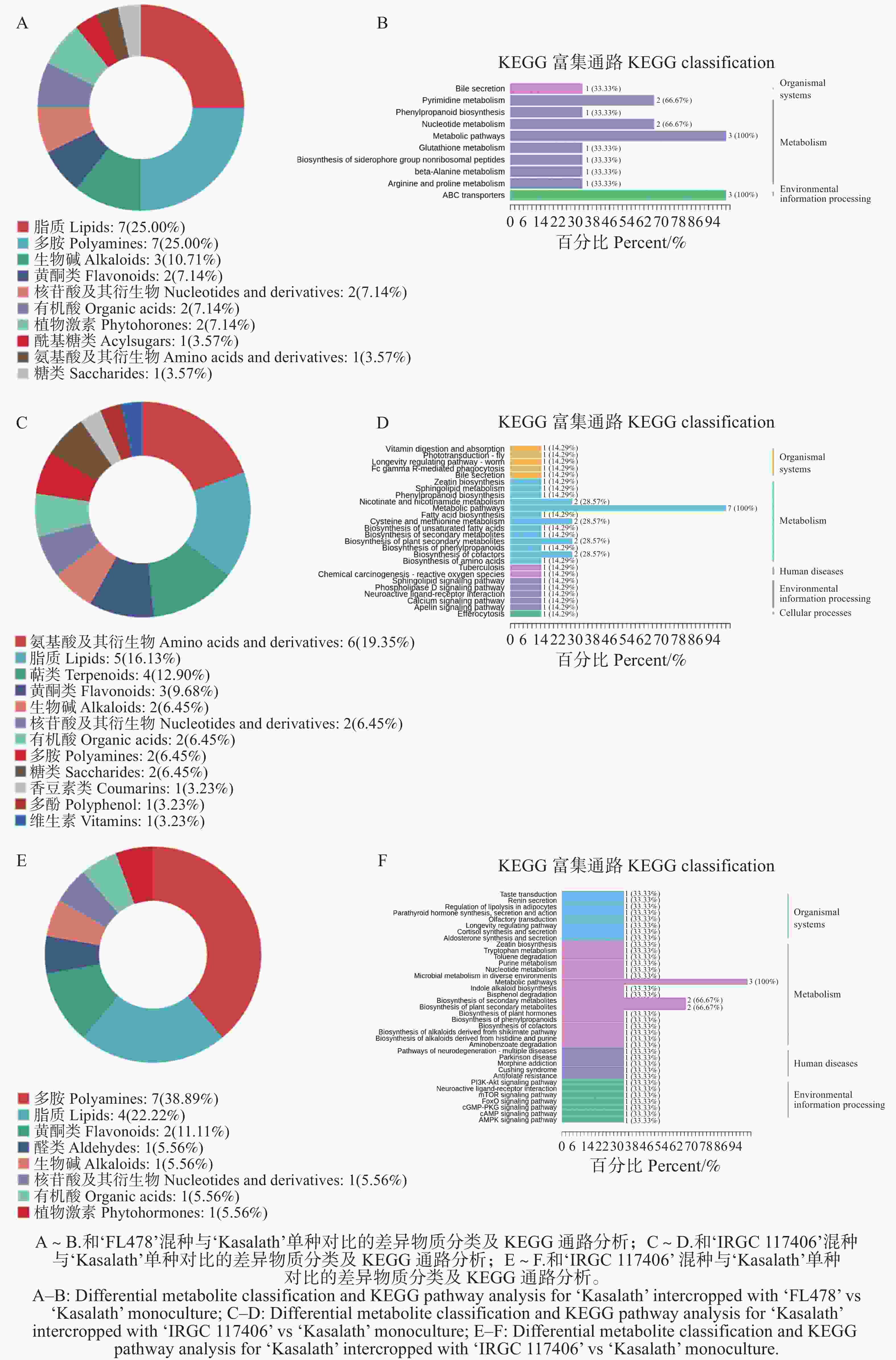
图 7 ‘Kasalath’在不同水稻品种混种与单种时的差异物质分类
Figure 7. Classification of differential metabolites in ‘Kasalath’ under mixed-culture with different rice varieties versus monoculture
整体来看,‘Kasalath’与‘FL478’混种后表现出较为复杂的代谢响应特征,其代谢变化不仅涉及基础代谢如核苷酸合成与能量平衡,还伴随抗逆性相关代谢的增强,说明‘Kasalath’在混种环境中需要通过多层次的代谢调节来适应与‘FL478’的互作过程。
‘Kasalath’在与‘IRGC
126961 ’混种后,其差异代谢物的类别分布模式较单种时呈现出明显差异(图7-C)。差异代谢物的类别分布较为多样,其中氨基酸及其衍生物占比最高(19.35%),其次为脂质(16.13%)、萜类(12.90%)、黄酮类(9.68%)以及生物碱、核苷酸、有机酸、多胺、糖类等类别(各占6.45%或以下)。该结果表明,‘Kasalath’在与‘IRGC126961 ’混种时主要涉及氨基酸代谢、脂质调控及多种次生代谢物的变化。KEGG分析显示,差异代谢物主要富集在代谢相关通路,包括半胱氨酸和蛋氨酸代谢、苯丙烷类合成、黄酮类代谢及尼古丁酸和烟酰胺代谢等多个代谢过程(图7-D)。其中,半胱氨酸和蛋氨酸代谢通路富集比例最高,提示含硫氨基酸相关的氧化还原调控可能在混种互作中发挥核心作用。此外,次生代谢相关通路(如苯丙烷代谢、黄酮类代谢)以及胁迫适应相关路径(如谷胱甘肽代谢、ABC转运蛋白)均有出现,说明混种条件显著影响‘Kasalath’的抗氧化能力、细胞保护机制以及代谢物跨膜运输活动。总体而言,‘Kasalath’在与‘IRGC126961 ’混种后呈现以氨基酸代谢和次生代谢调控为核心的代谢重构特征,伴随明显的氧化还原平衡调节与防御性代谢增强,反映其在混种互作中需要构建更强的代谢适应能力。对比分析显示,‘Kasalath’在与‘IRGC
117406 ’混种条件下,其差异代谢物的类别分布与单种条件存在明显差异(图7-E)。差异代谢物分类结果显示,多胺类占比最高(38.39%),其次为脂类脂质(22.22%)和黄酮类(11.11%),其余类别包括其他类代谢物、醛类、生物碱、核苷酸衍生物、有机酸和植物激素等(均占5.56%)。这些结果表明该混种组合中,‘Kasalath’的代谢调控主要集中在多胺代谢、脂类调控及少量次生代谢路径上。KEGG 分析进一步揭示,差异代谢物主要富集于代谢类路径,包括核苷酸代谢、核糖核苷酸代谢、色氨酸代谢以及生物碱生物合成等(图7-F)。其中核苷酸和核糖核苷酸代谢富集比例最高,说明混种条件显著影响与细胞能量代谢、遗传物质合成和信号分子的生物合成相关的代谢过程。此外,色氨酸代谢、苯丙烷代谢及多种防御性代谢路径亦有富集,提示混种可能诱导‘Kasalath’的胁迫响应增强。值得注意的是,与‘IRGC117406 ’混种的情况下,‘Kasalath’的代谢变化整体幅度较小,但呈现更集中于少数关键代谢节点的特点。总体来看,‘Kasalath’在与‘IRGC117406 ’混种时表现出以多胺代谢和核苷酸代谢为核心的代谢重塑特征,并涉及一定程度的次生代谢调控与防御反应。这表明该混种组合对‘Kasalath’的刺激较温和,但仍触发了调控生长、能量代谢与细胞稳态的关键代谢过程。 -
本研究通过根系形态测定与代谢组分析揭示了‘Kasalath’与3种水稻品种(‘FL478’、‘IRGC
126961 ’、‘IRGC117406 ’)在混种条件下的根际互作机制。结果显示,‘Kasalath’对其他品种根系生长具有显著的抑制效应,而其自身根生长在混种条件下保持稳定。同时,代谢组分析显示混种诱导的代谢变化具有双向性,‘Kasalath’与伴生品种之间存在明显的代谢互作。这种“表型单向、代谢双向”的互作模式,反映了根际竞争、化感信号与代谢稳态调节之间的复杂关系。首先,根系形态结果表明,‘FL478’、‘IRGC
126961 ’和‘IRGC117406 ’的根面积与最大根长在与‘Kasalath’混种时显著下降,提示‘Kasalath’在根际互作中占据明显优势。根际混种常导致植物间在养分吸收、水分利用及空间占据等方面产生竞争,还可能表现为化学生态干扰。例如,水稻可通过根系分泌特定的酚酸类物质,显著抑制邻近杂草慈姑的根系生长[17]。植物在竞争过程中既可通过根系性状提高资源获取效率,也可能通过化学抑制影响邻体生长,这些机制被认为共同塑造植物间的竞争关系[18]。在同种作物不同品种共存的情境下,由于根系形态与资源需求高度相似,单纯的空间或养分竞争往往不足以解释显著的生长抑制效应,此时根际信号调控与化学干扰可能发挥更加关键的作用。已有研究表明,植物根系可释放酚酸类、脂类信号分子及其他次生代谢物,这些化学因子能够直接影响邻体根系细胞伸长区的活性、分枝形成及激素平衡[10]。因此,在本研究中观察到的根系抑制现象,很可能是资源竞争与化学信号干扰协同作用的结果,其中,‘Kasalath’可能通过更早或更强的根际信号释放,在竞争初期即建立优势地位。
-
与伴生品种形成鲜明对比的是,‘Kasalath’在混种环境中根系表型保持稳定,未表现出显著的生长抑制。这一现象表明,‘Kasalath’不仅在竞争中占据优势,还能够在复杂根际环境下维持自身生长稳态。已有研究表明,不同水稻基因型在根系结构与生理调控方面存在显著差异,部分基因型能够在胁迫或竞争环境中通过维持根系发育与功能稳定性而表现出更强的适应能力[19]。此外,耐竞争型基因型往往表现出灵敏的根际信号感知与响应机制,从而在邻体存在的条件下快速调整代谢活动以维持生长[20]。
从生态策略角度看,这类基因型并非对邻体“无反应”,而是通过代谢层面的精细调控避免生长代价的累积。已有研究表明,植物在感知到邻体存在后,往往会提前调整碳氮分配、抗氧化系统以及膜脂组成,以缓冲潜在的资源竞争和化学干扰[21]。本研究中,‘Kasalath’虽未表现出明显的根系形态变化,但其代谢组显示出针对不同混种对象的定向调节,提示其具备较强的代谢可塑性。这种“感知-响应-稳态维持”的能力,可能是‘Kasalath’在混种体系中保持生长优势的重要生理基础。
-
代谢组分析进一步揭示了水稻品种间根际互作的代谢基础。3个伴生品种在混种条件下均发生了显著的代谢重构,主要涉及脂类、氨基酸、核苷酸以及多条次生代谢途径。从代谢调控角度看,脂类代谢变化通常与细胞膜结构调控、信号脂质合成及氧化胁迫响应密切相关[21-22];氨基酸代谢的显著改变通常反映植物在资源竞争压力下进行氮素再分配和蛋白质周转调节[23-24];而多胺、黄酮类等次生代谢物被认为参与化感反应、抗氧化保护以及根际信号传递等过程[25-26]。
这些代谢变化表明,伴生品种在混种条件下面临较强的生理压力,其代谢系统被迫向防御与补偿方向重编程,但这种代谢重构并未有效转化为根系生长维持能力,最终表现为明显的根系抑制表型。
值得注意的是,尽管‘Kasalath’的根系表型在混种条件下保持稳定,其代谢组同样表现出明确的响应特征。不同混种组合中,‘Kasalath’分别涉及脂类调控、多胺代谢或核苷酸代谢等通路的变化,表明其并非被动维持稳态,而是主动感知并响应邻体存在。这种“代谢响应但生长不受影响”的现象符合植物在竞争环境中维持生长稳健性的理论框架,即通过提前调节代谢网络来避免资源短缺或信号干扰对形态建成造成实质性影响。已有研究指出,植物即使在形态上未表现出竞争反应,其根系仍可通过改变代谢物分泌和信号通路调控根际微环境,从而影响邻体生长与微生物群落结构[11]。因此,‘Kasalath’的代谢变化不仅反映了其对邻体存在的感知能力,也揭示了其通过代谢稳态调控规避生长代价、并可能对邻体施加间接抑制的潜在机制。
总体来看,在幼苗期配对混种条件下,‘Kasalath’表现出较强的根系竞争优势,其根系生长在混种环境中保持相对稳定,而伴生品种的根系生长则受到不同程度的抑制。代谢组结果表明,‘Kasalath’在混种条件下能够感知邻体存在并对自身代谢进行定向调节,从而维持生长稳态;相比之下,伴生品种在混种处理中表现出更大幅度的代谢重构,其代谢扰动未能有效转化为根系生长的补偿,最终呈现出明显的根系抑制表型。这一结果提示,不同水稻品种在混种环境下对根际互作信号的响应方式和代谢调节能力存在显著差异。
结合根系形态变化与代谢组特征可以看出,混种条件下的根际互作并非单一过程所致,而可能是资源竞争与根际化学信号共同作用的结果。代谢组分析为识别这种互作提供了比单一表型指标更为敏感的信息层级,有助于揭示植株在竞争环境中发生的早期生理与代谢响应。本研究的结果证明水稻品种间的根际互作受基因型背景影响,不同品种在混种环境下表现出差异化的代谢响应和生长结果,这种差异可能与其代谢稳态维持能力和根际适应策略密切相关。
本研究从系统代谢层面揭示了水稻品种间根际互作及其非对称响应特征,旨在解析互作发生的代谢框架与生理背景,而并非直接鉴定具体的致效代谢物。根际互作,尤其是同种内互作,往往由多个代谢途径和信号网络协同调控,其效应具有明显的情境依赖性。未来研究有必要在此基础上,结合靶向代谢组学、生物测定以及外源添加实验,对潜在关键代谢物及其因果作用进行进一步验证。
总体而言,本研究通过整合根系表型与非靶向代谢组数据,为理解水稻品种在混种条件下的根际互作方式提供了新的代谢学证据,也为后续从根际互作角度探索品种配置策略、群体兼容性评价以及生态栽培模式的优化奠定了理论基础。
Asymmetric regulation of root growth and metabolism in different rice varieties by rice variety ‘Kasalath’ under mixed-cropping conditions
-
摘要: 水稻(Oryza sativa)品种在幼苗期的根际互作对群体结构建成与资源获取具有重要影响,为了探究其化学生态机制,在可控条件下,通过配对混种试验,比较了‘Kasalath’与‘FL478’、‘IRGC
126961 ’、‘IRGC117406 ’的根系互作效应,系统分析了根系形态差异及根系分泌物代谢特征。结果表明,混种条件下‘Kasalath’根系生长保持稳定,而3个伴生品种的根面积与最大根长均受到显著抑制,降幅达 30%~46%,呈现出明显的非对称抑制效应。代谢组学共鉴定到 1 307 种代谢物,受抑制品种在混种后均发生显著代谢重构,主要涉及脂类、氨基酸衍生物、黄酮类及多胺类等代谢途径。尽管‘Kasalath’表型稳定,但其代谢谱在不同混种组合中仍发生特异性变化,涉及核苷酸代谢、苯丙烷代谢和谷胱甘肽代谢等通路。综合表型与代谢组数据可知,‘Kasalath’通过较强的根际竞争能力与代谢稳态维持机制抑制伴生品种根系生长,而伴生品种代谢补偿能力不足,导致生长受抑。Abstract: The belowground interactions of rice varieties during the seedling stage play a critical role in population structure establishment and resource acquisition, whereas their underlying chemical ecological mechanisms remain poorly understood. Under controlled conditions, paired mixed-cultivation experiments were conducted to compare the root interaction effects between ‘Kasalath’ and three companion rice varieties (‘FL47’, ‘IRGC126961 ’, ‘IRGC117406 ’). Root morphological traits and root exudate metabolic profiles were systematically analyzed. The results showed that under mixed cultivation, root growth of ‘Kasalath’ remained stable, whereas all the three companion varieties exhibited significant inhibition in root surface area and maximum root length, the decrease of which ranged from 30% to 46%, indicating a clear asymmetric inhibitory effect. Metabolomics analysis identified a total of 1,307 metabolites. The inhibited varieties underwent significant metabolic reprogramming after mixed cultivation, primarily involving lipid, amino acid derivative, flavonoid, and polyamine metabolism pathways. Despite its stable phenotype, ‘Kasalath’ also displayed specific metabolic profile changes across different mixed combinations, including pathways such as nucleotide metabolism, phenylpropanoid metabolism, and glutathione metabolism. Integrating phenotypic and metabolomics data, it is demonstrated that ‘Kasalath’ suppresses root growth of companion varieties through stronger rhizosphere competitive ability and metabolic homeostasis maintenance mechanisms, while the limited metabolic compensation capacity of the companion varieties leads to growth inhibition. These findings reveal the chemical basis of root interactions between rice varieties at the metabolic level, providing a theoretical foundation for mixed-cultivation design and the development of efficient plant communities.-
Key words:
- rice variety interaction /
- root morphology /
- metabolomics /
- allelopathy
-
图 2 不同水稻品种单种与混种条件下根系形态特征比较
注:不同小写字母代表同一品种中处理间在α=0.05水平上存在显著差异。
Fig. 2 Comparison of root morphological traits of different rice varieties under monoculture and mixed-culture conditions
Note: Different lowercase letters indicate significant differences (P < 0.05)between treatments for a given variety.
-
[1] Cahill J F, Jr, Mcnickle G G. The behavioral ecology of nutrient foraging by plants [J]. Annual Review of Ecology, Evolution, and Systematics, 2011, 42: 289−311. https://doi.org/10.1146/annurev-ecolsys-102710-145006 doi: 10.1146/annurev-ecolsys-102710-145006 [2] 凌启鸿. 作物群体质量[D]. 扬州: 扬州大学, 2003. (查阅网上资料, 未找到本条文献信息, 请确认) [3] Zhu Y Y, Chen H R, Fan J H, et al. Genetic diversity and disease control in rice [J]. Nature, 2000, 406(6797): 718−722. https://doi.org/10.1038/35021046 doi: 10.1038/35021046 [4] Altieri M A. The ecological role of biodiversity in agroecosystems [J]. Agriculture, Ecosystems & Environment, 1999, 74(1/3): 19−31. https://doi.org/10.1016/S0167-8809(99)00028-6 doi: 10.1016/S0167-8809(99)00028-6 [5] Pretty J, Bharucha Z P. Sustainable intensification in agricultural systems [J]. Annals of Botany, 2014, 114(8): 1571−1596. https://doi.org/10.1093/aob/mcu205 doi: 10.1093/aob/mcu205 [6] Tilman D, Downing J A. Biodiversity and stability in grasslands [J]. Nature, 1994, 367(6461): 363−365. https://doi.org/10.1038/367363a0 doi: 10.1038/367363a0 [7] 陈天祥, 杨顺瑛, 王书伟, 等. 水稻氮素利用效率的基因型差异与调控途径[J]. 土壤, 2022, 54(5): 873−881. https://doi.org/10.13758/j.cnki.tr.2022.05.001 doi: 10.13758/j.cnki.tr.2022.05.001 [8] Li L, Sun J H, Zhang F S, et al. Wheat/maize or wheat/soybean strip intercropping: I. Yield advantage and interspecific interactions on nutrients [J]. Field Crops Research, 2001, 71(2): 123−137. https://doi.org/10.1016/S0378-4290(01)00156-3 doi: 10.1016/S0378-4290(01)00156-3 [9] Kong C H, Xuan T D, Khanh T D, et al. Allelochemicals and signaling chemicals in plants [J]. Molecules, 2019, 24(15): 2737. https://doi.org/10.3390/molecules24152737 doi: 10.3390/molecules24152737 [10] Bais H P, Weir T L, Perry L G, et al. The role of root exudates in rhizosphere interactions with plants and other organisms [J]. Annual Review of Plant Biology, 2006, 57: 233−266. https://doi.org/10.1146/annurev.arplant.57.032905.105159 doi: 10.1146/annurev.arplant.57.032905.105159 [11] Semchenko M, Saar S, Lepik A. Plant root exudates mediate neighbour recognition and trigger complex behavioural changes [J]. New Phytologist, 2014, 204(3): 631−637. https://doi.org/10.1111/nph.12930 doi: 10.1111/nph.12930 [12] Kato-Noguchi H, Peters R J. The role of momilactones in rice allelopathy [J]. Journal of Chemical Ecology, 2013, 39(2): 175−185. https://doi.org/10.1007/s10886-013-0236-9 doi: 10.1007/s10886-013-0236-9 [13] Macías F A, Molinillo J M G, Varela R M, et al. Allelopathy—a natural alternative for weed control [J]. Pest Management Science, 2007, 63(4): 327−348. https://doi.org/10.1002/ps.1342 doi: 10.1002/ps.1342 [14] Willis R J. Juglans spp. , juglone and allelopathy [J]. Allelopathy Journal, 2000, 7(1): 1−55. https://doi.org/10.1111/j.1365-2745.2006.01124.x doi: 10.1111/j.1365-2745.2006.01124.x [15] Schenk H J. Root competition: beyond resource depletion [J]. Journal of Ecology, 2006, 94(4): 725−739. https://doi.org/10.1111/j.1365-2745.2006.01124.x doi: 10.1111/j.1365-2745.2006.01124.x [16] 储薇, 郭信来, 张晨, 等. 丛枝菌根真菌-植物-根际微生物互作研究进展与展望[J]. 中国生态农业学报(中英文), 2022, 30(11): 1709−1721. https://doi.org/10.12357/cjea.20220093 doi: 10.12357/cjea.20220093 [17] Seal A N, Haig T, Pratley J E. Evaluation of putative allelochemicals in rice root exudates for their role in the suppression of arrowhead root growth [J]. Journal of Chemical Ecology, 2004, 30(8): 1663−1678. https://doi.org/10.1023/B:JOEC.0000042075.96379.71 doi: 10.1023/B:JOEC.0000042075.96379.71 [18] Lynch J P. Roots of the second green revolution [J]. Australian Journal of Botany, 2007, 55(5): 493−512. https://doi.org/10.1071/BT06118 doi: 10.1071/BT06118 [19] Henry A, Gowda V R P, Torres R O, et al. Variation in root system architecture and drought response in rice (Oryza sativa): phenotyping of the OryzaSNP panel in rainfed lowland fields [J]. Field Crops Research, 2011, 120(2): 205−214. https://doi.org/10.1016/j.fcr.2010.10.003 doi: 10.1016/j.fcr.2010.10.003 [20] Fang S Q, Clark R T, Zheng Y, et al. Genotypic recognition and spatial responses by rice roots [J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(7): 2670−2675. https://doi.org/10.1073/pnas.1222821110 doi: 10.1073/pnas.1222821110 [21] Wasternack C, Hause B. Jasmonates: biosynthesis, perception, signal transduction and action in plant stress response, growth and development. An update to the 2007 review in Annals of Botany [J]. Annals of Botany, 2013, 111(6): 1021−1058. https://doi.org/10.1093/aob/mct067 doi: 10.1093/aob/mct067 [22] Nakamura Y. Phosphate starvation and membrane lipid remodeling in seed plants [J]. Progress in Lipid Research, 2013, 52(1): 43−50. https://doi.org/10.1016/j.plipres.2012.07.002 doi: 10.1016/j.plipres.2012.07.002 [23] Masclaux-Daubresse C, Daniel-Vedele F, Dechorgnat J, et al. Nitrogen uptake, assimilation and remobilization in plants: challenges for sustainable and productive agriculture [J]. Annals of Botany, 2010, 105(7): 1141−1157. https://doi.org/10.1093/aob/mcq028 doi: 10.1093/aob/mcq028 [24] Hildebrandt T M, Nesi A N, Araújo W L, et al. Amino acid catabolism in plants [J]. Molecular Plant, 2015, 8(11): 1563−1579. https://doi.org/10.1016/j.molp.2015.09.005 doi: 10.1016/j.molp.2015.09.005 [25] Gill S S, Tuteja N. Polyamines and abiotic stress tolerance in plants [J]. Plant Signaling & Behavior, 2010, 5(1): 26−33. https://doi.org/10.4161/psb.5.1.10291 doi: 10.4161/psb.5.1.10291 [26] Hassan S, Mathesius U. The role of flavonoids in root–rhizosphere signalling: opportunities and challenges for improving plant–microbe interactions [J]. Journal of Experimental Botany, 2012, 63(9): 3429−3444. https://doi.org/10.1093/jxb/err430 doi: 10.1093/jxb/err430 -

 点击查看大图
点击查看大图
计量
- 文章访问数: 62
- HTML全文浏览量: 36
- 被引次数: 0



 下载:
下载: