-
在海洋中,虫黄藻与珊瑚可以形成共生关系,依靠虫黄藻、适盐生物与珊瑚宿主的相互结合来生活和适应不同的环境与压力条件[1-3]。由于人类活动和气候变化,在全球范围频繁发生大规模的珊瑚礁白化事件。虫黄藻密度的变化反映虫黄藻的光化学能力,测定虫黄藻的种类与密度对珊瑚白化具有重要的预测意义[4]。目前,快速、准确地检测海洋微藻细胞存在较大的局限[5],特别是在虫黄藻种群和计算密度的方法上。研究人员采样后需要将样品固定后带回实验室再进行检测鉴定,固定剂加上长时间的运输导致样品中绝大部分藻类死亡,而无法区分正常的虫黄藻细胞和白化珊瑚体内或游离的失去色素的虫黄藻细胞。此外,由于藻体离开了原本的生存环境,在运输途中易受到温度、光照和压强等其它因素的影响,使细胞的一些特征发生改变。被称为“芯片上的实验室”的微流控技术,具有微型化、集成化、高灵敏度和低成本的特点,已在单细胞研究领域引起了越来越多的关注。微流控芯片可用于微藻细胞实时检测和环境微生物监测[6-7],目前主要的检测方法是在芯片上采集微藻细胞的叶绿素荧光信号[8]、电化学信号[9-10]与施加外部物理场[11],这些方法很难对未知的和混合的微藻细胞进行分类和鉴定,因为许多亚型的甲藻细胞在外观上只表现出微小的差异,其物理和化学性质没有变化,而且建立电场和其他物理场对活体细胞有侵入性,不利于下游细胞分析。
深度学习神经网络(Deep Learning Neural Network,DLNN)是人工智能领域中的一种技术架构,其已被广泛应用于微流控芯片设计[12-13]、细胞检测[14-16]、细胞生长分析[17-18]、药物筛选[19]等;卷积神经网络(Convolutional Neural Network,CNN)是DLNN的一种类型,更适合于图像分析[20-21]。细胞图像包含表型变量、细胞内部结构及细胞的分子信息,如荧光标记的基因或蛋白质[22-23];通过神经网络能够对这些人眼难以识别的微妙特征,如在不同Cu2+溶液处理下微藻细胞的形态变化[24],不同培养条件下产脂微藻细胞中的脂滴大小[25-26]等。此外,神经网络还能将微藻细胞的光衍射信号重建为图像以检测细胞活力与类别[27-29]。微流控芯片技术避免了前期繁琐的样本处理,可以以动态的形式直接检测水样中的活细胞,检测后的样本还可以保存和进一步分析。显微成像技术可以实时采集藻类细胞图像数据,并利用深度学习神经网络自动化检测跟踪并识别在微流控芯片中动态流动的细胞。微流控芯片与神经网络能够根据不同的细胞类型灵活设计,为实时检测多种海洋微藻细胞提供了可能性。本研究结合微流控芯片技术、显微成像技术与深度神经网络,提出了一种基于深度神经网络的活体虫黄藻检测方法,用于快速、无标签、低成本地检测活体虫黄藻。
-
在微流控芯片上设计了多个不同宽度的通道,使该芯片能够承载不同大小与形状的细胞的样本溶液。每个通道具有相同的结构,由1个样品入口、1个出口、1个聚焦微通道和1个成像区域组成。样品入口和出口的直径和深度分别为0.7 mm和3 mm。通过1个截面逐渐缩小的聚焦微通道,虫黄藻细胞能够有序地流动至成像区,避免了细胞因通道界面突然变小而导致的细胞聚集和堵塞。4个聚焦微通道的宽度分别为50 、75 、100 、125 μm,长度均为5 mm。成像区在聚焦微通道的中间,宽300 μm,长500 μm。微流控芯片是由标准软光刻技术将单层聚二甲基硅氧烷(Polydimethylsiloxane,PDMS)(Slygard184,道康宁,美国)与玻璃键合而成。
-
选用虫黄藻作为检测对象,从西岛(海南三亚,北纬18°14′16″,东经109°21′54″)的硬骨珊瑚Galaxea fascicularis中分离出来,通过测序鉴定,该虫黄藻的种株为Cladocopium sp. C1 (Symbiodiniaceae)。培养虫黄藻所用的培养基为标准的f/2人工海水培养基。将虫黄藻菌株的种子接种至培养基后放入恒温光照培养箱(PGX-450D, 宁波赛福实验仪器有限公司,中国)中培养,培养箱温度设置为25 ℃,光照条件为200 μmol·m−2·s−1,光暗周期为12 h∶12 h。每隔2 d取少许藻液,用分光光度计(ThermoSpectronic Genesys® 10-S, 贝克曼库尔特有限公司,美国)测定3次样品在730 nm处的光密度(OD730)并取平均值,监测液体培养中虫黄藻细胞的生长。用血细胞计数法测量藻液密度。
采用热胁迫的方式使虫黄藻失活褪色。在超净工作台下取部分健康的虫黄藻溶液于锥形瓶中,将锥形瓶放入恒温光照培养箱中,培养箱温度为40 ℃,光照条件为200 μmol·m−2·s−1,在该条件下对虫黄藻进行2 h的热胁迫。
-
采用直径为20 μm的PMMA(polymethyl methacrylate)微球(东莞科迈新材料有限公司,中国)对神经网络模型进行性能测试。微球密度为1.19 g·cm−3,粒径偏差小于10%。称取6.0 g的微球,将其溶解于10 mL磷酸盐缓冲盐水中(PBS×1,武汉赛维尔生物科技有限公司 ,中国)。取5 mL健康的虫黄藻细胞溶液与5 mL漂白的虫黄藻细胞溶液,用PBS缓冲液分别将两者的细胞密度稀释至6×105 个·mL−1。本试验不对藻细胞溶液进行离心过滤,以保留溶液中死亡的细胞、细胞破裂碎片等杂质,模拟在野外条件采集的水体样本,同时,这些溶液中的杂质也作为图像中的噪声存在,在进行神经网络模型训练过程中可以增加模型的鲁棒性,在结果测试中杂质的存在也能更加全面地反映细胞检测的精确度。
-
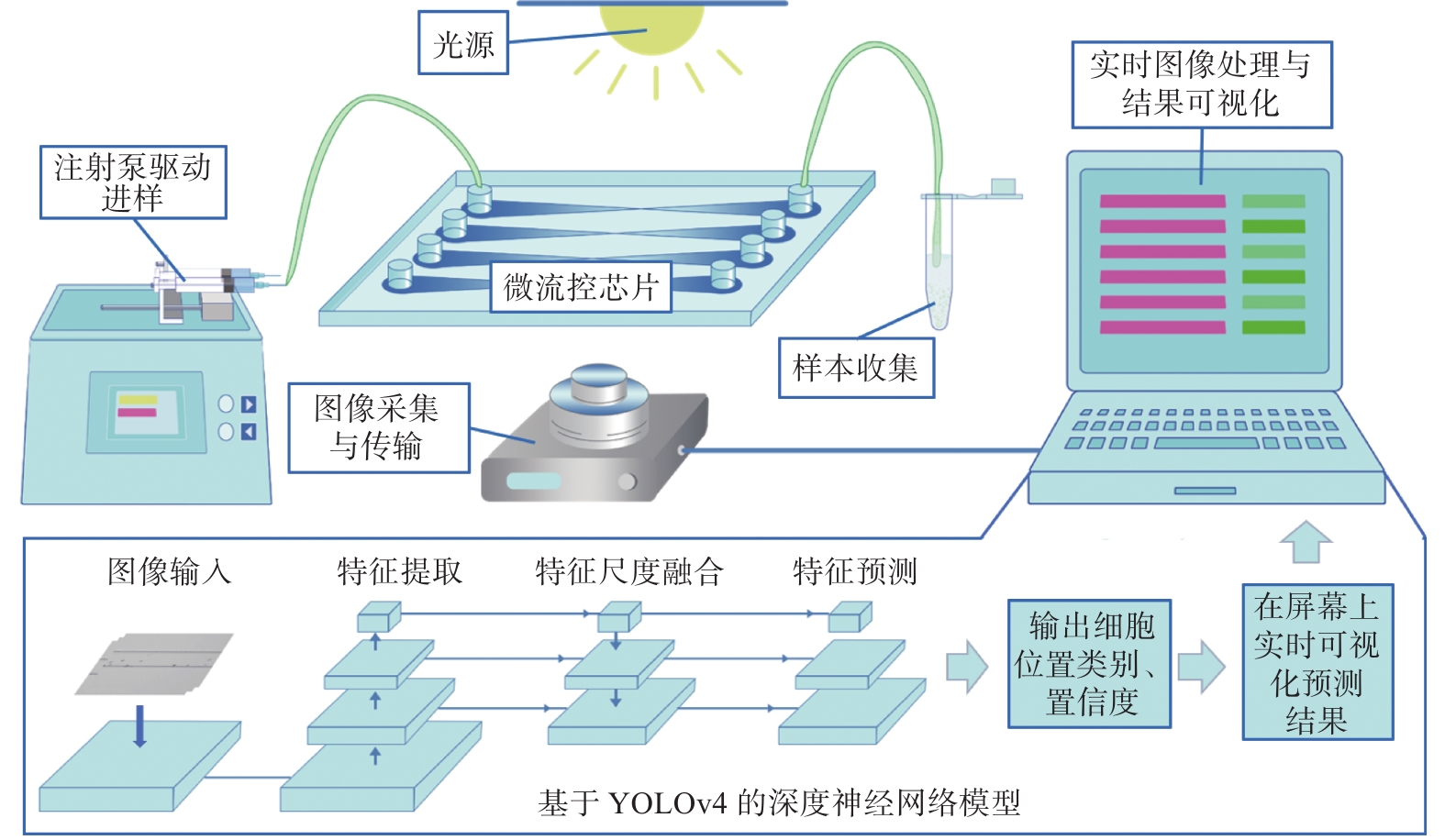
基于深度神经网络的活体虫黄藻检测方法主要包括3个模块,分别为进样模块、微流控模块、数据采集与处理模块(图1)。进样模块负责控制和驱动甲藻细胞通过注射器泵进入微通道;微流控模块承载细胞流动以进行实时的细胞图像捕获;数据采集与处理模块包括1个CMOS相机与1台微型计算机 (同方“机械革命Z2-Air”,中国),CMOS相机收集、传输藻细胞在微通道中的图像至计算机中,通过计算机中的深度神经网络模型对图像中的所有目标进行定位与分类,并将预测的细胞坐标、类别与置信度实时可视化与屏幕上。检测完成后可以通过评价指标的计算算法评价神经网络模型的准确性。
由于采集的芯片上图像属于大背景小目标图像,因此,本研究所用的深度神经网络在原有YOLOv4神经网络[30]的基础上进行了优化。优化方法包括增大图像输入分辨率、数据归一化、缩放检测框,使神经网路模型更加适用于芯片上细胞检测。活体虫黄藻图像的详细检测流程如下:微通道中的虫黄藻图像被采集后重设其分辨率,输入图像至深度神经网络模型,神经网络经过逐层的上采样提取图像特征,由大到小生成3层特征图;随后进行特征尺度融合,将上层的语义信息表征能力强但分辨率低的特征图传递到分辨率高但语义信息表征能力弱的下层特征图上,使神经网络更为准确地检测与分割目标。模型将预测融合后的特征图并输出细胞的位置、类别与置信度,最后选择置信度最高的检测框可视化于屏幕上,做到实时的活体虫黄藻检测。
-
图像采集和处理是基于1个倒置的显微镜成像平台(DS-FI3,尼康,日本)。在倒置显微镜上安装1个具有7.18 mm×5.32 mm视场和590万像素的CMOS(Complementary Metal-Oxide-Semiconductor Transistor)图像传感器的高清相机。相机能够以2 880 × 2 048像素的分辨率采集图像,并通过USB 3.0端口传输,采集时的曝光时间设定为1 μs。
在20倍显微镜物镜(CFI PlanFluor,NA = 0.45)下分别采集了600张微球、600张正常的虫黄藻细胞和600张漂白的虫黄藻图像,总的图像数据集大小为1 800张。采用LabelImg软件以PASCAL VOC2007的格式手动标注整个数据集。将总数据集图像和对应的标注文件进行随机打乱,然后将其中20%用于模型训练后的测试(微球:120张;正常虫黄藻:120张;漂白虫黄藻:120张),80%用于训练和验证(微球:480张;正常虫黄藻:480张;漂白虫黄藻:480张)。3个子集之间没有重复的图像。
-
神经网络模型输出的预测框与人为标注的真实框的交集和并集的比值称为交并比(Intersection over Union, IoU)。IoU的值越大说明预测框与真实框越贴合,检测效果越好。根据IoU与预设阈值的比较,可以将图像中的所有目标物分为3类。当神经网络所预测的物体类别与物体实际类别相同,且IoU大于或等于预设阈值时,该预测框被标记为真阳性(True Positive,TP),这意味着该物体被神经网络模型成功分类和定位;当神经网络所预测的类别与实际类别不同,或是IoU小于预设阈值时,该预测框被标记为假阳性(False Positive,FP);此外,对于同一个对象,若产生了多个预测框,且IoU均大于预设阈值,那么只有IoU最大的预测框被标记为TP,其余的框被标记为FP;其余的经过人工标注但未被检测到的目标物被标记为假阴性(False Negative,FN)。根据TP、FP和FN的数量,神经网络模型的性能可以用精确度(Precision, P)、召回率(Recall, R)和F1值来评估。其计算公式如下:
$$ {\rm{Precision}} =\frac{{{N_{{\rm{TP}}}}}}{{{N_{{\rm{TP}}}} + {N_{{\rm{FP}}}}}} $$ (1) $$ {\rm{Recall}} = \frac{{{N_{{\rm{TP}}}}}}{{{N_{{\rm{TP}}}} + {N_{{\rm{FN}}}}}} $$ (2) $$ F1 = \frac{{2 \times P \times R}}{{P + R}} $$ (3) 式中,NTP为神经网络模型成功分类和定位的目标总数;NFP为神经网络模型分类错误或定位错误的目标总数;NFN为神经网络模型未能检测到的目标总数。
某一类别的对象在不同预设阈值下的精确度和召回率的曲线下面积为平均精确度(Average Precision,AP),对所有类别的AP值取平均值,得到mAP(mean Average Precision)。mAP值反映了神经网络模型的整体检测性能,范围从0到100%,值越高说明模型的检测精确度及抗干扰程度越强。
-
采用Python 3.7进行算法编写、神经网络模型构建和训练与数据预处理。神经网络训练的过程采用TensorBoard X记录并可视化。采用OpenCV软件库与GraphPad Prism 8.0软件进行分析作图。
-
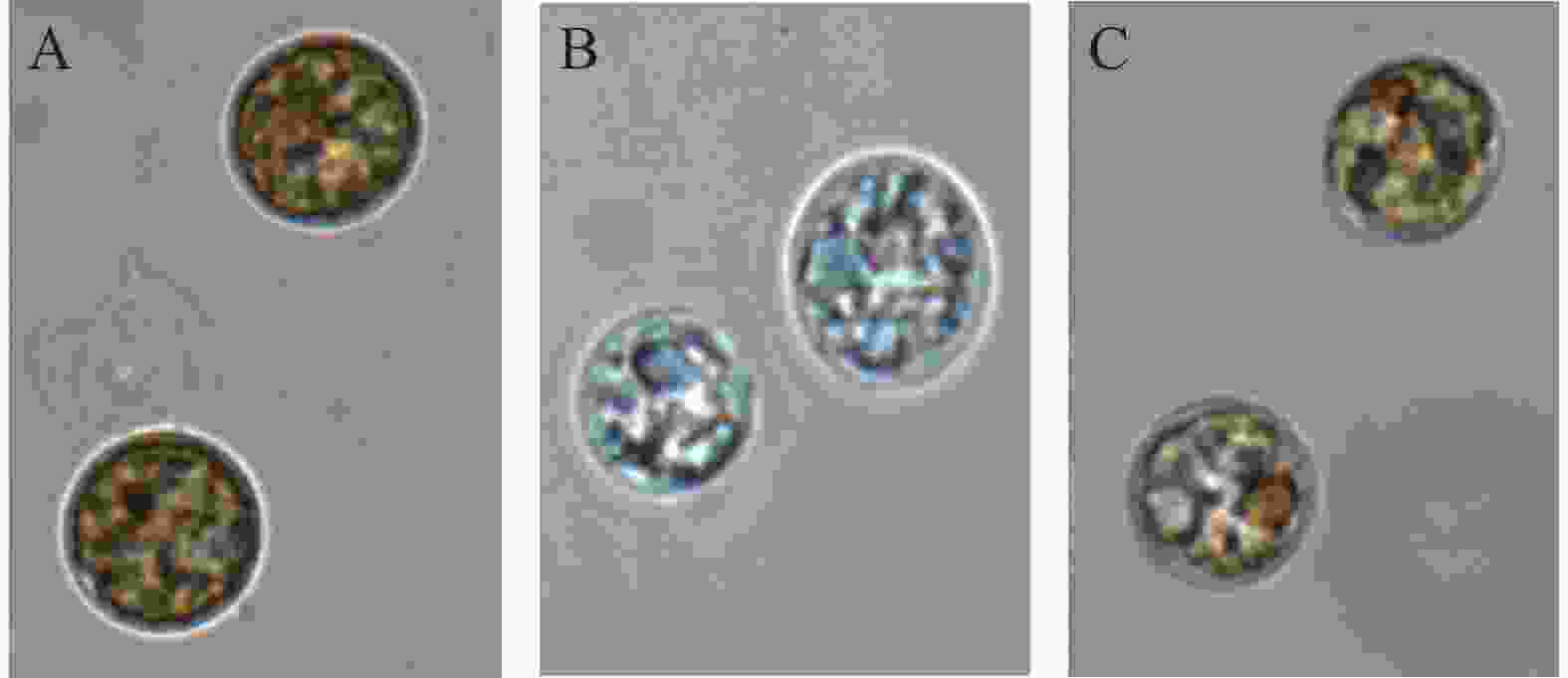
虫黄藻是一种微小的单细胞甲藻,能够进行光合作用。当虫黄藻在正常条件下生长时,它们是黄褐色的(图2-A)。热胁迫后可能会变成白色(图2-B)。在上述2种颜色之间,有一些淡褐色的虫黄藻细胞,处于从正常生长条件到漂白状态的过渡阶段(图2-C)。
-
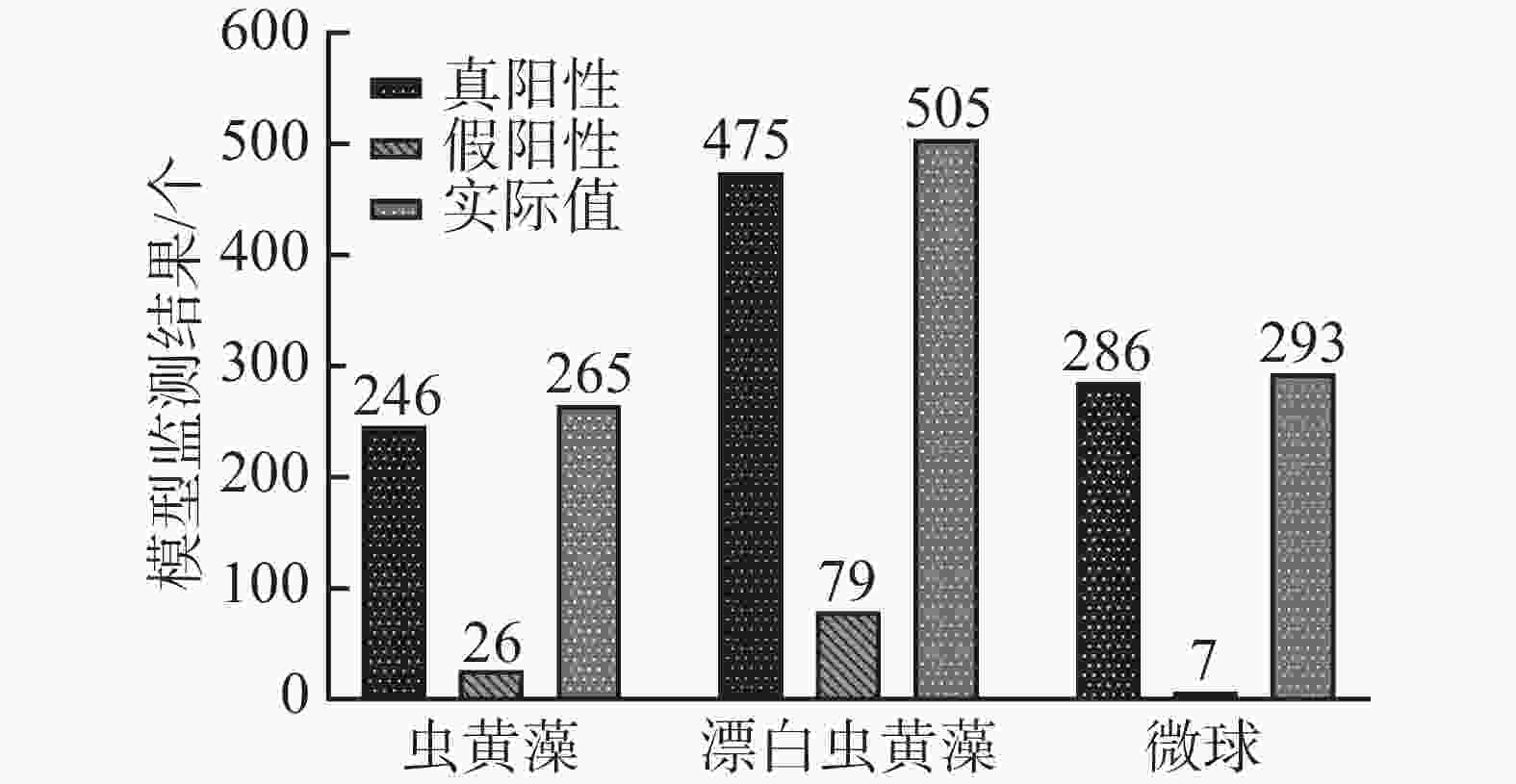
将1 mL正常虫黄藻溶液、2 mL漂白的虫黄藻溶液和1 mL微球溶液充分混合,设置进样流率为0.20 μL·min−1,采集藻类细胞流动的视频数据。随机视频中的抽取300张关键帧,并对每帧图像上的目标细胞进行标注,记作实际值。随后用训练好的YOLOv4甲藻细胞检测模型检测未标注的300帧图像上的目标细胞,检测结果记作检测值。对比分析检测值与真实值的差异并计算mAP值。多目标混合溶液的检测值与实际值如图3所示。
将图3中真阳性样本数和实际值进行对比,结果发现,在测试数据的所有目标对象上,共有19个正常虫黄藻细胞、30个褪色虫黄藻细胞和7和微球为未被正确地识别出来。而假阳性样本,即模型的误检结果中,却有26个虫黄藻细胞、79个褪色的虫黄藻细胞和7个微球。
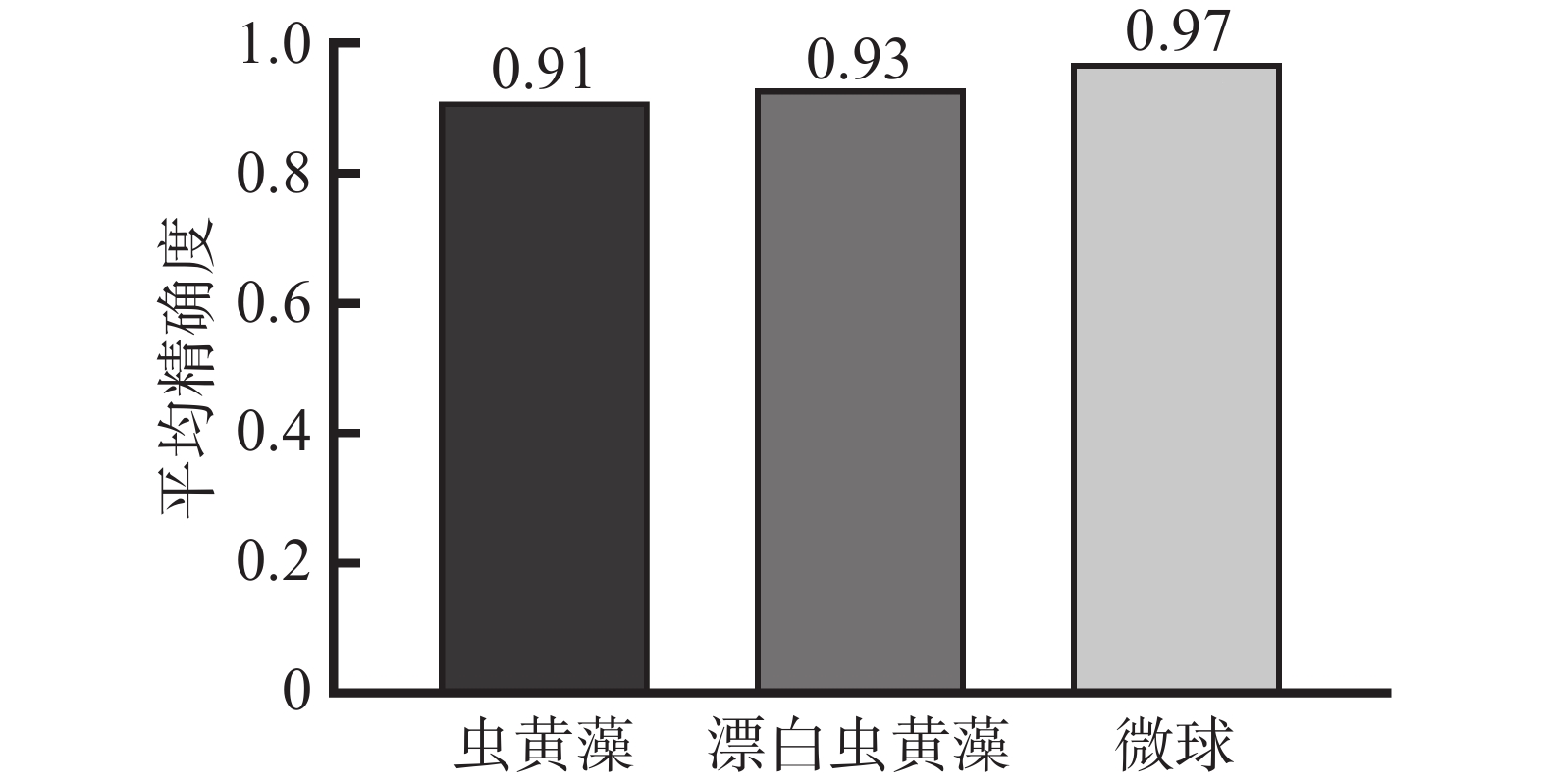
本研究目前采用神经网络模型在对微流控芯片通道内多类别物体检测的mAP值为94%,如图4所示。说明使用明场显微图像进行训练的模型能够较好地泛化至微流控芯片上虫黄藻细胞的实际检测上,且做到精确的检测。正常和漂白的虫黄藻细胞的mAP值为92%左右,说明约有8%的已被人为标注的虫黄藻细胞未能被神经网络模型检测到或检测到但定位错误,造成这种情况的可能原因如下:1)部分虫黄藻细胞没有完全漂白,呈微黄色,或者在热应激下直接死亡,这两种情况都使基于其算法提取虫黄藻细胞特征的深度神经网络模型难以解释;2)在微流控通道中,由于细胞间距小,可能导致细胞之间保持紧密或重叠,导致检测模型将多个细胞识别为单一细胞,导致漏检其他细胞。
-
将本研究中提出的方法(简称为“本方法”)与其他通过分析图像进行微流控芯片上细胞检测的方法对比,结果如表1所示。由于本研究采用的神经网络检测算法可以自动化地跟踪定位相机视场中的每一个目标细胞,因此在溶液进样时可无需设置额外的注射泵与通道进行鞘流聚焦。设计的芯片通道宽度约为待检细胞的8倍,不易发生堵塞,且检测算法可只识别想要检测的细胞类别,具有一定的抗噪能力,因此无需前处理细胞溶液,可直接注入通道中。检测算法将细胞分割、细胞分类与细胞定位集成在一个深度学习神经网络模型中,可以以93.85%平均精确度实现多目标分类,但本方法还不能将检测分类后的细胞进行精确分选,这尚需进一步完善。
表 1 本方法与其他图像激活的微流控细胞检测方法对比
综上所述,与正常的虫黄藻细胞相比,在2 h热胁迫下漂白的虫黄藻的藻体的形态与结构特征基本不发生改变。笔者提出的基于深度神经网络的活体虫黄藻检测方法以92%精确度从两者混合的虫黄藻溶液中识别出正常的虫黄藻,并且还能以94%平均精确度区分出细胞与非细胞目标,展现了基于深度神经网络的细胞检测方法在多种类混合微藻溶液、异质性微藻溶液乃至人体细胞上进行自动化实时快检的潜力。
-
在对海洋微藻细胞的检测上,往往无法做到实时性,固定剂与运输过程中的理化因素影响将导致样品中的部分藻类死亡,而目前的检测手段主要通过人为镜检,不仅检测结果受主观判断的影响大、步骤繁琐、效率低下,而且检测后的样本无法进行后续的细胞分析。此外,一些自动化检测设备存在着系统复杂庞大、价格昂贵且前处理耗时长等问题。
基于此,笔者结合了计算机视觉领域的目标检测算法,微流控技术和显微成像技术,实现了对不同生理阶段的细胞和多类别细胞的自动检测与分类。本实验在明场显微镜下采集了微球、正常培养与热胁迫下的虫黄藻细胞图像,训练神经网络模型并应用于微流控芯片上的细胞多分类检测与定位。结果表明:模型对微流控芯片通道内多类别目标检测的平均精确度可达94%,并且能够以98%的精确度在混合样本中区分出细胞样本与非细胞样本,以92%精确度检测出细胞样本的不同生理状态。本方法无需在微流控芯片上添加鞘流通道且无需前处理,通过目标检测神经网络实现了高精度的细胞多分类与定位,不仅方便了下游的细胞分析,也为细胞分类检测提供了新的思路与视角。
A deep neural network-based method for on-chip detection of in vivo Symbiodinium
-
摘要: 为了对活体虫黄藻进行快速、无标签和低成本的检测,笔者结合微流控技术、显微图像处理和深度学习神经网络,提出了基于深度神经网络的活体虫黄藻检测方法,对混合了微球、正常与漂白虫黄藻细胞溶液进行检测,结果表明:使用明场显微图像进行训练的神经网络模型泛化至微流控芯片上细胞检测当中,且能以93.9%的平均识别精确度识别不同生理状态下的虫黄藻细胞与其他目标,说明该方法可从大量复杂、异质的细胞群体中快速准确地识别出目标细胞。Abstract: Measuring the species and density of Symbiodinium is important for predicting coral bleaching. At present, the rapid detection method of microalgae cells has great limitations. Manual microscopic inspection is time-consuming and laborious, while benchtop automated instruments are not suitable for large-scale real-time detection during field sampling. This research combines microfluidics, microscopic image processing and a deep learning neural network to proposes a deep neural network-based method for the microfluidic detection of in vivo Symbiodinium. The results of the detection of solution samples mixed with polymethylmethacrylate microspheres, normal and bleached methanogens cells show the neural network model trained using bright-field microscopic images is generalized to microfluidic on-chip cell detection, and is able to identify different physiological states of Symbiodinium cells and other objects with an average precision of 93.9%, demonstrating the feasibility, accuracy and sensitivity of the method.
-
Key words:
- neural network /
- Symbiodinium /
- microfluidic chip /
- computer vision /
- symbiotic microalgae
-
[1] LESSER M P J S N. Coral bleaching: causes and mechanisms[M]. Berlin: Springer Netherlands, 2011. [2] STAMBLER N. Coral symbiosis under stress[M]. Berlin: Springer Netherlands, 2010. [3] HUGHES T P, BAIRD A H, BELLWOOD D R, et al. Climate change, human impacts, and the resilience of coral reefs [J]. Science, 2003, 301(5635): 929 − 933. doi: 10.1126/science.1085046 [4] WICKS L C, HILL R, DAVY S K. The influence of irradiance on tolerance to high and low temperature stress exhibited by Symbiodinium in the coral, Pocillopora damicornis, from the high-latitude reef of Lord Howe Island [J]. Limnology And Oceanography, 2010, 55(6): 2476 − 2486. doi: 10.4319/lo.2010.55.6.2476 [5] LITTMAN R A, OPPEN M, WILLIS B L. Methods for sampling free-living Symbiodinium (zooxanthellae) and their distribution and abundance at Lizard Island (Great Barrier Reef) [J]. Journal of Experimental Marine Biology and Ecology, 2008, 364(1): 48 − 53. doi: 10.1016/j.jembe.2008.06.034 [6] OZDALGIC B, USTUN M, DABBAGH S R, et al. Microfluidics for microalgal biotechnology [J]. Biotechnol Bioeng, 2021, 118(4): 1545 − 1563. doi: 10.1002/bit.27671 [7] ANTONACCI A, SCOGNAMIGLIO V. Biotechnological advances in the design of algae-based biosensors [J]. Trends Biotechnol, 2020, 38(3): 334 − 347. doi: 10.1016/j.tibtech.2019.10.005 [8] WANG J, WANG G, CHEN M, et al. An integrated microfluidic chip for treatment and detection of microalgae cells [J]. Algal Research, 2019, 42: 101593. doi: 10.1016/j.algal.2019.101593 [9] WANG Y, WANG J, ZHOU C, et al. A Microfluidic prototype system towards microalgae cell separation, treatment and viability characterization [J]. Sensors (Basel), 2019, 19(22): 4940. doi: 10.3390/s19224940 [10] WANG Y, WANG J, WU X, et al. Dielectrophoretic separation of microalgae cells in ballast water in a microfluidic chip [J]. Electrophoresis, 2019, 40(6): 969 − 978. doi: 10.1002/elps.201800302 [11] YUAN Q, MIRZAJANI H, EVANS B, et al. A disposable bulk-acoustic-wave microalga trapping device for real-time water monitoring [J]. Sensors And Actuators B-Chemical, 2020, 304: 127388. doi: 10.1016/j.snb.2019.127388 [12] RAYMOND S J, COLLINS D J, O'RORKE R, et al. A deep learning approach for designed diffraction-based acoustic patterning in microchannels [J]. Sci Rep, 2020, 10: 8745. doi: 10.1038/s41598-020-65453-8 [13] STOECKLEIN D, LORE K G, DAVIES M, et al. Deep learning for flow sculpting: insights into efficient learning using scientific simulation data [J]. Sci Rep, 2017, 7: 46368. doi: 10.1038/srep46368 [14] FEIZI A, ZHANG Y, GREENBAUM A, et al. Rapid, portable and cost-effective yeast cell viability and concentration analysis using lensfree on-chip microscopy and machine learning [J]. Lab Chip, 2016, 16(22): 4350 − 4358. doi: 10.1039/C6LC00976J [15] ISOZAKI A, HARMON J, ZHOU Y, et al. AI on a chip [J]. Lab Chip, 2020, 20(17): 3074 − 3090. doi: 10.1039/D0LC00521E [16] NITTA N, SUGIMURA T, ISOZAKI A, et al. Intelligent image-activated cell sorting [J]. Cell, 2018, 175(1): 266 − 276. doi: 10.1016/j.cell.2018.08.028 [17] KIM K, KIM S, JEON J S. Visual estimation of bacterial growth level in microfluidic culture systems [J]. Sensors (Basel), 2018, 18(2): 447. doi: 10.3390/s18020447 [18] ZHANG Z, CHEN L, WANG Y, et al. Label-free estimation of therapeutic efficacy on 3D cancer spheres using convolutional neural network image analysis [J]. Anal Chem, 2019, 91(21): 14093 − 14100. doi: 10.1021/acs.analchem.9b03896 [19] MATSUMOTO Y, SAKAKIHARA S, GRUSHNIKOV A, et al. A microfluidic channel method for rapid drug-susceptibility testing of Pseudomonas aeruginosa [J]. PLoS One, 2016, 11(2): e0148797. doi: 10.1371/journal.pone.0148797 [20] FUKUSHIMA K. Training multi-layered neural network neocognitron [J]. Neural Netw, 2013, 40: 18 − 31. doi: 10.1016/j.neunet.2013.01.001 [21] LECUN Y, BENGIO Y, HINTON G. Deep learning [J]. Nature, 2015, 521(7553): 436 − 444. doi: 10.1038/nature14539 [22] HAASEN D, SCHOPFER U, ANTCZAK C, et al. How phenotypic screening influenced drug discovery: lessons from five years of practice [J]. Assay Drug Dev Technol, 2017, 15(6): 239 − 246. doi: 10.1089/adt.2017.796 [23] MATTIAZZI USAJ M, STYLES E B, VERSTER A J, et al. High-content screening for quantitative cell biology [J]. Trends Cell Biol, 2016, 26(8): 598 − 611. doi: 10.1016/j.tcb.2016.03.008 [24] XU M, HARMON J, YUAN D, et al. Morphological indicator for directed evolution of Euglena gracilis with a high heavy metal removal efficiency [J]. Environ Sci Technol, 2021, 55(12): 7880 − 7889. doi: 10.1021/acs.est.0c05278 [25] GUO B, LEI C, KOBAYASHI H, et al. High-throughput, label-free, single-cell, microalgal lipid screening by machine-learning-equipped optofluidic time-stretch quantitative phase microscopy [J]. Cytometry A, 2017, 91(5): 494 − 502. doi: 10.1002/cyto.a.23084 [26] CHEN C L, MAHJOUBFAR A, TAI L C, et al. Deep learning in label-free cell classification [J]. Sci Rep, 2016, 6: 21471. doi: 10.1038/srep21471 [27] XIONG B, HONG T Q, SCHELLHORN H, et al. Dual-modality imaging microfluidic cytometer for onsite detection of phytoplankton [J]. Photonics, 2021, 8(10): 435. doi: 10.3390/photonics8100435 [28] WANG J, YU X, WANG Y, et al. Detection of viability of micro-algae cells by optofluidic hologram pattern [J]. Biomicrofluidics, 2018, 12(2): 024111. doi: 10.1063/1.5021179 [29] GRCS Z, TAMAMITSU M, BIANCO V, et al. A deep learning-enabled portable imaging flow cytometer for cost-effective, high-throughput, and label-free analysis of natural water samples [J]. Light Sci Appl, 2018, 7: 66. doi: 10.1038/s41377-018-0067-0 [30] BOCHKOVSKIY A, WANG C Y, LIAO H. YOLOv4: optimal speed and accuracy of object detection [J]. arXiv e-prints, 2020, arXiv: 2004.10934. [31] GIRAULT M, KIM H, ARAKAWA H, et al. An on-chip imaging droplet-sorting system: a real-time shape recognition method to screen target cells in droplets with single cell resolution [J]. Sci Rep, 2017, 7: 40072. doi: 10.1038/srep40072 [32] HEO Y J, LEE D, KANG J, et al. Real-time image processing for microscopy-based label-free imaging flow cytometry in a microfluidic chip [J]. Sci Rep, 2017, 7: 11651. doi: 10.1038/s41598-017-11534-0 -




 下载:
下载: