-
炭疽菌(Colletotrichum spp.)是一类分布范围广泛的植物病原真菌[1]。橡胶树炭疽病在橡胶种植园区普遍发生,是橡胶树的叶部“两病”之一,可造成叶片脱落、嫩梢回枯、果实腐烂等一系列症状,导致割胶时间推迟,产胶量下降[2]。在多数植胶园中,橡胶树主要受胶孢炭疽菌复合群(Collectotrichum gloeosporioides species complex
)侵染,部分报道受尖孢炭疽菌复合群(Collectotrichum acutatum species complex)侵染[3]。在我国,橡胶树炭疽菌主要由胶孢炭疽菌复合群下的暹罗炭疽菌(Collectotrichum. siamense)和果生炭疽菌(Collectotrichum fructicola)所引起[4]。 鉴于炭疽菌危害的严重性,其分类问题一直是植物病理学与菌物学研究的热点。炭疽菌繁多的种类,丰富的遗传多态性,复杂的复合种群为在种水平上的鉴定带来了诸多困难。目前,针对炭疽菌的有效分类手段还是以形态学与分子生物学为基础[5]。在形态学分类中,菌落特征、分生孢子、分生孢子梗、产孢细胞形态、附着孢形态以及其他结构为主要鉴定依据,并结合培养特征和寄主范围作为辅助手段[6],但炭疽菌的特征往往随不同的环境条件的变化而改变,即使在纯培养的条件下,分生孢子与分生孢子梗、附着孢和菌丝的形态与大小也未必是完全相同的。不稳定的因素、种水平上相似的结构特征与繁琐的实验流程影响了炭疽菌的快速识别与鉴定。基因序列分析满足了快速、准确、灵敏的分类要求,例如核糖体DNA内转录间隔区(Internal transcribed spacer, ITS)序列是炭疽菌分子手段鉴定中使用最广泛的片段[7-10],但基于ITS的序列分析依然存在不准确性,对于复合群间或复合群内的近缘相似种,单基因序列的分析并不能准确反应其亲缘关系,因此无法在种水平上准确识别。多基因系统分析研究物种分离、群体构成以及群体的系统进化亲缘关系已经成为流行趋势。结合形态学基础与利用多基因序列分析可以更精准地将炭疽菌鉴定至属以下水平,因此多基因谱系分析被越来越广泛地在炭疽菌分类研究中使用[11-12]。但多基因序列分析需要依靠庞大的DNA条形码数据库来运作,需要一定生物信息学基础,整个流程也需要不少的人力与时间成本。
拉曼散射是一种与入射光频率不同的非弹性散射,通过拉曼散射可以准确地获取样品的分子结构信息,对于构成样品体内的基本生化物质的检测非常灵敏,因此拉曼光谱被称作“指纹”图谱[13-15]。拉曼光谱仪与共聚焦显微技术的有机结合被称为共聚焦显微拉曼技术,其为物种区分与鉴定带来了新的途径。共聚焦显微拉曼技术利用代谢物的拉曼散射以快速、免处理、无损、宽谱带的方式提供样本所含的拉曼特征信号,例如碳水化合物、脂质、蛋白质以及核酸等均可生成特定的拉曼光谱,进而可了解生物体内的物质成分与结构信息。共聚焦显微拉曼技术仅采集物镜焦点区域的拉曼信号,通过拉曼光谱和共聚焦显微镜的结合,将焦点以外的背景信号拒之门外,更有利于小体积样本的扫描分析,例如细胞或孢子;此外,生物体内的水分是在其他光谱测量中重要的干扰因素,而水的拉曼峰是很弱的,几乎不会对检测结果造成干扰,这对含水的微生物样本的检测具有一定的优势,因此共聚焦显微拉曼技术已经被用于真菌与细菌的分类鉴定中[16-18]。例如,甘琴华等[19]使用共焦拉曼显微技术在变种水平上进行细菌的免培养检测,鉴定的效率与准确性不亚于16s rDNA。Evelin 等[20-21]使用拉曼光谱技术在属和种水平上实现了人类皮肤癣菌以及建筑物损坏相关真菌的检测和鉴定。然而,基于共聚焦拉曼显微技术的检测和鉴定在植物病原真菌孢子的研究上,尤其在炭疽菌分类研究中鲜有报道。在本研究中,笔者利用共聚焦显微拉曼光谱仪对造成橡胶树炭疽病的3种炭疽菌孢子的拉曼光谱进行了扫描,并以一种非橡胶树炭疽菌孢子作为对照,分析了其拉曼光谱特征,并结合主成分分析(PCA)实现了3种炭疽菌在种水平上的区分,表明了占据主要区分主要权重所依靠的3个主要峰值。本研究旨在为病原真菌孢子的鉴别与分类提供高效、可靠、灵敏的诊断方法。
-
热带炭疽菌(Colletotrichum tropicale)菌株HN15、暹罗炭疽菌(Colletotrichum siamense)菌株HN08、尖孢炭疽菌(Colletotrichum acutatum)菌株HN02,这3种橡胶树炭疽菌均为本实验室保存菌种。将这3种橡胶树炭疽菌与对照即非橡胶树炭疽菌(Colletotrichum alatae)接种在PDA培养基上,放置于28 ℃下培养。接种3 d后,刮去表面菌丝破坏菌落结构,在光照条件下进行诱导产孢。3 d后使用ddH2O洗下孢子并通过Miracloth滤布(Merck KgaA, 孔径22~25 μm, Germany)过滤去除杂质。继而收集孢子悬浮液,孢子悬浮液在10 000 r·min−1下离心3 min并去除上清,ddH2O洗涤2次,稀释至105 CFU·mL−1。取孢子悬浮液20 μL(≈ 2×103个孢子) 滴在抛光一面的单晶硅基底上(1 cm × 1 cm),吹干备用。
-
使用共聚焦显微拉曼光谱仪(inVia Reflex of Renishaw, England)对炭疽菌孢子进行拉曼光谱测量,配备了可产生514 nm激发光的50 mW激光光源。在测量前使用单晶硅位于520 cm−1的拉曼峰校准仪器,使用50倍物镜采集单个孢子的拉曼光谱,光谱采集范围为600 ~ 2 000 cm−1,作用在样品上的最终激光功率约为5 mW,光斑直径为1 μm,采集时间为10 s,依靠2 400 cm−1 光栅可实现光谱分辨率为1 cm−1。
每种炭疽菌的测量包含3个生物学重复,每个生物学重复实验取视野中随机的10个孢子来测量拉曼光谱,为了避免激光照射损伤,每个孢子只采集1次光谱。最后使用每种真菌收集到的30张光谱进行数据分析。此外,随机选取30个空白处作为基底的背景光谱。
-
由于背景噪声、孢子本身的差异、实验设备的影响,同种炭疽菌孢子的拉曼光谱可能会略微有所不同。同时,孢子本身的荧光背景、随机和非相关因素也可能会影响收集到的光谱,例如产生基线升高和杂峰。因此,需对光谱数据进行统一的前处理。其中包括减去单晶硅基底光谱,使用Labspec 5(HORIBA Scientific, Orsay, France) 进行曲线平滑、基线校正(5阶,Polymon)。使用EXCEL (Microsoft, USA)计算实验采集范围内每个波数对应拉曼强度的标准偏差以评估光谱的精确性和可重复性,并以阴影部分表示。使用Origin 2021b(教育版,OriginLab,USA)进行作图和PCA分析。
-
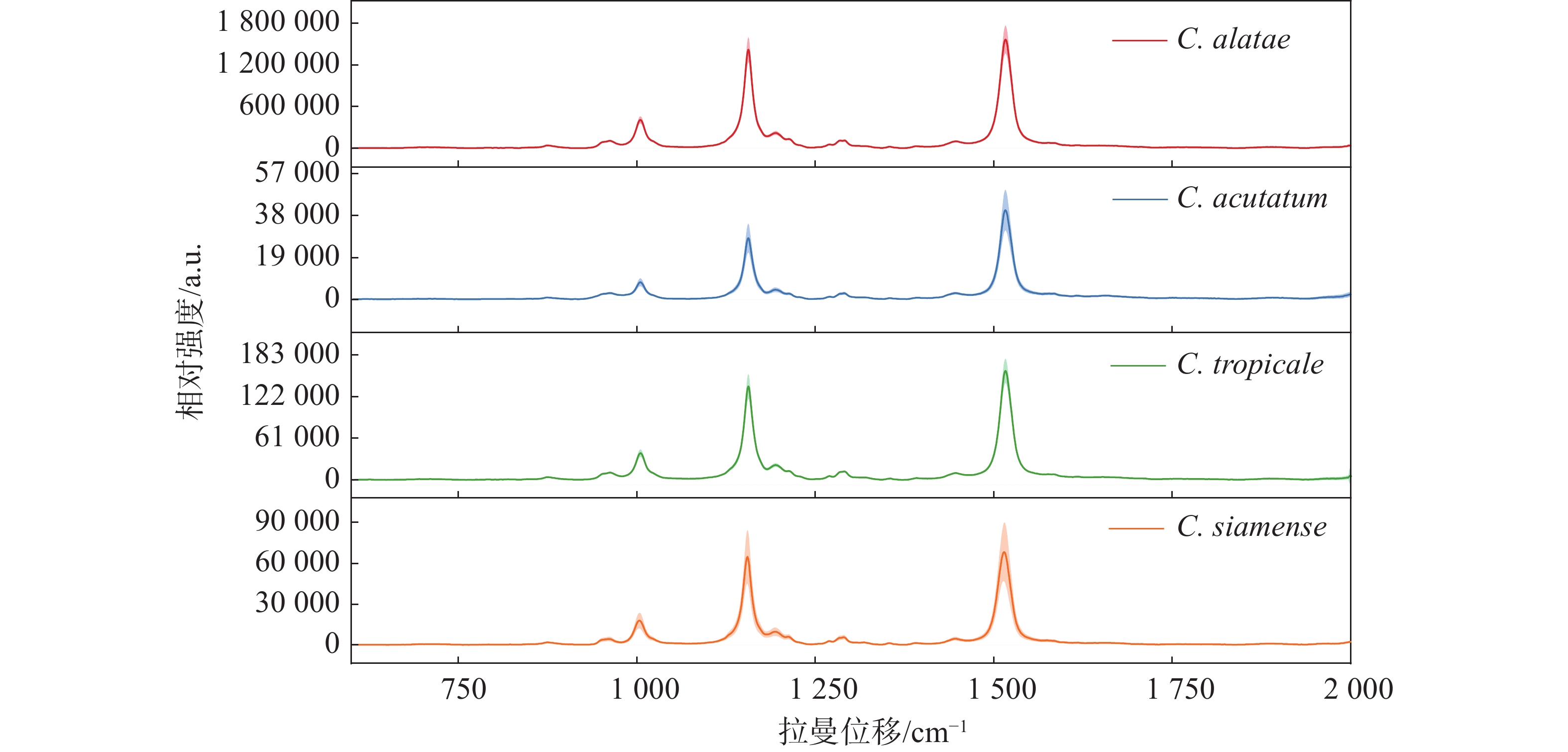
在测量的4种炭疽菌中,热带炭疽菌(C. tropicale)和暹罗炭疽菌(C. siamense)同属于胶孢炭疽菌复合群(C. gloeosporioides species complex),而尖孢炭疽菌(C. acutatum)属于尖孢炭疽复合群(C. acutatum species complex),作为不同寄主对照的非橡胶树炭疽菌(C. alatae)也同属于胶孢炭疽菌复合群(C. gloeosporioides species complex)。对热带炭疽菌(C. tropicale)、暹罗炭疽菌(C. siamense)、尖孢炭疽菌(C. acutatum)以及非橡胶树炭疽菌(C.alatae)孢子的光谱扫描的结果(图1)显示,4种炭疽菌的孢子几乎具有相同的光谱模式和峰位。不同的是,非橡胶树炭疽菌(C. alatae)孢子产生了极强的拉曼信号,为了避免由不同发育阶段或生长状态所带来的测量误差,对其不同阶段均进行了测量,结果与之前一致,说明其具有极强的拉曼信号,这或许是由不同寄主或种间差异所导致。相同的光谱模式表明它们产生拉曼峰的内源性物质组成是几乎一致的,区别在于含量高低与组成比例的不同,导致产生的拉曼信号强度与峰值比例不同。其最重要的特征在于,4种炭疽菌孢子的拉曼光谱均在3个相同的波数处共有3个主要的特征峰,分别位于1 005、1 155、1 515 cm−1。同时,笔者推测相同的拉曼光谱模式与特征峰位置可能是众多炭疽菌所共有的,而不仅限于实验中所测量的复合群与种类。根据以往的报道,1 005 cm−1处的峰值被认为由细胞质蛋白中的苯丙氨酸的分子环呼吸振动的贡献[22-24];1 155 cm−1处的峰值代表着细胞壁中C-C和C-N拉伸[18-25];1 515 cm−1处峰值则由N-H键、C-H键和C=C双键伸缩所导致[26]。
除此以外,根据它们的平均拉曼光谱还观察到了位于960、1 194、1 290、1 446 cm−1的几处共有的小峰。960 cm−1处峰值由C-N键伸缩与C=C键变形所产生;1 194 cm−1处的峰值由芳香族氨基酸产生;1 290 cm−1处的峰值由酰胺III和胸腺嘧啶产生;1 446 cm−1处峰值由脂肪链的C-H键弯曲产生[22-24,27]。虽然4种炭疽菌孢子显示出了相同的光谱趋势和几乎相同的峰值位置,但3个主要波数1 005、1 155和1 515 cm−1处的峰值强度明显不同。同时,即使受孢子本身状态与仪器光电噪声的影响,它们的平均拉曼光谱依然显示出了较小的标准误差范围(以阴影部分表示)。此外,不仅仅是3个主要峰值的强度不同,其峰值之间的比例也不同。例如,尖孢炭疽菌(C. acutatum)、热带炭疽菌(C. tropicale)和暹罗炭疽菌(C. siamense)孢子的拉曼光谱在1 155和1 515 cm−1这2个波数处的峰值强度比也是不同的,分别为0.69、0.86和0.95,这也导致了同一张平均光谱中不同峰位对应的峰面积占总体峰面积的比例不同,比如尖孢炭疽菌(C. acutatum)在1 155 cm−1处对应峰面积所占总体峰面积的比例明显小于其他3种。3个主要峰值的信号强度差异、峰值比例与光谱趋势成为了区分它们的良好依据。
-
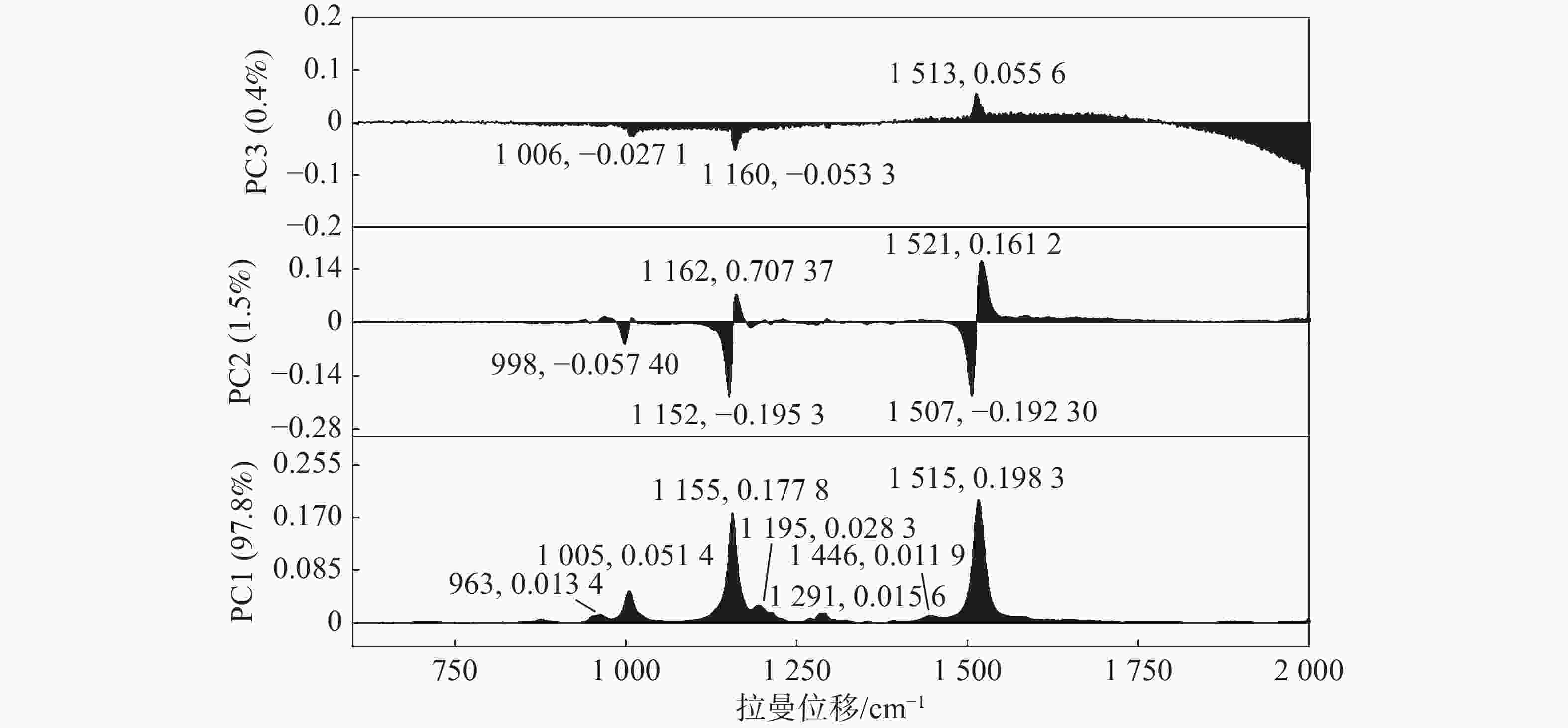
使用PCA对3种橡胶树炭疽菌孢子预处理后的拉曼光谱进行聚类分析,而鉴于非橡胶树炭疽菌(C. alatae)的拉曼光谱具有肉眼可辨的极强的峰值特征,因此未将其包含在PCA聚类分析中。依靠每张孢子拉曼光谱的PC1、PC2和PC3得分,将散点图投影到三维空间中,位置和大小反映了孢子的分类关系。该投影分析(图2)显示了在种水平上较高的灵敏度,3种橡胶树炭疽菌孢子的拉曼光谱可以准确地划分为3个簇。它们的95%置信区间有一定程度的重叠,这是由于一些不稳定的样本因素所导致的,整体来看并不影响聚类效果。图3所示,前3个主成分共同占据了99.70%的解释度,其中,依靠1 005 cm−1,1 155 cm−1和 1 515 cm−1 3个主要峰值的差异,PC1占据了97.76%的分辨能力。位于963,1 195,1 291,1 446 cm−1处的几个小的峰值也在PC1中占据了一定的权重,但不如3个主要峰值明显。同时,PC2与PC3这两个主成分也能在一定程度上有效解释区分度,例如,998、1 152和1 507 cm−1在PC2与PC3的负轴上也占据了一定的权重。但是,在PCA特征提取的过程中,更倾向于自动选择降维后较大的特征值与特征向量。总体而言,3种橡胶树炭疽菌孢子的拉曼光谱很容易聚类为3个簇,表明使用拉曼光谱技术在种水平上区分炭疽菌孢子种类是可行的。
-
本研究依靠共聚焦显微拉曼技术对3种橡胶树炭疽菌孢子以及1种非橡胶树炭疽菌孢子的拉曼光谱进行了特征分析,找出了共有的1 005、1 155和1 515 cm−1波数处3个主要拉曼峰,以及位于960、1 194、1 290、1 446 cm−1处的次级拉曼峰,继而对峰值贡献的归属进行了初步判断。结合PCA聚类分析在三维空间得分图中快速有效地将3种橡胶树炭疽菌孢子聚类为3个簇,在荷载图中展示了区分它们在PC1、PC2以及PC3中主要依靠的拉曼峰。结果表明,1 005、1 155和1 515 cm−1 波数处3个拉曼峰不仅是炭疽菌所共有的,更是利用拉曼光谱结合PCA在种水平上区分它们的主要依据。
本研究提供了一种鉴别炭疽菌孢子的全新的方法,与基于形态学和分子生物学的鉴定方法相比,共聚焦显微拉曼技术具有灵敏与微量样品需求的优势,且样品是无需预富集和预处理的,有望达到单孢子水平的检测与鉴定。本研究为病害防控,快速鉴别,真菌分类提供有力的技术支撑。当然,为了提高诊断的可靠性和可选择性,更多的种类炭疽菌孢子样本应在的实验中进一步引入,以便建立完整的孢子光谱数据库。
Raman spectroscopic characteristics of Colletotrichum spores associated with anthranose of rubber tree and their application in cluster analysis
-
摘要: 为了弥补分类学上单纯依靠形态学与分子生物学鉴定炭疽菌的不足,基于共聚焦显微拉曼技术的橡胶树炭疽菌3个种群孢子聚类分析方法,对橡胶树炭疽菌孢子的拉曼光谱进行扫描,找到了其共有的位于1 005 cm−1, 1 155 cm−1和1 515 cm−1处的3个主要的拉曼峰以及其他次级峰,并对峰的产生来源进行初步确认。结合拉曼光谱与主成分分析(PCA),依靠3个主要的拉曼峰以及其他次级峰在PC1、PC2以及PC3的三维空间得分图中快速有效地将3种橡胶树炭疽菌孢子区分开来,为鉴别炭疽菌孢子提供了一种新方法,满足了灵敏与微量的要求,且样品无需预富集和预处理。Abstract: In order to solve the shortcomings of traditional identification of Colletotrichum spp. by morphology and molecular biology, a clustering method based on confocal Raman microscopy was proposed for identification of spores of three Colletotrichum species causing anthracnose of rubber trees. Raman spectra of the spores were scanned to show their spectral trends and peak characteristics, and three main Raman peaks at 1005 cm−1, 1155 cm−1, and 1515 cm−1 in common and other secondary peaks were found to preliminary confirm the contributing source of the peaks. Then, combined with Raman spectroscopy and principal component (PCA) analysis, the spores of three Colletotrichum species were quickly and efficiently distinguished by the three key Raman peaks and other secondary peaks in the 3D score plot of PC1, PC2, and PC3 components. This method provides a new way for discrimination and identification of Colletotrichum spores of the anthracnose of rubber trees, and satisfies the sensitivity and trace requirements without pre-enrichment and pre-treatment of the samples.
-
[1] ARZANLOU M, BAKHSHI M, KARIMI K, et al. Multigene phylogeny reveals three new records of Colletotrichum spp. and several new host records for the mycobiota of Iran [J]. Journal of Plant Protection Research., 2015, 55(2): 198 − 211. doi: 10.1515/jppr-2015-0027 [2] LIU X B, LI B X, CAI J M, et al. Colletotrichum species causing anthracnose of rubber trees in China [J]. Scientific Reports, 2018, 8(1): 10435. doi: 10.1038/s41598-018-28166-7 [3] 林春花, 孙董董, 韩丹, 等. 中国橡胶树苗圃2种炭疽病菌分子鉴定及分布分析[J]. 热带作物学报, 2014, 35(9): 1802 − 1808. doi: 10.3969/j.issn.1000-2561.2014.09.024 [4] 连文旭, 王萌, 张宇, 等. 橡胶树胶孢炭疽菌复合群LAMP检测方法的建立及应用[J/OL]. 植物病理学报, [2021−12−17]. https://doi.org/10.13926j.cnki./apps.000780. [5] 向梅梅, 张云霞, 刘霄. 炭疽菌属真菌分类的研究进展[J]. 仲恺农业工程学院学报, 2017, 30(1): 60 − 66. [6] 王晓鸣. 陕西省炭疽菌的研究[J]. 真菌学报, 1987, 6(4): 21 − 28. [7] SREENIVASAPRASAD S, MILLS P R, MEEHAN B M, et al. Phylogeny and systematics of 18 Colletotrichum species based on ribosomal DNA spacer sequences [J]. Genome, 1996, 39(3): 499 − 512. doi: 10.1139/g96-064 [8] MORIWAKI J, TSUKIBOSH T, SATO T. Grouping of Colletotrichum species in Japan based on rDNA sequences [J]. Journal of General Plant Pathology, 2002, 68(4): 307 − 320. doi: 10.1007/PL00013096 [9] 彭仁, 习平根, 曾大兴, 等. 我国南方姜科作物炭疽病菌的鉴定及ITS序列分析[J]. 华南农业大学学报(自然科学版), 2008, 24(2): 194 − 196. [10] 刘晓倩, 祁建民, 陈玉森, 等. 中国红麻炭疽病病原菌的分离鉴定及rDNA-ITS序列分析[J]. 中国农业科学, 2012, 45(17): 3515 − 3521. [11] CARLOS A D, DAMM U, BARONCELLI R, et al. Species of the Colletotrichum acutatum complex associated with anthracnose diseases of fruit in Brazil [J]. Fungal Biology, 2016, 120(4): 547 − 561. doi: 10.1016/j.funbio.2016.01.011 [12] HYDE K, CAI L, CANNON P, et al. Colletotrichum—names in current use [J]. Fungal Diversity, 2009, 39: 147 − 182. [13] MADZHAROVA F, HEINER Z, GÜHLKE M, et al. Surface-enhanced hyper raman spectra of adenine, guanine, cytosine, thymine, and uracil [J]. Journal of Physical Chemistry C:Nanomaterials and Interfaces, 2016, 120(28): 15415 − 15423. doi: 10.1021/acs.jpcc.6b02753 [14] IVLEVA N P, WAGNER M, HORN H, et al. In situ surface-enhanced Raman scattering analysis of biofilm [J]. Analytical Chemistry, 2008, 80(22): 8538 − 8544. doi: 10.1021/ac801426m [15] GELDER J D, GUSSEM K D, VANDENABEELE P, et al. Reference database of Raman spectra of biological molecules [J]. Journal of Raman Spectroscopy, 2007, 38(9): 1133 − 1147. doi: 10.1002/jrs.1734 [16] HUANG W E, GRIFFITHS R I, THOMPSON I P, et al. Raman microscopic analysis of single microbial cells [J], Analytical Chemistry, 2004, 76(15): 4452 - 4458. [17] HUANG W E, LI M Q, JARVIS R M, et al. Chapter 5 - shining light on the microbial world: the application of Raman microspectroscopy [J]. Elsevier Science & Technology, 2010, 70: 153 − 186. [18] HUANG Y S, KARASHIMA T, YAMAMOTO M, et al. Molecular-level investigation of the structure, transformation, and bioactivity of single living fission yeast cells by time- and space-resolved Raman spectroscopy [J]. Biochemistry, 2005, 44(30): 10009 − 10019. doi: 10.1021/bi050179w [19] GAN Q H, WANG X T, YUN W, et al. Culture-free detection of crop pathogens at the single-cell level by micro-Raman spectroscopy [J]. Advanced Science, 2017, 4(11): 1700127. doi: 10.1002/advs.201700127 [20] WITKOWSKA E, JAGIELSKI T, KAMIŃSKA A. Genus- and species-level identification of dermatophyte fungi by surface-enhanced Raman spectroscopy [J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2017, 192(5): 285 − 290. [21] GHOSAL S, MACHER J M, AHMED K. Raman microspectroscopy-based identification of individual fungal spores as potential indicators of indoor contamination and moisture-related building damage [J]. Environmental Science & Technology, 2012, 46(11): 6088 − 6095. [22] NOOTHALAPATI V H N, SHIGETO S. Stable isotope-labeled Raman imaging reveals dynamic proteome localization to lipid droplets in single fission yeast cells [J]. Chemistry & Biology, 2012, 19(11): 1373 − 1380. [23] LIN C C, YANG Y M, LIAO P H, et al. A filter-like AuNPs@MS SERS substrate for Staphylococcus aureus detection [J]. Biosensors & Bioelectronics, 2014, 53(15): 519 − 527. [24] RUAN C, WEI W, GU B. Surface-enhanced Raman scattering for perchlorate detection using cystamine-modified gold nanoparticles [J]. Analytica Chimica Acta, 2006, 567(1): 114 − 120. doi: 10.1016/j.aca.2006.01.097 [25] NOOTHALAPATI H, SASAKI T, KAINO T, et al. Label-free chemical imaging of fungal spore walls by Raman microscopy and multivariate curve resolution analysis [J]. Scientific Reports, 2016(6): 1 − 10. [26] SAYIN I, KAHRAMAN M, SAHIN F, et al. Characterization of yeast species using surface-enhanced Raman scattering [J]. Applied Spectroscopy, 2009, 63(11): 1276 − 1282. doi: 10.1366/000370209789806849 [27] NOOTHALAPATI H, SHIGETO H. Exploring metabolic pathways in vivo by a combined approach of mixed stable isotope-labeled Raman microspectroscopy and multivariate curve resolution analysis [J]. Analytical Chemistry, 2014, 86(15): 7828 − 7834. doi: 10.1021/ac501735c -





 下载:
下载: