-
细菌感染严重威胁全球公共卫生安全,并对农业造成巨大经济损失。副溶血性弧菌、金黄色葡萄球菌、大肠杆菌等常见病原菌[1-2],不仅会导致水产及畜禽患病,还会引发人类皮肤感染、食物中毒、肺炎等多种疾病[3-4]。为应对这一双重挑战,医学与农业领域持续寻求高效、可靠的抗菌手段。1928年青霉素的发现标志着抗生素时代的开端,显著降低了细菌感染的发病率与死亡率,极大地改变了传统医学领域的治疗模式[5]。然而,抗生素的不合理使用,导致大量药物在环境中残留,加速了抗生素耐药菌(antibiotic resistance bacteria,ARB)和抗生素抗性基因(antibiotic resistance genes,ARGs)的出现与传播[6-8]。例如,在水产养殖中,食源性病原菌副溶血性弧菌已被检测出携带 blaCTX-M 和 tetA 等多重耐药基因[9];临床分离的粪肠球菌也显示携带着多种耐药基因[10]。此外,抗生素的广谱特性会破坏正常微生物群,因此开发具有靶向性、安全性的新型抗菌药物迫在眉睫[11]。噬菌体(bacteriophages)是一种宿主专一性强,能够特异性感染并杀死细菌的病毒[12]。噬菌体因其高度的宿主特异性、有效破坏细菌生物被膜及高效的裂解活性等优势被认为是抗生素的有效替代药物[13]。噬菌体高度的特异性能够有效避免对共生菌群的非特异性杀伤,满足了精准医疗的需求[14],但这一特性也限制了噬菌体裂解的宿主范围,导致其难以应对由多种不同种属细菌引起的复杂感染[15]。更为严峻的是,在噬菌体与细菌的长期对抗中,细菌进化出了一系列复杂的防御机制(原核免疫系统),导致噬菌体治疗受到了限制[16]。此外,部分噬菌体可能携带ARGs,这加重了耐药基因传播[17]。研究发现,移动整合子(mobile integrons,MIs)不仅可以捕获ARGs,以增强细菌对抗生素的耐药性,还能整合噬菌体抗性整合子盒(bacteriophage resistance integron cassettes,BRiCs),赋予宿主对噬菌体的防御能力[18]。MIs常位于接合性质粒上,BRiCs可通过接合转移在病原菌间快速传播,使细菌同时获得对抗生素和噬菌体的多重抗性,限制了噬菌体疗法的长期有效性[18]。相比之下,噬菌体裂解酶(bacteriophage lysin)是噬菌体侵染细菌后编码的一类细胞壁水解酶,能够在感染后期从细菌内部裂解细菌细胞壁中高度保守的肽聚糖[19],这种高度保守性导致细菌很难对裂解酶产生抗性[20]。当裂解酶以外源重组蛋白形式作用时,无需依赖噬菌体吸附、注入等复杂感染过程,可从胞外快速裂解不含外膜屏障的革兰氏阳性菌[21-23]。外源表达的噬菌体裂解酶不涉及核酸递送,因而自身不会介导ARGs传播。此外,其模块化结构赋予其优异的可塑性,可通过蛋白质工程手段优化裂解酶的宿主范围、稳定性和活性[24]。基于噬菌体裂解酶独特的优势与应用潜力,噬菌体裂解酶已成为新型抗菌药物研发的热点。本文综述了噬菌体裂解酶成为新型抗菌药物的研究进展,全面概述了噬菌体裂解酶的结构特征与作用机制,并分析了其在应用过程中所面临的挑战。此外,本文还探讨了基于噬菌体裂解酶模块化结构的蛋白质工程改造策略,通过定点突变、截断、重组交换等手段优化裂解酶的性能,以期获得更具广泛抗菌活性、更高稳定性和更强靶向性的噬菌体裂解酶。
-
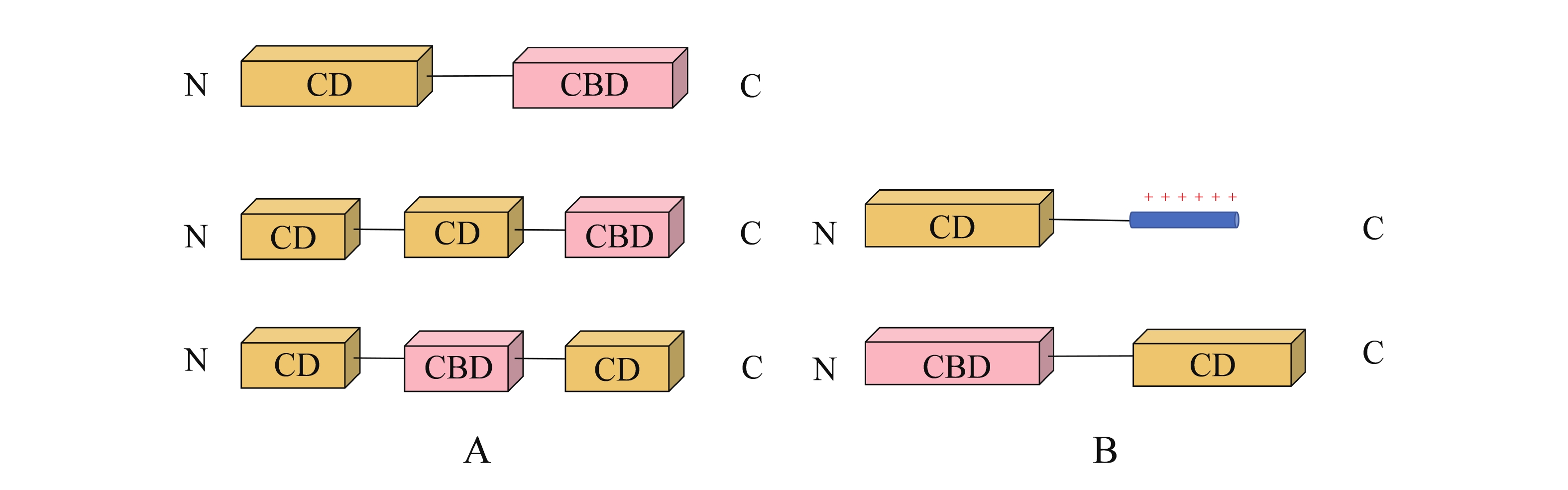
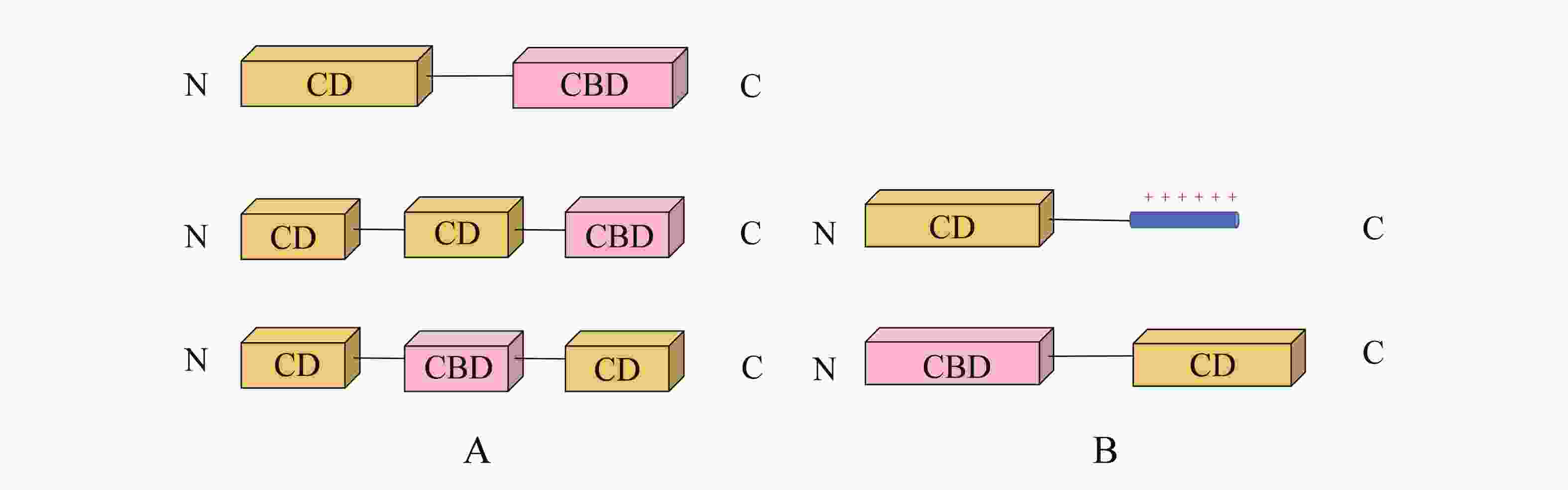
根据细菌细胞壁的结构特点,可将细菌分为具有独特外膜结构的革兰氏阴性菌和无外膜结构的革兰氏阳性菌两大类。革兰氏阳性菌细胞壁由内膜和肽聚糖层两部分组成;而革兰氏阴性菌的细胞壁由三部分组成,从外到内分别是外膜、肽聚糖层和内膜;外膜是不对称的双分子层,由外小叶的脂多糖、外膜蛋白和内小叶的磷脂组成[25]。针对革兰氏阴性和革兰氏阳性细菌的噬菌体裂解酶的结构和作用机制因二者细胞壁结构的差异而有所不同(图1)。针对革兰氏阳性菌的噬菌体裂解酶通常具有模块化结构,由保守的催化结构域(catalytic domain,CD)和细胞壁结合结构域(cell wall binding domain,CBD)组成,二者通过可变长度的短肽连接序列(Linker)连接(图1-A)。CBD与细菌细胞壁上的受体特异性结合,促进CD锚定裂解肽聚糖中的化学键,共同导致细菌细胞壁的裂解[26]。革兰氏阳性菌噬菌体裂解酶通常由一个或多个CD和一个CBD组成,例如金黄色葡萄球菌裂解酶LysP4具有高效的裂解活性,由N端的半胱氨酸-组氨酸依赖性酰胺水解酶/肽酶(cysteine-histidine-dependent amidohydrolases/peptidases,CHAP)、中心的酰胺酶(amidase)两个催化结构域,和一个C端SH3b结合结构域组成[27]。分枝杆菌噬菌体裂解酶LysinA由两个CD和一个CBD组成,无乳链球菌噬菌体裂解酶λSA2的结构由两端的CD和1个中间的CBD组成[28-29]。而后续的研究也发现了含有多个CBD的噬菌体裂解酶,例如,链球菌裂解酶 Ply1228 有两个 CD 和两个 CW-7 CBD[30]。靶向引起人兽共患病的绿色气球菌的噬菌体裂解酶AVPL由一个N端N-乙酰壁酰基-L-丙氨酸酰胺酶催化结构域和C端两个LysM 结合结构域组成[31]。革兰氏阴性菌噬菌体裂解酶通常呈现单结构域球状结构,由单个催化结构域组成,C末端通常带有正电荷(图1-B)[32]。革兰氏阴性菌外膜结构的存在导致外源裂解酶无法与细菌细胞壁直接结合,这也许可以解释革兰氏阴性菌裂解酶大多是不存在CBD的。但少部分的革兰氏阴性菌裂解酶也存在模块化结构,不同的是革兰氏阴性菌的CD一般位于C端,而CBD位于N端,与革兰氏阳性菌裂解酶结构域位置相反(图1-B)[33]。基于噬菌体裂解酶的模块化结构,可以对裂解酶的结构域进行理性设计,开发具有针对性的噬菌体裂解酶。
-
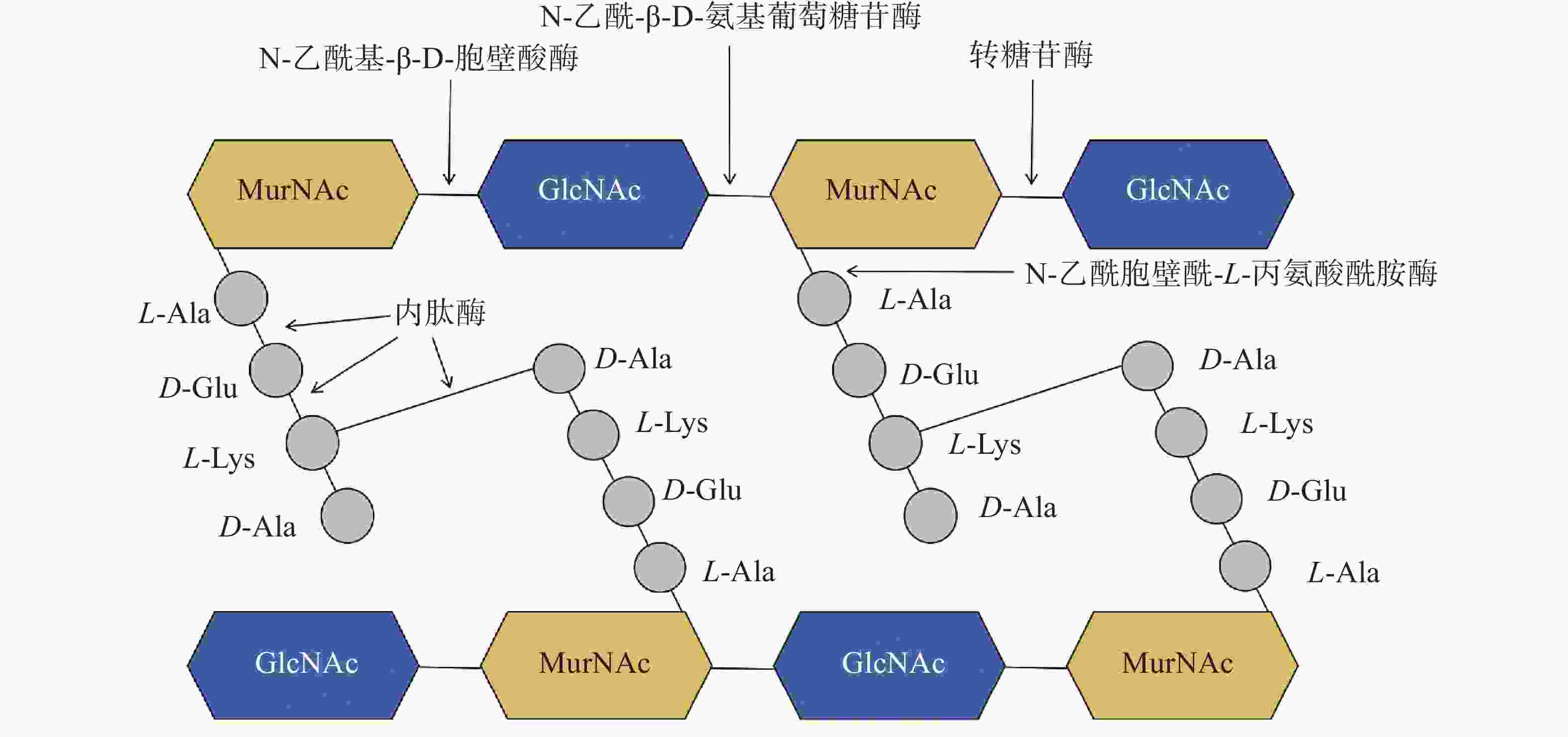
噬菌体裂解酶作为噬菌体侵染细菌后的产物,在宿主内合成后从内到外裂解细菌细胞壁。噬菌体通过吸附细菌完成复制周期后,在细菌内产生裂解酶与穿孔素(Holin),经典裂解机制是由穿孔素先插入到细菌的细胞质膜中,形成孔洞,帮助裂解酶到达肽聚糖层完成对肽聚糖的裂解[34-35]。肽聚糖层是细菌细胞壁的重要组成部分,它由双糖N-乙酰胞壁酸(N-acetylmuramic acid,MurNAc)与N-乙酰-D-葡萄糖胺(N-acetylglucosamine,GlcNAc)通过β-1,4-糖苷键连接形成的线性聚合物网络[36]。这些双糖单位通过短肽进一步交联形成了复杂的网状结构,赋予细胞壁机械强度并决定了细胞的固定形状,有助于维持细胞壁的完整性[37]。CD能够特异地识别并切断肽聚糖中的化学键,破坏肽聚糖骨架结构,促使细菌裂解死亡。根据CD在肽聚糖上的作用位点和功能差异,可以将噬菌体裂解酶分为不同的类型(图2):1)N-乙酰基-β-D-胞壁酸酶,切割从N-乙酰胞壁酸到N-乙酰-D-葡萄糖胺之间的β-1,4糖苷键;2)转糖苷酶,切割位置与N-乙酰-β-D-胞壁酰胺酶相同,但作用机制独特,这种反应不依赖水分子,它们通过催化邻近糖分子内的反应导致糖苷键的断裂,并通过转移糖基团形成新的糖苷键,从而改变肽聚糖多糖链的结构和连接方式;3)N-乙酰-β-D-氨基葡萄糖苷酶,切割从N-乙酰-D-葡萄糖胺到N-乙酰胞壁酸的β-1,4糖苷键;4)N-乙酰胞壁酰-L-丙氨酸酰胺酶,切割N-乙酰胞壁酸和L-丙氨酸之间的酰胺键[38];5)内肽酶,切割肽侧链和肽桥上的肽键。不同细菌物种间肽聚糖的酰胺键和氨基糖苷之间的β-1,4-糖苷键往往是保守的,因此相比于其他几种裂解酶,酰胺酶具有更广泛的裂解谱[39]。
CBD能够特异性地识别并与宿主细菌细胞壁上的特异性底物结合,促进催化结构域的活性,决定裂解酶的裂解谱[40]。CBD在裂解细胞后仍能够结合在细胞壁碎片上,可以阻止裂解酶的扩散,从而保护周围健康的细胞[41]。生物被膜是由细菌自产的细胞外聚合物组成的复杂微生物群落,这些群落被包裹在由多糖、蛋白质、脂质和细胞外DNA(eDNA)构成的自合成层中[42]。生物被膜往往顽固地黏附、聚集于一些生物或非生物表面,以适应环境、保护内部细菌,阻碍了抗生素发挥作用,加剧了细菌对抗生素的耐药性[43],细菌生物被膜对常见抗生素的耐药性比浮游细菌要高出一百倍至数千倍[44]。噬菌体裂解酶与生物被膜之间的研究大多集中在展示裂解酶对生物被膜的破坏效果及抑制生物被膜形成的能力上。Gutierrez等[45]的研究结果表明,噬菌体裂解酶LysH5能够有效去除葡萄球菌生物被膜,且不依赖于生物被膜基质的具体化学组成(无论是多糖、蛋白质还是DNA),这表明裂解酶主要是通过杀死生物被膜中的细菌来破坏生物被膜,而并非直接作用于生物被膜基质。而近期有研究发现,噬菌体裂解酶不仅能够通过降解细菌细胞壁中的肽聚糖来杀灭细菌,还能够与生物被膜中的关键成分(如多糖和eDNA)相互作用,破坏这些结构成分的完整性,从而导致生物被膜的松散和解体[46]。
-
革兰氏阴性菌的外膜不仅阻碍了抗生素,也同样限制了裂解酶的作用。目前报道的大多数天然裂解酶主要针对革兰氏阳性菌,对革兰氏阴性菌的裂解作用有限[47]。脂多糖是一种两亲性分子,由3种结构成分组成,包括疏水性脂质A、亲水性核心寡聚糖和由糖残基重复单位构成的亲水性O-抗原链,脂多糖远端的糖残基部分、脂质A及内核心寡糖含有大量的负电荷[48-50]。脂多糖表面的二价阳离子(Mg2+和Ca2+)会与脂多糖表面所带负电基团发生相互作用,形成一个疏水性化合物几乎不可渗透的高度有序的网络结构,有利于促进外膜的稳定性[51-52]。一些天然的多黏菌素类多阳离子抗生素可通过竞争性置换外膜中稳定的二价阳离子,与裂解酶产生协同抗菌作用,破坏革兰氏阴性菌外膜结构[53-54]。对革兰氏阴性菌具有裂解作用的天然裂解酶通常在C端或者N端携带正电荷,或者含有一个或多个两亲螺旋结构。带正电荷的裂解酶可以通过静电作用与脂多糖的负电荷基团结合,竞争性地取代维持脂多糖结构稳定的二价阳离子,帮助裂解酶穿透外膜屏障。这种电荷间相互作用是裂解酶能够穿透革兰氏阴性菌外膜的关键。Lood等人的研究结果发现裂解酶PlyF307的C末端含有高度正电荷[55],在体外和体内实验中,PlyF307能够显著减少浮游和生物被膜状态下的鲍曼不动杆菌,这也推测出阳离子肽介导的裂解酶可能具有穿透外膜的能力,为裂解酶的工程化设计提供了依据。Larpin等人的研究结果发现[56]的研究结果进一步寻找了与PlyF307类似的裂解酶PlyE146,证实了同样含有高度正电荷的C端结构域的PlyE146对革兰氏阴性菌也具有显著的裂解作用。不仅如此,通过结构的诱变发现即使革兰氏阴性菌裂解酶不存在CBD,通过调整催化结构域的净电荷,也可以在某种程度上弥补缺乏完整CBD所带来的影响,催化结构域上的正电荷仍然可以促进裂解酶与细胞壁的相互作用,保持其杀菌活性[57]。两亲性螺旋结构一部分具有疏水性,可以插入到脂质双层中,而另一亲水性部分则与脂多糖发生相互作用,这种结构特性使得裂解酶能够更容易地穿透革兰氏阴性菌的外膜。研究发现带有两亲性螺旋结构的LysAB2的抗菌率大于99%,当LysAB2的C端两亲螺旋区域被删除后,抗菌率低于40%,印证了两亲螺旋的存在增强了裂解酶的裂解活性[58]。
-
除筛选具有天然外膜穿透能力的噬菌体裂解酶外,利用人工手段破坏外膜完整性,是实现革兰氏阴性菌高效裂解的重要补充策略[59]。一些裂解活性小或没有裂解活性的裂解酶可以利用物理或化学手段破坏细菌的外膜。例如使用高静水压等物理干预,破坏细菌外膜结构,帮助裂解酶裂解细菌[60]。化学方法包括添加膜通透剂,如EDTA、弱酸(如柠檬酸)、阳离子多肽类抗生素(如黏菌素或多黏菌素B)。EDTA可以螯合细胞壁中与负电荷相互作用的阳离子,破坏外膜的稳定性,打破外膜屏障[47];柠檬酸可以通过酸化作用和螯合维持外膜结构稳定的二价阳离子,破坏革兰氏阴性菌外膜的稳定性[61]。阳离子肽类抗生素的正电荷会与脂多糖的负电荷相互作用,竞争性取代维持外膜稳定性的Ca2+和Mg2+,从而破坏外膜的稳定性[53]。有研究将不同长短的阳离子肽与具有多种弧菌裂解活性的裂解酶Lysqdvp001的C端融合,以氨基酸数量依赖型的方式提高了其对副溶血性弧菌的杀菌能力[62]。Ma等人将带正电荷的氨基酸插入裂解酶Lysep3的C端[63],结果发现裂解酶杀菌活性随着C端正电荷数目增加而提升。Mancoš等人将噬菌体裂解酶Lyt μ1/6与源自T4溶菌酶的两亲性阳离子肽融合[64],构建了一种新型嵌合型裂解酶LytAmfi,LytAmfi不仅保留了Lyt μ1/6对金色链霉菌等革兰氏阳性菌的高效裂解活性,而且对革兰氏阴性菌的裂解活性范围也有所扩大。有研究利用天然裂解酶的两亲性螺旋结构,将含有两亲性螺旋结构的抗菌肽天蚕肽Cecropin A的37个氨基酸序列与裂解酶mtEC340的N端融合,构建了新的裂解酶LNT113,LNT113相较于改造前的裂解酶,抗菌活性显著增加[65]。
-
基于噬菌体裂解酶的模块化结构特征,利用蛋白质工程对裂解酶的结构域进行改造,可以提高裂解酶的活性、调节宿主范围。常见的方法包括定点突变、结构域的拆分与组合。常见的蛋白分子设计改造方法包括体外分子定向进化和理性设计。体外分子定向进化是指通过随机诱变或基因重组等方式实现基因多样化,在体外模拟自然进化过程构建突变体文库,并结合高通量筛选以获得性能优化的突变蛋白[66]。而理性设计则是基于对蛋白质催化机理、结构和功能的深入了解,指导精准突变,显著缩短了定向进化策略中繁琐的筛选迭代过程。其通常依赖X射线晶体学或冷冻电镜等实验手段解析蛋白质结构,或者借助AlphaFold等生物信息学进行结构预测[67]。在此基础上,利用分子动力学模拟,进一步预测可能影响酶活性、稳定性或底物特异性的突变位点,并通过定点突变构建功能优化的突变体[68]。通过定点突变改变蛋白质中特定氨基酸残基,可系统探究氨基酸组成对蛋白质功能的影响,进而获得裂解活性更高、裂解范围更广的工程化裂解酶。Chang等[69]为了提升裂解酶LysDZ25 的裂解活性和热稳定性,首先利用RoseTTAFold软件预测其三维结构,随后对LysDZ25的三个结构域进行分子动力学模拟,以实现对蛋白空间构象的动态采样。基于分子动力学模拟结果,设计小规模突变文库,最终获得了最优的单点、双点和三点突变体(S333V、S333V/N245R 和 S333V/N245R/D299L),它们具有更高的裂解活性和热稳定性,有效提升了LysDZ25在高温环境中的应用潜力。研究发现从氨基酸倾向性分析来看,极性残基,特别是谷氨酰胺(Gln)和丝氨酸(Ser),以及谷氨酸(Glu)和赖氨酸(Lys)等带电残基,有利于提高蛋白质的热稳定性[70]。高占比的α-螺旋和β-折叠结构以及极性氨基酸可能是导致裂解酶LysF23s2和H256D1具有热稳定性的重要原因[71]。因此基于氨基酸倾向性设计点突变,将其他氨基酸突变为极性残基或带电残基,是提升噬菌体裂解酶热稳定性的有效策略。
-
对于含有多个结构域的噬菌体裂解酶,可以将其结构域进行拆分,分别研究每个结构域的功能特性。基于各结构域的功能特点,重新组合结构域,设计出广谱或高度特异性的裂解酶。CBD能够特异性地与细菌细胞壁中的特定底物结合,这一特性决定了裂解酶的特异性以及底物谱[72];噬菌体裂解酶的CBD多种多样,而CD赋予裂解酶对特定底物的裂解能力,CD通过剪切肽聚糖中的化学键发挥作用,直接决定了裂解酶的裂解活性[73]。对既存在CBD又存在CD的噬菌体裂解酶进行拆分,在去除CBD后,仅含CD的裂解酶活性一般会低于全长裂解酶的活性[74];但有研究发现有的裂解酶的CBD通过分子内相互作用抑制了催化域的活性,当CBD与细胞壁结合后,相互作用与抑制作用才能被消除,在去除裂解酶的CBD后,催化域不再受到CBD的抑制作用,其活性显著增加[75]。N端的内肽酶催化结构域具有一定的底物特异性,即使去除了CBD,催化域仍然能够识别其底物,含有正电荷的CD可以依靠静电相互作用与细胞壁表面结合,维持裂解酶的裂解活性[76]。研究发现,某些含有多个CD和一个CBD的噬菌体裂解酶,在单独缺失CBD时裂解活性基本不变;而当CBD与中间的CD同时缺失后,裂解活性仍可维持,且裂解进程加快。例如,仅含CHAP结构域的截短蛋白LysLF1A,其裂解活性与全长LysLF1相当,且裂解动力学更快[77]。金黄色葡萄球菌噬菌体裂解酶大部分含有两个催化结构域和一个细胞壁结合结构域,由N末端CHAP酰胺水解酶/肽酶,中心的Ami-2酰胺酶和一个C端SH3b结构域组成,其裂解活性主要依赖于N末端的CHAP结构域。在浊度降低实验中发现,去除Ami-2酰胺酶结构域后,保留的CHAP和SH3b结构域的融合蛋白的裂解活性并不低于全长裂解酶LysK,而仅包含CHAP结构域的裂解酶展现出比全长裂解酶LysK更高的活性[78]。CD和CBD之间的连接序列在决定噬菌体裂解酶活性方面也起着关键作用,连接序列通过影响结构域间的灵活性,进而影响酶的整体功能,如果连接序列太短或太长,都会干扰这两个域之间的协同作用机制,截短ClyJ两个结构域的连接序列,连接序列最短的重组裂解酶ClyJ-3具有比原长裂解酶更高的杀菌活性[79]。
-
除了定点突变和拆分组合噬菌体裂解酶外,重组不同噬菌体裂解酶的结构域是开发新型抗菌药物的一种有效策略[80]。裂解酶的CBD与CD之间的功能相互独立,通过重组嵌合不同裂解酶功能域,可拓宽、缩小甚至重定向裂解酶的活性范围,获得更高效的裂解酶。噬菌体裂解酶很难有效地裂解多个属的细菌,裂解酶结构域的重组交换一般会导致嵌合噬菌体裂解酶裂解谱和裂解活性的变化,选择广谱裂解酶的CBD与其他裂解酶的CD结合后,广谱性特征会保留到嵌合型裂解酶中,而CD则影响着嵌合裂解酶的裂解活性[81]。有研究将仅对葡萄球菌高效的金黄色葡萄球菌噬菌体裂解酶 LysGH15 的半胱氨酸和组氨酸依赖性酰胺水解酶结构域(CHAP)与来自粪肠球菌噬菌体裂解酶PlyV12的CBD组合得到嵌合型裂解酶ClyQ,嵌合型裂解酶相比于原裂解酶LysGH15,具有更广泛的宿主范围和高效的裂解活性[82]。通过将不同噬菌体裂解酶的功能结构域组合在一起,嵌合型裂解酶MEndoB不仅继承了各个结构域的优点,还获得了更高的杀菌效率、更好的稳定性和更广泛的裂解谱[83]。也有研究致力于通过嵌合不同裂解酶的结构域,以获得具有更高特异性的裂解酶。具有高裂解活性以及高度特异性的裂解酶Ply187的CHAP结构域与裂解酶PlySs2的CBD,常被用作开发新型嵌合裂解酶。研究发现单独表达Ply187的CHAP结构域,其活性高于全长的金黄色葡萄球菌裂解酶Ply187,将该CHAP结构域与裂解酶LysSA97中对金黄色葡萄球菌具有特异性结合作用的非SH3b结合结构域进行嵌合,得到的嵌合裂解酶ClyC相较于原亲本酶具有更高的裂解活性及特异性[84]。此外,已有证据表明,具有多个 CBD组合的噬菌体裂解酶的耐药性风险更低[85]。裂解酶还可以与现有的抗生素发生协同效应,增强裂解酶的整体抗菌效果[15],嵌合型裂解酶MEndoB与传统抗生素联合使用表现出显著的协同效应,增强了整体抗菌效果[83]。嵌合裂解酶Cpl-711由不同裂解酶的结构域组成,显示优越的抗生物被膜和抗菌效果,在与抗生素联用后。其裂解活性进一步增强[86]。
-
细菌的抗生素耐药问题导致了多重耐药细菌的出现,开发新的抗菌药物迫在眉睫。迄今为止,裂解酶已被广泛应用于医药、食品、农业等多个领域,并取得了良好效果。噬菌体裂解酶作为一种潜在的新型抗菌药物,在对抗细菌方面展现出巨大的潜力。噬菌体裂解酶具有以下5个明显优势:1)快速的杀菌作用;2)高度的特异性;3)不易引起细菌产生耐药性;4)作为外源蛋白应用时,其蛋白质结构易于改造;5)能够有效地抑制或破坏生物被膜。然而,噬菌体裂解酶后续的研究与应用仍面临着如下4个挑战。1)特异性与广谱性的权衡:利用裂解酶的模块化结构,对裂解酶的结构域进行叠加或交换可以扩大或缩小裂解酶的裂解谱,但还是缺乏更深入的机制研究,设计既具特异性又能广泛作用于不同细菌的裂解酶是一个关键问题。2)表达毒性与生产成本问题:在使用细菌系统表达噬菌体裂解酶时常面临表达毒性问题—裂解酶可能提前裂解宿主细胞肽聚糖,导致细胞自溶影响蛋白产量。已经有研究选择酵母、哺乳动物细胞或植物细胞作为噬菌体裂解酶的表达宿主,从而避免可能的毒性问题。然而,这些真核表达系统的大规模生产和应用导致生产成本显著上升,限制了其规模化应用。3)储存稳定性:外源应用的噬菌体裂解酶为蛋白质,其活性高度依赖于特定的温度、pH和离子强度等环境条件。在储存或运输过程中,易因温度波动、反复冻融或极端pH导致蛋白聚集、变性。4)裂解酶的递送难度大且递送成本高:在临床上裂解酶目前的应用仅限于治疗皮肤和阴道感染,肠道中复杂的微生物群限制了裂解酶的应用。为克服递送难题,研究者利用聚合物颗粒、脂质体、无机纳米颗粒等递送系统及水凝胶等宏观递送系统,成功实现了对局部、全身、肺部及肌肉骨骼感染的精准递送。然而,目前这些技术尚未被用于将裂解酶输送到肠道内以调节微生物群落。但不可忽视的是,噬菌体裂解酶在抗菌领域展现出巨大潜力,为解决耐药菌问题带来了新的希望。
Research progress on bacteriophage lysin as novel antimicrobial drugs
-
摘要: 近年来,抗生素的滥用导致细菌耐药性问题日益严重,急需新的安全替代药物。噬菌体裂解酶是噬菌体侵染宿主细菌后产生的一种肽聚糖水解酶,能够水解细菌肽聚糖,导致细菌破裂、死亡并释放子代噬菌体。噬菌体裂解酶因其高度特异性、高效裂解活性及不易引起细菌产生耐药性等优势,在治疗细菌感染方面具有巨大的潜力。文章系统综述了噬菌体裂解酶作为新型抗菌药物的研究进展,重点围绕裂解酶的结构组成、作用机制及工程化改造策略,以期为设计高效、高质量的新型噬菌体裂解酶抗菌药物提供理论依据与技术参考。Abstract: In recent years, the overuse and misuse of antibiotics have led to the problem of bacterial drug resistance, and new safe alternative drugs are urgently needed. Bacteriophage lysin is a peptidoglycan hydrolase encoded by bacteriophages after infestation of host bacteria, which can hydrolyze bacterial peptidoglycan from the inside of bacteria, leading to bacterial rupture, death and release of progeny phage. Bacteriophage lysins have great potential in the treatment of bacterial infections due to their high specificity, efficient lysis, and the fact that they do not easily cause bacterial drug resistance. This article provides a systematic review of the research progress on bacteriophage lysins as novel antimicrobial drugs, focusing on the structural composition, mechanism of action, and engineering modification strategies of bacteriophage lysins, with the aim of providing theoretical basis and technical references for the design of efficient and high-quality novel antimicrobial drugs.
-
Key words:
- bacteriophage /
- bacteriophage lysin /
- novel antimicrobial agents /
- drug resistance /
- mechanism of action
-
[1] Qin Y Q, Ren X Y, Zhang Y K, et al. Distribution characteristics of antibiotic resistance genes and microbial diversity in the inshore aquaculture area of Wenchang, Hainan, China [J]. Science of the Total Environment, 2024, 914: 169695. https://doi.org/10.1016/j.scitotenv.2023.169695 doi: 10.1016/j.scitotenv.2023.169695 [2] 曹晏文, 刘丰铭, 乔明强. 噬菌体裂解酶抑菌功能与应用研究进展[J]. 农产品加工, 2024(12): 74−78. https://doi.org/10.16693/j.cnki.1671-9646(X).2024.12.017 doi: 10.16693/j.cnki.1671-9646(X).2024.12.017 [3] 闵德省. 葡萄球菌噬菌体裂解酶的研究进展[J]. 福建畜牧兽医, 2025, 47(2): 64−67. https://doi.org/10.3969/j.issn.1003-4331.2025.02.019 doi: 10.3969/j.issn.1003-4331.2025.02.019 [4] 张瑞凌, 冼盈, 张扣兴. 鲍曼不动杆菌感染与免疫研究进展[J]. 中国感染与化疗杂志, 2017, 17(2): 224−228. https://doi.org/10.16718/j.1009-7708.2017.02.022 doi: 10.16718/j.1009-7708.2017.02.022 [5] Fleming A. Classics in infectious diseases: on the antibacterial action of cultures of a penicillium, with special reference to their use in the isolation of B. influenzae by Alexander Fleming, reprinted from the British Journal of Experimental Pathology 10: 226−236, 1929 [J]. Reviews of Infectious Diseases, 1980, 2(1): 129−139. https://doi.org/10.1093/clinids/2.1.129 doi: 10.1093/clinids/2.1.129 [6] Christaki E, Marcou M, Tofarides A. Antimicrobial resistance in bacteria: mechanisms, evolution, and persistence [J]. Journal of Molecular Evolution, 2020, 88(1): 26−40. https://doi.org/10.1007/s00239-019-09914-3 doi: 10.1007/s00239-019-09914-3 [7] Lopatek M, Wieczorek K, Osek J. Antimicrobial resistance, virulence factors, and genetic profiles of Vibrio parahaemolyticus from seafood [J]. Applied and Environmental Microbiology, 2018, 84(16): e00537−18. https://doi.org/10.1128/AEM.00537-18 doi: 10.1128/AEM.00537-18 [8] Marshall B M, Levy S B. Food animals and antimicrobials: impacts on human health [J]. Clinical Microbiology Reviews, 2011, 24(4): 718−733. https://doi.org/10.1128/CMR.00002-11 doi: 10.1128/CMR.00002-11 [9] Preena P G, Swaminathan T R, Kumar V J R, et al. Antimicrobial resistance in aquaculture: a crisis for concern [J]. Biologia, 2020, 75(9): 1497−1517. https://doi.org/10.2478/s11756-020-00456-4 doi: 10.2478/s11756-020-00456-4 [10] 王昕, 韩语, 潘纪汶, 等. 临床分离粪肠球菌的毒力基因和耐药基因检测及其耐药性研究[J]. 热带生物学报, 2023, 14(2): 138−144. https://doi.org/10.15886/j.cnki.rdswxb.2023.02.001 doi: 10.15886/j.cnki.rdswxb.2023.02.001 [11] Fishbein S R S, Mahmud B, Dantas G. Antibiotic perturbations to the gut microbiome [J]. Nature Reviews Microbiology, 2023, 21(12): 772−788. https://doi.org/10.1038/s41579-023-00933-y doi: 10.1038/s41579-023-00933-y [12] Mäntynen S, Laanto E, Oksanen H M, et al. Black box of phage-bacterium interactions: exploring alternative phage infection strategies [J]. Open Biology, 2021, 11(9): 210188. https://doi.org/10.1098/rsob.210188 doi: 10.1098/rsob.210188 [13] Łusiak-Szelachowska M, Weber-Dąbrowska B, Górski A. Bacteriophages and lysins in biofilm control [J]. Virologica Sinica, 2020, 35(2): 125−133. https://doi.org/10.1007/s12250-019-00192-3 doi: 10.1007/s12250-019-00192-3 [14] Golban M, Charostad J, Kazemian H, et al. Phage-derived endolysins against resistant Staphylococcus spp: a review of features, antibacterial activities, and recent applications [J]. Infectious Diseases and Therapy, 2025, 14(1): 13−57. https://doi.org/10.1007/s40121-024-01069-z doi: 10.1007/s40121-024-01069-z [15] Rahman M U, Wang W X, Sun Q Q, et al. Endolysin, a promising solution against antimicrobial resistance [J]. Antibiotics, 2021, 10(11): 1277. https://doi.org/10.3390/antibiotics10111277 doi: 10.3390/antibiotics10111277 [16] Hampton H G, Watson B N J, Fineran P C. The arms race between bacteria and their phage foes [J]. Nature, 2020, 577(7790): 327−336. https://doi.org/10.1038/s41586-019-1894-8 doi: 10.1038/s41586-019-1894-8 [17] Pfeifer E, Bonnin R A, Rocha E P C. Phage-plasmids spread antibiotic resistance genes through infection and lysogenic conversion [J]. mBio, 2022, 13(5): e01851−22. https://doi.org/10.1128/mbio.01851-22 doi: 10.1128/mbio.01851-22 [18] Kieffer N, Hipólito A, Ortiz-Miravalles L, et al. Mobile integrons encode phage defense systems [J]. Science, 2025, 388(6747): eads0915. https://doi.org/10.1126/science.ads0915 doi: 10.1126/science.ads0915 [19] Singh A N, Singh A, Singh S K, et al. Klebsiella pneumoniae infections and phage therapy [J]. Indian Journal of Medical Microbiology, 2024, 52: 100736. https://doi.org/10.1016/j.ijmmb.2024.100736 doi: 10.1016/j.ijmmb.2024.100736 [20] Müller S, Wolf A J, Iliev I D, et al. Poorly cross-linked peptidoglycan in MRSA due to mecA induction activates the inflammasome and exacerbates immunopathology [J]. Cell Host & Microbe, 2015, 18(5): 604−612. https://doi.org/10.1016/j.chom.2015.10.011 doi: 10.1016/j.chom.2015.10.011 [21] Abdelrahman F, Gangakhedkar R, Nair G, et al. Pseudomonas phage ZCPS1 endolysin as a potential therapeutic agent [J]. Viruses, 2023, 15(2): 520. https://doi.org/10.3390/v15020520 doi: 10.3390/v15020520 [22] Vázquez R, García E, García P. Sequence-function relationships in phage-encoded bacterial cell wall lytic enzymes and their implications for phage-derived product design [J]. Journal of Virology, 2021, 95(14): e00321−21. [23] Schmelcher M, Donovan D M, Loessner M J. Bacteriophage endolysins as novel antimicrobials [J]. Future Microbiology, 2012, 7(10): 1147−1171. https://doi.org/10.2217/fmb.12.97 doi: 10.2217/fmb.12.97 [24] Dams D, Briers Y. Enzybiotics: enzyme-based antibacterials as therapeutics [J]. Advances in Experimental Medicine and Biology, 2019, 1148: 233−253. https://doi.org/10.1007/978-981-13-7709-9_11 doi: 10.1007/978-981-13-7709-9_11 [25] Qiao S, Luo Q S, Zhao Y, et al. Structural basis for lipopolysaccharide insertion in the bacterial outer membrane [J]. Nature, 2014, 511(7507): 108−111. https://doi.org/10.1038/nature13484 doi: 10.1038/nature13484 [26] Chang Y. Bacteriophage-derived endolysins applied as potent biocontrol agents to enhance food safety [J]. Microorganisms, 2020, 8(5): 724. https://doi.org/10.3390/microorganisms8050724 doi: 10.3390/microorganisms8050724 [27] 喻鑫婷. 金黄色葡萄球菌噬菌体裂解酶和穿孔素的克隆表达及抗菌活性研究[D]. 合肥: 安徽医科大学, 2022. https://doi.org/10.26921/d.cnki.ganyu.2022.000642 [28] Pritchard D G, Dong S L, Kirk M C, et al. LambdaSa1 and LambdaSa2 prophage lysins of Streptococcus agalactiae [J]. Applied and Environmental Microbiology, 2007, 73(22): 7150−7154. https://doi.org/10.1128/AEM.01783-07 doi: 10.1128/AEM.01783-07 [29] 滕铁山. 分枝杆菌噬菌体LiyA的分离、序列测定和基因组结构以及比较基因组分析及其gp29/gp30编码产物的裂解酶活性研究[D]. 重庆: 西南大学, 2012. [30] Wang Z J, Liu X, Shi Z X, et al. A novel lysin Ply1228 provides efficient protection against Streptococcus suis type 2 infection in a murine bacteremia model [J]. Veterinary Microbiology, 2022, 268: 109425. https://doi.org/10.1016/j.vetmic.2022.109425 doi: 10.1016/j.vetmic.2022.109425 [31] 袭恒豫. 绿色气球菌噬菌体裂解酶AVPL的生物学特性及其抗菌作用机制[D]. 长春: 吉林大学, 2024. https://doi.org/10.27162/d.cnki.gjlin.2024.000769 [32] Roach D R, Donovan D M. Antimicrobial bacteriophage-derived proteins and therapeutic applications [J]. Bacteriophage, 2015, 5(3): e1062590. https://doi.org/10.1080/21597081.2015.1062590 doi: 10.1080/21597081.2015.1062590 [33] 杨航, 余军平, 危宏平. 裂解酶治疗的研究进展与应用前景[J]. 微生物学通报, 2015, 42(1): 178−184. https://doi.org/10.13344/j.microbiol.china.140623 doi: 10.13344/j.microbiol.china.140623 [34] Pastagia M, Schuch R, Fischetti V A, et al. Lysins: the arrival of pathogen-directed anti-infectives [J]. Journal of Medical Microbiology, 2013, 62 (Pt 10): 1506−1516. https://doi.org/10.1099/jmm.0.061028-0 [35] Young R. Phage lysis: three steps, three choices, one outcome [J]. Journal of Microbiology, 2014, 52(3): 243−258. https://doi.org/10.1007/s12275-014-4087-z doi: 10.1007/s12275-014-4087-z [36] Garde S, Chodisetti P K, Reddy M. Peptidoglycan: structure, synthesis, and regulation [J]. EcoSal Plus, 2021, 9(2): eESP−0010-2020. https://doi.org/10.1128/ECOSALPLUS.ESP-0010-2020 doi: 10.1128/ECOSALPLUS.ESP-0010-2020 [37] Vollmer W, Blanot D, De Pedro M A. Peptidoglycan structure and architecture [J]. FEMS Microbiology Reviews, 2008, 32(2): 149−167. https://doi.org/10.1111/j.1574-6976.2007.00094.x doi: 10.1111/j.1574-6976.2007.00094.x [38] Loessner M J. Bacteriophage endolysins—current state of research and applications [J]. Current Opinion in Microbiology, 2005, 8(4): 480−487. https://doi.org/10.1016/j.mib.2005.06.002 doi: 10.1016/j.mib.2005.06.002 [39] Broendum S S, Buckle A M, Mcgowan S. Catalytic diversity and cell wall binding repeats in the phage-encoded endolysins [J]. Molecular Microbiology, 2018, 110(6): 879−896. https://doi.org/10.1111/mmi.14134 doi: 10.1111/mmi.14134 [40] Abdelrahman F, Easwaran M, Daramola O I, et al. Phage-encoded endolysins [J]. Antibiotics, 2021, 10(2): 124. https://doi.org/10.3390/antibiotics10020124 doi: 10.3390/antibiotics10020124 [41] Cheng X, Zhang X, Pflugrath J W, et al. The structure of bacteriophage T7 lysozyme, a zinc amidase and an inhibitor of T7 RNA polymerase [J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(9): 4034−4038. https://doi.org/10.1073/pnas.91.9.4034 doi: 10.1073/pnas.91.9.4034 [42] Yang S P, Meng X P, Zhen Y Q, et al. Strategies and mechanisms targeting Enterococcus faecalis biofilms associated with endodontic infections: a comprehensive review [J]. Frontiers in Cellular and Infection Microbiology, 2024, 14: 1433313. https://doi.org/10.3389/fcimb.2024.1433313 doi: 10.3389/fcimb.2024.1433313 [43] 刘宝玲, 陈天宝, 柳旭辉, 等. 新型抗生物膜药物研究进展[J]. 动物医学进展, 2023, 44(11): 84−88. https://doi.org/10.16437/j.cnki.1007-5038.2023.11.015 doi: 10.16437/j.cnki.1007-5038.2023.11.015 [44] Wu J A, Kusuma C, Mond J J, et al. Lysostaphin disrupts Staphylococcus aureus and Staphylococcus epidermidis biofilms on artificial surfaces [J]. Antimicrobial Agents and Chemotherapy, 2003, 47(11): 3407−3414. https://doi.org/10.1128/AAC.47.11.3407-3414.2003 doi: 10.1128/AAC.47.11.3407-3414.2003 [45] Gutiérrez D, Ruas-Madiedo P, Martínez B, et al. Effective removal of staphylococcal biofilms by the endolysin LysH5 [J]. PLoS One, 2014, 9(9): e107307. https://doi.org/10.1371/journal.pone.0107307 doi: 10.1371/journal.pone.0107307 [46] Lendel A M, Antonova N P, Grigoriev I V, et al. Biofilm-disrupting effects of phage endolysins LysAm24, LysAp22, LysECD7, and LysSi3: breakdown the matrix [J]. World Journal of Microbiology and Biotechnology, 2024, 40(6): 186. https://doi.org/10.1007/s11274-024-03999-9 doi: 10.1007/s11274-024-03999-9 [47] Gutiérrez D, Briers Y. Lysins breaking down the walls of Gram-negative bacteria, no longer a no-go [J]. Current Opinion in Biotechnology, 2021, 68: 15−22. https://doi.org/10.1016/j.copbio.2020.08.014 doi: 10.1016/j.copbio.2020.08.014 [48] 刘菲, 黄柯, 郑积敏. 革兰氏阴性菌细胞表面多糖的应用潜力[J]. 化学教育(中英文), 2024, 45(6): 1−10. https://doi.org/10.13884/j.1003-3807hxjy.2023020111 doi: 10.13884/j.1003-3807hxjy.2023020111 [49] 周巾煜, 李倩倩, 黄纯翠, 等. 细菌脂多糖及其寡糖链结构分析技术研究进展[J]. 生物化学与生物物理进展, 2017, 44(1): 49−57. https://doi.org/10.16476/j.pibb.2016.0306 doi: 10.16476/j.pibb.2016.0306 [50] 莫婷, 刘马峰, 程安春. 革兰氏阴性菌脂多糖运输系统的构成及作用机制[J]. 微生物学报, 2018, 58(9): 1521−1530. https://doi.org/10.13343/j.cnki.wsxb.20170517 doi: 10.13343/j.cnki.wsxb.20170517 [51] Briers Y, Lavigne R. Breaking barriers: expansion of the use of endolysins as novel antibacterials against Gram-negative bacteria [J]. Future Microbiology, 2015, 10(3): 377−390. https://doi.org/10.2217/fmb.15.8 doi: 10.2217/fmb.15.8 [52] Ruiz N, Kahne D, Silhavy T J. Transport of lipopolysaccharide across the cell envelope: the long road of discovery [J]. Nature Reviews Microbiology, 2009, 7(9): 677−683. https://doi.org/10.1038/nrmicro2184 doi: 10.1038/nrmicro2184 [53] Blasco L, Ambroa A, Trastoy R, et al. In vitro and in vivo efficacy of combinations of colistin and different endolysins against clinical strains of multi-drug resistant pathogens [J]. Scientific Reports, 2020, 10(1): 7163. https://doi.org/10.1038/s41598-020-64145-7 doi: 10.1038/s41598-020-64145-7 [54] Kim J, Wang J, Ahn J. Combined antimicrobial effect of phage-derived endolysin and depolymerase against biofilm-forming Salmonella typhimurium [J]. Biofouling, 2023, 39(7): 763−774. https://doi.org/10.1080/08927014.2023.2265817 doi: 10.1080/08927014.2023.2265817 [55] Lood R, Winer B Y, Pelzek A J, et al. Novel phage lysin capable of killing the multidrug-resistant Gram-negative bacterium Acinetobacter baumannii in a mouse bacteremia model [J]. Antimicrobial Agents and Chemotherapy, 2015, 59(4): 1983−1991. https://doi.org/10.1128/AAC.04641-14 doi: 10.1128/AAC.04641-14 [56] Larpin Y, Oechslin F, Moreillon P, et al. In vitro characterization of PlyE146, a novel phage lysin that targets Gram-negative bacteria [J]. PLoS One, 2018, 13(2): e0192507. https://doi.org/10.1371/journal.pone.0192507 doi: 10.1371/journal.pone.0192507 [57] Low L Y, Yang C, Perego M, et al. Role of net charge on catalytic domain and influence of cell wall binding domain on bactericidal activity, specificity, and host range of phage lysins [J]. Journal of Biological Chemistry, 2011, 286(39): 34391−34403. https://doi.org/10.1074/jbc.M111.244160 doi: 10.1074/jbc.M111.244160 [58] Lai M J, Lin N T, Hu A R, et al. Antibacterial activity of Acinetobacter baumannii phage ϕAB2 endolysin(LysAB2) against both Gram-positive and Gram-negative bacteria [J]. Applied Microbiology and Biotechnology, 2011, 90(2): 529−539. https://doi.org/10.1007/s00253-011-3104-y doi: 10.1007/s00253-011-3104-y [59] Breijyeh Z, Jubeh B, Karaman R. Resistance of Gram-negative bacteria to current antibacterial agents and approaches to resolve it [J]. Molecules, 2020, 25(6): 1340. https://doi.org/10.3390/molecules25061340 doi: 10.3390/molecules25061340 [60] Briers Y, Cornelissen A, Aertsen A, et al. Analysis of outer membrane permeability of Pseudomonas aeruginosa and bactericidal activity of endolysins KZ144 and EL188 under high hydrostatic pressure [J]. FEMS Microbiology Letters, 2008, 280(1): 113−119. https://doi.org/10.1111/j.1574-6968.2007.01051.x doi: 10.1111/j.1574-6968.2007.01051.x [61] Sisson H M, Fagerlund R D, Jackson S A, et al. Antibacterial synergy between a phage endolysin and citric acid against the Gram-negative kiwifruit pathogen Pseudomonas syringae pv. actinidiae [J]. Applied and Environmental Microbiology, 2024, 90(3): e01846−23. https://doi.org/10.1128/aem.01846-23 doi: 10.1128/aem.01846-23 [62] Ning H Q, Cong Y, Lin H, et al. Development of cationic peptide chimeric lysins based on phage lysin Lysqdvp001 and their antibacterial effects against Vibrio parahaemolyticus: a preliminary study [J]. International Journal of Food Microbiology, 2021, 358: 109396. https://doi.org/10.1016/j.ijfoodmicro.2021.109396 doi: 10.1016/j.ijfoodmicro.2021.109396 [63] Ma Q, Guo Z M, Gao C C, et al. Enhancement of the direct antimicrobial activity of Lysep3 against Escherichia coli by inserting cationic peptides into its C terminus [J]. Antonie van Leeuwenhoek, 2017, 110(3): 347−355. https://doi.org/10.1007/s10482-016-0806-2 doi: 10.1007/s10482-016-0806-2 [64] Mancoš M, Šramková Z, Peterková D, et al. Functional expression and purification of tailor-made chimeric endolysin with the broad antibacterial spectrum [J]. Biologia, 2020, 75(11): 2031−2043. https://doi.org/10.2478/s11756-020-00508-9 doi: 10.2478/s11756-020-00508-9 [65] Hong H W, Kim Y D, Jang J, et al. Combination effect of engineered endolysin EC340 with antibiotics [J]. Frontiers in Microbiology, 2022, 13: 821936. https://doi.org/10.3389/fmicb.2022.821936 doi: 10.3389/fmicb.2022.821936 [66] Wang Y J, Xue P, Cao M F, et al. Directed evolution: methodologies and applications [J]. Chemical Reviews, 2021, 121(20): 12384−12444. https://doi.org/10.1021/acs.chemrev.1c00260 doi: 10.1021/acs.chemrev.1c00260 [67] 陈涛, 赖锦涛, 胡美林, 等. 蛋白质优化设计与从头合成引领的疫苗研发革命[J/OL]. 合成生物学, 2025[2025-09-24]. https://link.cnki.net/urlid/10.1687.Q.20250820.1317.002. [68] 张颜伊, 汪路杰, 程峰, 等. 酶定向进化技术在单细胞蛋白生产中的研究进展[J/OL]. 科学通报, 2025[2025-09-24]. https://link.cnki.net/urlid/11.1784.N.20250827.1008.004. [69] Chang Y, Li Q B, Zhang S H, et al. Identification and molecular modification of Staphylococcus aureus bacteriophage lysin LysDZ25 [J]. ACS Infectious Diseases, 2023, 9(3): 497−506. https://doi.org/10.1021/acsinfecdis.2c00493 doi: 10.1021/acsinfecdis.2c00493 [70] Kumar S, Duggineni V K, Singhania V, et al. Unravelling and quantifying the biophysical– biochemical descriptors governing protein thermostability by machine learning [J]. Advanced Theory and Simulations, 2023, 6(3): 2200703. https://doi.org/10.1002/adts.202200703 doi: 10.1002/adts.202200703 [71] 石甜, 史聪聪, 衡冰冰, 等. 副溶血弧菌噬菌体裂解酶LysF23s2和LysH256D1的表达及活性分析[J]. 武汉轻工大学学报, 2024, 43(5): 10−17. [72] Loessner M J, Kramer K, Ebel F, et al. C-terminal domains of Listeria monocytogenes bacteriophage murein hydrolases determine specific recognition and high-affinity binding to bacterial cell wall carbohydrates [J]. Molecular Microbiology, 2002, 44(2): 335−349. https://doi.org/10.1046/j.1365-2958.2002.02889.x doi: 10.1046/j.1365-2958.2002.02889.x [73] 张时雨. 裂解酶Ply0643及其嵌合体的体内外抑菌活性以及菲啶酮类化合物抑菌活性的分析[D]. 南京: 南京农业大学, 2021. doi: 10.27244/d.cnki.gnjnu.2021.001210 [74] Sass P, Bierbaum G. Lytic activity of recombinant bacteriophage φ11 and φ12 endolysins on whole cells and biofilms of Staphylococcus aureus [J]. Applied and Environmental Microbiology, 2007, 73(1): 347−352. https://doi.org/10.1128/AEM.01616-06 doi: 10.1128/AEM.01616-06 [75] Low L Y, Yang C, Perego M, et al. Structure and lytic activity of a Bacillus anthracis prophage endolysin [J]. Journal of Biological Chemistry, 2005, 280(42): 35433−35439. https://doi.org/10.1074/jbc.M502723200 doi: 10.1074/jbc.M502723200 [76] Mayer M J, Garefalaki V, Spoerl R, et al. Structure-based modification of a Clostridium difficile-targeting endolysin affects activity and host range [J]. Journal of Bacteriology, 2011, 193(19): 5477−5486. https://doi.org/10.1128/JB.00439-11 doi: 10.1128/JB.00439-11 [77] 黄春正. 裂解酶LysLF1的生物学特性及其抗链球菌的效果评价[D]. 长春: 吉林大学, 2023. https://doi.org/10.27162/d.cnki.gjlin.2023.004564 [78] Horgan M, O'flynn G, Garry J, et al. Phage lysin LysK can be truncated to its CHAP domain and retain lytic activity against live antibiotic-resistant staphylococci [J]. Applied and Environmental Microbiology, 2009, 75(3): 872−874. https://doi.org/10.1128/AEM.01831-08 doi: 10.1128/AEM.01831-08 [79] Yang H, Luo D H, Etobayeva I, et al. Linker editing of pneumococcal lysin ClyJ conveys improved bactericidal activity [J]. Antimicrobial Agents and Chemotherapy, 2020, 64(2): e01610−e01619. https://doi.org/10.1128/AAC.01610-19 doi: 10.1128/AAC.01610-19 [80] Yang H, Yu J P, Wei H P. Engineered bacteriophage lysins as novel anti-infectives [J]. Frontiers in Microbiology, 2014, 5: 542. https://doi.org/10.3389/fmicb.2014.00542 doi: 10.3389/fmicb.2014.00542 [81] Dong Q H, Wang J, Yang H, et al. Construction of a chimeric lysin Ply187N-V12C with extended lytic activity against staphylococci and streptococci [J]. Microbial Biotechnology, 2015, 8(2): 210−220. https://doi.org/10.1111/1751-7915.12166 doi: 10.1111/1751-7915.12166 [82] Duan X C, Li X X, Li X M, et al. Exploiting broad-spectrum chimeric lysin to cooperate with mupirocin against Staphylococcus aureus-induced skin infections and delay the development of mupirocin resistance [J]. Microbiology Spectrum, 2023, 11(3): e05050−22. https://doi.org/10.1128/spectrum.05050-22 doi: 10.1128/spectrum.05050-22 [83] Roehrig C, Huemer M, Lorgé D, et al. MEndoB, a chimeric lysin featuring a novel domain architecture and superior activity for the treatment of staphylococcal infections [J]. mBio, 2024, 15(2): e02540−23. https://doi.org/10.1128/mbio.02540-23 doi: 10.1128/mbio.02540-23 [84] Li X H, Wang S J, Nyaruaba R, et al. A highly active chimeric lysin with a calcium-enhanced bactericidal activity against Staphylococcus aureus in vitro and in vivo [J]. Antibiotics, 2021, 10(4): 461. https://doi.org/10.3390/antibiotics10040461 doi: 10.3390/antibiotics10040461 [85] Becker S C, Roach D R, Chauhan V S, et al. Triple-acting lytic enzyme treatment of drug-resistant and intracellular Staphylococcus aureus [J]. Scientific Reports, 2016, 6: 25063. https://doi.org/10.1038/srep25063 doi: 10.1038/srep25063 [86] Letrado P, Corsini B, Díez-Martínez R, et al. Bactericidal synergism between antibiotics and phage endolysin Cpl-711 to kill multidrug-resistant pneumococcus [J]. Future Microbiology, 2018, 13(11): 1215−1223. https://doi.org/10.2217/fmb-2018-0077 doi: 10.2217/fmb-2018-0077 -




 下载:
下载: