-
东南亚低海拔地区的热带雨林主要由龙脑香科(Dipterocarpaceae)树种构成,它们占低地雨林森林底面积的20%~50%,而冠层中龙脑香科的比例高达50%及以上[1-2]。海南岛地处亚洲热带北缘,水热条件不如东南亚典型热带地区优越,天然分布的龙脑香科仅有2属3种,包括坡垒属的坡垒(Hopea hainanensis )与铁凌(Hopea reticulata ),青梅属的青梅(Vatica mangachapoi )[3]。坡垒是中国热带雨林的标志性物种之一[4],在低地雨林建成与生态系统服务功能上发挥重要作用。坡垒也是珍贵用材树种,其木材纹理交错,坚硬致密,极耐腐蚀与浸渍,是有名的高强度用材。由于过度采伐与林地开垦,海南岛低地雨林中残存的天然坡垒数量极少,被《中国植物红皮书》列为濒危物种[5],也是国家一级保护植物[6],世界自然保护联盟(IUCN)将坡垒评定为“濒危”(EN)[7],因此,亟需开展坡垒的保护遗传学、交配系统与种群生存力等研究,以实现种群恢复,拯救这一珍贵树种。
坡垒的研究目前主要集中在种群与群落生态、种子特性、幼苗生长,以及遗传多样性等方面。1983年胡玉佳[8]报导坡垒为海南岛低地雨林的冠层树种,其种群为林区的优势种群,在林内的重要值达156.5。Lan等[9]指出坡垒种子不耐脱水,对低温敏感,通过研究发现开花后173 d或183 d是采集坡垒种子贮藏的最佳阶段,种子的最佳保存温度为15~20 ℃。坡垒幼苗竞争力较弱,生物与非生物因素的干扰,因此从幼苗过渡到幼树阶段存在严重的增补限制[10]。基于微卫星和全基因组单核苷酸多态性,研究发现海南岛坡垒种群的遗传多样性显著低于同属的非濒危种,甚至低于部分已测序的濒危林木,如银杏(Ginkgo biloba)、银杉(Cathaya argyrophylla)、漾濞槭(Acer yangbiense)、朱红大杜鹃(Rhododendron griersonianum)等,剧烈的种群瓶颈被认为是坡垒遗传变异损失的主要原因[11-12]。Wang等[13]通过高通量测序组装了坡垒染色体级别的基因组序列,揭示坡垒为同源四倍体,所携带的纯合有害突变较少。坡垒较低的遗传负荷可能与其为同源四倍体有关。
繁殖方式对于种群遗传变异的数量和分布有重要影响,是濒危植物保护研究的重要内容[14]。有性生殖是高等植物的主要繁殖方式,无性生殖可以看作是植物不依靠传粉媒介的一种适应机制[15]。无融合生殖是指不经过雌雄性细胞融合,后代由母本直接产生的繁殖方式,被认为是无性生殖的一种[16],研究表明植物的无融合生殖与多倍化密切相关[17],多倍体减数分裂时因多条染色体(
$ \ge $ 3条)联会,同源染色体极易发生不均衡分离,导致配子不育,因此,多倍体大多只能通过无融合生殖产生种子;而二倍体可以正常减数分裂,能够产生有性生殖的种子。多倍化是植物进行无融合生殖的诱因。无融合生殖可能会促进新基因型的出现与固定,有益于种群增长与扩散[18-19],但无融合生殖不能从种群基因库中清除有害突变,并不利于种群的长期生存[20]。对于濒危植物,探究其繁殖方式不但有助于分析濒危机制,也有利于制定合理的保护策略[21]。但目前还未有坡垒交配系统的研究报道。
龙脑香科二倍体物种也普遍采取有性生殖,而多倍体往往绕过减数分裂,通过无融合生殖的方式产生种子[1]。如娑罗双属的三倍体Shorea resinosa通过无融合生殖产生有活力的种子[22],而同源四倍体S. ovalis spp. sericea也有很大比例的种子是无融合生殖产生的[23],一些坡垒属的多倍体,如Hopea latifolia(3X),Hopea odorata(3X)和Hopea subalata(3X)等都被认为存在无融合生殖现象[1]。海南岛的坡垒为同源四倍体,是与同属的多倍体一样行无融合生殖,还是有性生殖,或为兼性无融合生殖?确定坡垒的繁殖方式将加深对其遗传结构,多样性与进化历史的理解,也有助于评估其进化潜力和制定种群保护及恢复方案。
分子标记技术能够提供准确且丰富的基因型变异信息,且检测结果不受环境和材料部位等的影响[24]。微卫星(Microsatellites),亦称简单重复序列(simple sequence repeats, SSR),是以1~6个碱基为单位的串联重复DNA序列,广泛分布于核基因组和叶绿体基因组[25]。微卫星标记是一种共显性标记,其应用具有样品需求量小,多态性高,易于检测等优点[26],是保护遗传学理想的分子标记。单核苷酸多态性(single nucleotide polymorphisms,SNP)是个体间单个核苷酸差异(因单碱基转换、颠换、插入和缺失而产生)[27],基因组中有数量丰富的SNP变异。随着高通量测序技术的发展和成熟,SNP标记被广泛用于种群遗传学和保护生物学研究[28-30]。
本研究首先检测了霸王岭和尖峰岭10株坡垒母树及其子代的微卫星基因型,然后从10个家系中随机选取4个(BW4, BW7, BW8,JF),采用简化基因组测序获得全基因组水平的SNP变异。通过综合分析母树和子代的微卫星与SNP基因型,探究坡垒的繁殖方式,为坡垒种群进化历史与遗传保育提供重要见解。
-
研究区位于海南热带雨林国家公园的霸王岭片区( 18°48′ - 19°12′N,108°55′ - 109°17′E)和尖峰岭片区(18°38′—18°48′N,108°47′—109°02′E )(表1)。本研究选取霸王岭坡垒分布区内开花结果的9棵成年树为母本,挑选无病害且成熟的叶片,硅胶干燥保存备用。同时每棵母树采集大约25枚翅果,带回实验基地萌发并培育种苗。在尖峰岭片区,选择方圆500 m范围内仅有1棵成年且开花的坡垒个体作为研究对象,采用相同的方法采集叶片和果实。
表 1 10个坡垒家系的编号、子代数量、采集地点与海拔
Table 1. Code, seedlings number, district and altitude of ten family lines of Hopea hainanensis
地区
District家系
Family line子代数量 SSR(SNP)
Number of seedlings
genotyped by SSR(SNP)经度
Longitude纬度
Latitude海拔/m
Altitude/m霸王岭
Bawang MountainBW1 20 109.1331 °E19.0985 °N461 BW2 20 109.1333 °E19.0985 °N458 BW3 20 109.1335 °E19.0984 °N465 BW4 20(9) 109.1159 °E19.0914 °N338 BW5 0 109.1449 °E19.0785 °N785 BW6 2 109.3017 °E19.0789 °N626 BW7 20(19) 109.1192 °E19.0782 °N741 BW8 20(20) 109.1183 °E19.0798 °N687 BW9 20 109.1178 °E19.0819 °N748 尖峰岭
Jianfeng MountainJF 21(20) 109.7529 °E18.7349 °N688 -
将坡垒翅果带回实验室,去掉果翅,用0.1%的高锰酸钾溶液侵泡20分钟进行表面消毒处理,然后置于海南大学农科实验基地的沙床中萌发,待幼苗长出3~4片真叶,采集叶片提取子代基因组DNA。用于微卫星基因型检测实验的10个坡垒家系的子代个数(表1)。
采用改良的CTAB法提取母树与子代的基因组DNA,使用Wang等[31]开发的12对微卫星引物进行微卫星基因分型。PCR 扩增在 Eppendorf的热循环仪中进行。总反应体积20 μL,包含2 μL gDNA,0.2 μL正向荧光引物,0.2 μL反向引物,10 μL 2xTaq PCR MasterMix,余下体积用ddH2O补齐。循环程序如下:94 ℃预变性6 min,然后32个循环(94 ℃变性15 s,59~63 ℃退火30 s,72 ℃延伸60 s),最后72 ℃延伸7 min。使用ABI3730XL 分析仪(Applied Biosystems)进行片段分析, SSR等位基因的长度用GeneMarker 2.2确定,然后人工校正基因分型结果。
为验证微卫星基因型结果的可靠性,本研究团队从10个家系中随机选取4个(BW4, BW7, BW8, JF),通过简化基因组测序获得母树及子代全基因组水平的SNP标记。基因组DNA由限制性内切酶EcoRI和MseI消化,添加测序接头,电泳后选择400~500 bp的片段,经PCR扩增制备简化基因组测序文库[32] ,文库由广州捷锐生物科技公司的Illumina HiSeq测序平台进行高通量测序。
-
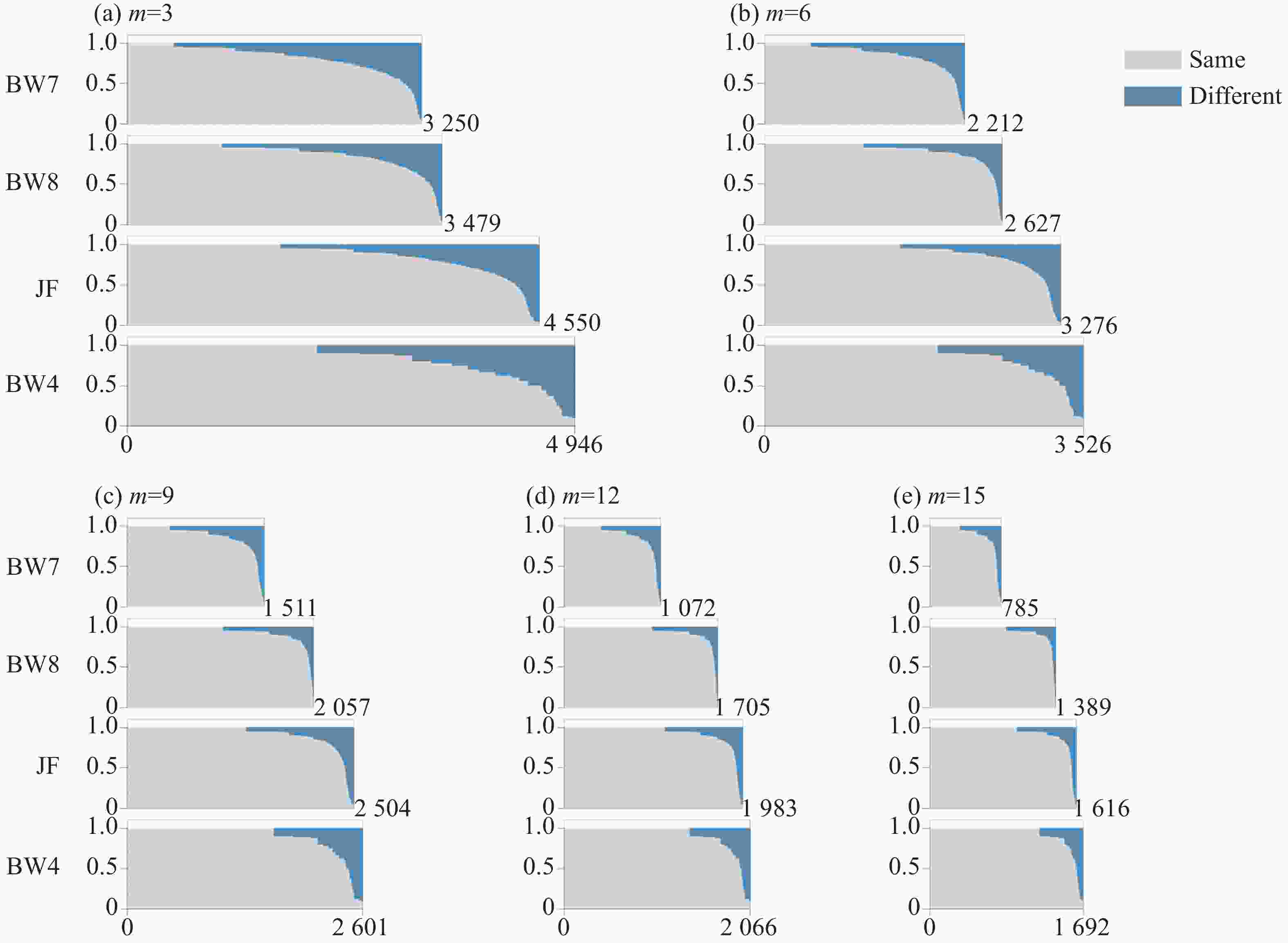
使用Stacks软件包的process_radtags.pl程序对原始下机数据进行拆分[33] ,过滤低质量碱基。运行denovo_map.pl程序,不依赖参考基因组组装酶切片段。改变个体两个等位基因之间允许错配的数量(M)和种群中任意两个等位基因之间允许错配的数量(n),但保持二者相等,以确定适宜数据的M值,作为组装位点的最佳参数值。使用Stacks软件中的populations程序判读SNP,要求位点缺失数据的比例小于20%,其他过滤条件使用默认值。参数m控制组装一个位点所需的reads数,为了检验reads数对组装和SNP判读结果的影响,本研究团队使用了5个逐步增大的m值(m=3, 6, 9, 12, 15),分析SNP判读结果的变化。基于母本杂合位点开展后续数据分析,确定每个子代与母本SNP位点基因型相同的位点个数。对于同一个家系的母本与子代,分析SNP位点基因型的情况,统计每个SNP位点与母本基因型相同和单个等位基因差异的子代比例,使用python程序绘图可视化统计结果。
-
12对微卫星引物对10个坡垒母树基因分型的结果见表2。霸王岭9棵母树的微卫星基因型完全相同,并且母树与其子代的基因型也完全相同。尖峰岭母树与霸王岭母树在微卫星基因型上存在差异,但尖峰岭母树与其子代的基因型完全相同。母树与子代基因型完全相同提示坡垒可能行专性无融合生殖,也可能是因为微卫星标记的分辨率不够。为此,本研究团队利用简化基因组测序分析基因组水平上的SNP变异模式,进一步探究坡垒的繁殖模式。
表 2 10个坡垒家系母树SSR基因型
Table 2. SSR genotypes of mother trees in ten family lines of Hopea hainanensis
HHA01 HHA03 HHA04 HHA11 HHA14 HHA24 HHA27 HHA32 HHA41 HHA49 HHA50 HHA62 BW1 103,108 / / / / / / / 126 / / 86,92 BW2 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW3 / / / 144,150 / 141,145 / / 126 / / / BW4 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW5 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW6 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW7 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW8 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW9 103,108 155,159 126,132 144,150 / 141,145 129,135 224,228 126 159 159,163 86,92 JF 103,108 159,163 126,134 144,150 116 143 133 224,230 126,129 159 157,167 84 注:“/”表示缺失数据。 Note:"/" indicates missing data. SNP位点变异模式分析在家系内进行。与微卫星基因型的结果不同,子代SNP位点的基因型与母本并不完全相同(表3)。当m=3时,与母本SNP基因型相同的位点在4个家系中均占到81%以上。考虑到坡垒为同源四倍体,有高比例的重复序列,基因组复杂;且SNP判读结果是通过从头组装,在无参考基因组的条件下获得的,因此不排除部分SNP位点可能是组装错误导致的非真实SNP变异。参数m控制组装一个酶切位点所需的reads数,提高m值可以提高组装位点的可靠性[34]。分析显示,SNP变异位点的数量随参数m值增大而减少,这是因为m值增加导致组装的酶切片段减少,但与母本SNP基因型相同位点的比例逐渐提高。从m取值3到15,与母本具有相同SNP的比例提高了大约7至10个百分点(表3)。这一分析结果表明SNP变异位点中确实存在因组装错误而导致的虚假SNP,而提高参数m的值可以在一定程度上避免虚假SNP的干扰。我们进一步按照与母本基因型相同与单个等位差异统计每个家系在每个SNP变异位点上的组成情况(图1)。伴随m值的增大,与母本完全相同的SNP位点的数量略微减少;但与母本有一个等位差异的SNP位点,其差异比例的分布模式发生明显改变,从连续平缓的增加变为快速陡峭的增长。表明相当比例的SNP位点,其子代与母本SNP基因型的差异很可能是由于子代SNP基因型因组装错误而导致的判读错误。当m值逐步增加,一部分较少个数reads支持的酶切片段不再被组装,因此虚假SNP的数量减少,与母本差异SNP的位点个数减少。同时我们发现,即便m取值为15,仍然存在部分SNP位点其子代与母本存在很高比例的差异(图1),这些SNP位点的产生还有待进一步研究。
表 3 4个坡垒家系母树与子代SNP基因型比较分析结果
Table 3. Comparative analysis of SNP genotype between mother trees and their seedlings in four family lines of Hopea hainanensis
家系
Familym=3 m=6 m=9 m=12 m=15 子代与母本
具有相同
SNP基因型
的比例
Prop. SNPs
the same as
its motherarent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因
型的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因
型的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因型
的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因
型的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPsBW7 0.812 ± 0.057 2883.2 0.854 ± 0.051 1943.5 0.873 ± 0.048 1311.0 0.881 ± 0.045 920.3 0.883 ± 0.042 671.9 BW8 0.869 ± 0.062 3182.5 0.910 ± 0.052 2401.2 0.930 ± 0.048 1855.6 0.939 ± 0.042 1518.2 0.942 ± 0.042 1223.3 JF 0.857 ± 0.034 4189.7 0.888 ± 0.029 3026.5 0.911 ± 0.026 2310.0 0.926 ± 0.025 1822.5 0.935 ± 0.024 1478.7 BW4 0.814 ± 0.044 4518.1 0.859 ± 0.032 3225.6 0.883 ± 0.028 2377.7 0.902 ± 0.028 1880.8 0.917 ± 0.026 1530.4 -
无融合生殖现象在植物中很常见,目前已在被子植物的70余科300多个属中都发现了无融合生殖现象[35]。鉴定是无融合生殖研究的前提与关键环节,形态观察、胚胎观察、去雄套袋实验和分子标记检测等是无融合生殖常用的鉴定方法[36]。早期龙脑香科的无融合生殖主要通过细胞和胚胎学观察进行研究,发现娑罗双属(Shorea),坡垒属(Hopea)和龙脑香属(Dipterocarpus)的多倍体物种存在无融合生殖现象[37–38, 38]。近年来分子标记技术被越来越多用于无融合生殖检测。如Pangsuban等[39]利用RAPD标记发现Garcinia atroviridis种群中58%的子代与母本具有完全相同的条带组成,认为它们是无融合生殖产生的。通过SSR、AFLP和叶绿体片段测序分析,Majesky等[40]发现Taraxacum scanicum的无融合繁殖种群遗传上高度同质,不同种群内检测到少量的特有基因型,认为是由体细胞突变导致的基因型差异。Yu等[41]利用SNP标记鉴定出了Iris domestica通过无融合生殖产生的后代及与I. dichotoma的杂交后代。分子标记普遍具有灵敏,稳定的特性,可为无融合生殖鉴定提供有力证据。
本研究通过SSR标记分析显示坡垒的子代与其母树的SSR基因型完全相同,由此推测海南岛的坡垒可能行无融合生殖。基于简化基因组测序获得的覆盖全基因组的SNP变异进一步支持上述推断,显示霸王岭和尖峰岭4个坡垒家系的子代具有很高比例的与母树SNP基因型相同的变异位点。如果通过母树的自交或异交来解释这些变异位点,其发生概率是以0.5为底数,SNP位点个数为指数的幂,数值几乎等于零,因此坡垒母树与子代的SNP变异模式不可能是有性生殖产生的。虽然部分SNP位点观察到一定比例的子代其SNP基因型与母树存在差异,通过提高组装酶切位点的reads数可降低差异SNP位点的比例,显示这部分差异SNP位点很可能是酶切片段组装偏差所致(图1)。由于坡垒为同源四倍体,并且酶切片段是在无参考基因型的情况下组装的,因此酶切片段组装结果中可能存在同源性偏误,进而导致虚假的差异SNP位点。
兼性无融合生殖是指一物种同时进行有性生殖和无融合生殖[42],因此无融合生殖产生的子代将与母本基因型完全相同,而有性生殖的子代其基因型与母本可能存在差异。SSR标记的检测结果显示所有子代SSR基因型均与其母本完全相同,支持坡垒为专性无融合生殖。SNP标记分析结果显示子代与母本的SNP变异在大多数位点上保持一致,但子代与母本SNP基因型存在少量差异,这一差异很可能来自体细胞突变[43],也可能是无参组装时的偏差所致,后续还应当通过母树与种子基因组重测序分析坡垒是否进行兼性无融合生殖。
坡垒遗传变异研究结果也为其无融合生殖提供了间接证据。Wang等[11]利用SSR评估了海南岛坡垒种群的遗传多样性,发现其显著低于其他非濒危的坡垒属树种。Tang等[12]通过简化基因组测序发现海南岛坡垒的核苷酸多样性水平很低,比已经报道的其他极小种群和濒危植物的多样性水平还低。坡垒低水平的种群遗传变异可能与其无融合生殖的繁殖方式有关。
SSR标记结果还显示尖峰岭母树与霸王岭母树基因型存在差异,刘超男[44]、Wang等[11]以及Tang等[12]基于遗传聚类分析,分别发现海南岛坡垒种群有2个或5个遗传类群,表明坡垒种群维持了一定水平的遗传变异。坡垒个体间的遗传变异可能是体细胞突变产生的,多次起源也是产生遗传多样性的可能原因。要准确分析坡垒SNP变异的进化来源,需要获得高质量的坡垒参考基因组,通过基因组重测序进一步探究坡垒遗传变异产生的原因。
当植物无法通过减数分裂产生可存活的种子,或植物因气候、空间隔离、传粉者限制等因素不能进行有性生殖时,无融合生殖可起到繁殖保障的作用[45]。坡垒为同源四倍体,减数分裂很难产生能育的配子,无融合结实是坡垒更新种群的必然选择。但无融合生殖通常不产生新的变异组合,不利于濒危物种遗传多样性恢复,加剧了坡垒濒危的困境。因此,必须做好现存坡垒个体的保护,以尽可能维持种群的遗传多样性。由于坡垒树体高大,很难直接观察到坡垒的花,后续研究可以通过去雄套袋实验进一步验证坡垒无融合生殖,并通过重测序数据分析评估子代与亲本的遗传差异,探究坡垒遗传变异的输入与维持机制。
-
繁殖方式是影响种群遗传变异产生与维持的重要因素,决定了物种的进化潜力。基于微卫星和单核苷酸多态性变异,本研究揭示坡垒的繁殖方式为无融合生殖,这与其同源四倍体的性质相符。明确繁殖方式一方面能够促进对坡垒种群进化历史、遗传变异的水平与分布的理解,另一方面也有助于制定合理的保护措施,促进坡垒种群规模与遗传多样性恢复。对于冠层乔木坡垒,很难直接观察和操纵其花部器官,因此基于分子标记的亲子两代变异模式分析是研究这类高大林木繁殖方式的有效手段。基于覆盖全基因组的SNP变异还能提供无融合生殖物种体细胞突变的准确估计,解析坡垒种群遗传变异的来源。后续研究可以创造条件利用去雄套袋实验验证坡垒的无融合生殖能力。
Apomixis of Hopea hainanensis Merrill & Chun based on SSR and whole genome SNP variations
-
摘要: 利用微卫星和单核苷酸多态性标记检测坡垒(Hopea hainanensis )母树与子代的基因型,由此推断坡垒的繁殖方式。微卫星标记显示10个坡垒家系的母本与子代在12个微卫星位点上具有完全相同的基因型组成。随机选择4个家系通过简化基因组测序分析单核苷酸多态性(SNP)的变异模式,发现母树和子代在绝大多数SNP变异位点上保持一致,有性生殖产生这一变异模式的可能性几乎为零。微卫星和SNP标记分析均支持坡垒的繁殖方式为无融合生殖。作为同源四倍体,坡垒不能通过减数分裂产生能育配子,无融合生殖生产种子是其维持和更新种群的必然途径。体细胞突变是无融合生殖产生变异的主要途径,因此,要恢复坡垒种群的遗传变异,必须严格保护现存的坡垒种群,避免多样性的进一步丧失;同时利用高通量测序筛选变异种苗,在自然生境中重建坡垒种群,逐步恢复遗传多样性,实现其长期稳定续存。Abstract: Hopea hainanensis Merrill & Chun, a large evergreen tree belonging to the family Dipterocarpaceae, is a representative species in tropical rainforests of Hainan Island. The population size of H. hainanensis has contracted seriously due to over exploitation and deforestation, and as a result this species has been listed as a national first-class protected plant in China. The reproductive system determines the transmission of genes between generations, which is an important research topic on endangered plant species. Mother trees of H. hainanensis and their seedlings were genotyped using both microsatellite markers and single nucleotide polymorphisms, on which the reproduction mode of this species was inferred. Results showed that the genotypes of the seedlings of 10 mother trees were identical to their corresponding parents at 12 microsatellite loci. Four randomly selected mother trees and their seedlings were further genotyped using single nucleotide polymorphisms generated by reduced-representation genome sequencing. Most of the SNP loci in the seedlings were found to be identical to their parents, which could be hardly explained by sexual reproduction. In summary, the reproduction of H. hainanensis was inferred to be apomictic based on both microsatellite and SNP markers. As an autotetraploid species, it is impossible for H. hainanensis to reproduce through meiosis, and apomixis is the only way to produce seeds thus maintaining its population. Somatic mutation is the main way to generate variation in apomictic species. In order to restore the genetic variation in H. hainanensis, the existing populations must be strictly conserved to avoid further loss of diversity. Moreover, high-throughput sequencing such as reduced-representation genome sequencing should be conducted to screen seedlings which are different from its parent. The seedlings with somatic mutations could be used to rebuild H. hainanensis populations in natural habitats, so that genetic diversity may be gradually restored and the long-term survival of this species could be achieved at last.
-
表 1 10个坡垒家系的编号、子代数量、采集地点与海拔
Table 1 Code, seedlings number, district and altitude of ten family lines of Hopea hainanensis
地区
District家系
Family line子代数量 SSR(SNP)
Number of seedlings
genotyped by SSR(SNP)经度
Longitude纬度
Latitude海拔/m
Altitude/m霸王岭
Bawang MountainBW1 20 109.1331 °E19.0985 °N461 BW2 20 109.1333 °E19.0985 °N458 BW3 20 109.1335 °E19.0984 °N465 BW4 20(9) 109.1159 °E19.0914 °N338 BW5 0 109.1449 °E19.0785 °N785 BW6 2 109.3017 °E19.0789 °N626 BW7 20(19) 109.1192 °E19.0782 °N741 BW8 20(20) 109.1183 °E19.0798 °N687 BW9 20 109.1178 °E19.0819 °N748 尖峰岭
Jianfeng MountainJF 21(20) 109.7529 °E18.7349 °N688 表 2 10个坡垒家系母树SSR基因型
Table 2 SSR genotypes of mother trees in ten family lines of Hopea hainanensis
HHA01 HHA03 HHA04 HHA11 HHA14 HHA24 HHA27 HHA32 HHA41 HHA49 HHA50 HHA62 BW1 103,108 / / / / / / / 126 / / 86,92 BW2 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW3 / / / 144,150 / 141,145 / / 126 / / / BW4 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW5 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW6 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW7 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW8 103,108 155,159 126,132 144,150 116 141,145 129,135 224,228 126 159 159,163 86,92 BW9 103,108 155,159 126,132 144,150 / 141,145 129,135 224,228 126 159 159,163 86,92 JF 103,108 159,163 126,134 144,150 116 143 133 224,230 126,129 159 157,167 84 注:“/”表示缺失数据。 Note:"/" indicates missing data. 表 3 4个坡垒家系母树与子代SNP基因型比较分析结果
Table 3 Comparative analysis of SNP genotype between mother trees and their seedlings in four family lines of Hopea hainanensis
家系
Familym=3 m=6 m=9 m=12 m=15 子代与母本
具有相同
SNP基因型
的比例
Prop. SNPs
the same as
its motherarent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因
型的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因
型的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因型
的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPs子代与母本
具有相同
SNP基因
型的比例
Prop. SNPs
the same as
its maternal
parent平均SNP
位点数
Mean no.
SNPsBW7 0.812 ± 0.057 2883.2 0.854 ± 0.051 1943.5 0.873 ± 0.048 1311.0 0.881 ± 0.045 920.3 0.883 ± 0.042 671.9 BW8 0.869 ± 0.062 3182.5 0.910 ± 0.052 2401.2 0.930 ± 0.048 1855.6 0.939 ± 0.042 1518.2 0.942 ± 0.042 1223.3 JF 0.857 ± 0.034 4189.7 0.888 ± 0.029 3026.5 0.911 ± 0.026 2310.0 0.926 ± 0.025 1822.5 0.935 ± 0.024 1478.7 BW4 0.814 ± 0.044 4518.1 0.859 ± 0.032 3225.6 0.883 ± 0.028 2377.7 0.902 ± 0.028 1880.8 0.917 ± 0.026 1530.4 -
[1] Ghazoul J. Dipterocarp biology, ecology, and conservation[M]. Oxford: Oxford University Press, 2016: 2. [2] Cvetković T, Hinsinger D D, Strijk J S. Exploring evolution and diversity of Chinese Dipterocarpaceae using next-generation sequencing [J]. Scientific Reports, 2019, 9(1): 11639. https://doi.org/10.1038/s41598-019-48240-y doi: 10.1038/s41598-019-48240-y [3] 中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京: 科学出版社, 2004: 120. [4] 肖云学, 殷霖昊, 郁文彬, 等. 迁地栽培坡垒的种群结构与幼苗更新研究[J]. 植物科学学报, 2023, 41(5): 604−612. https://doi.org/10.11913/PSJ.2095-0837.22294 doi: 10.11913/PSJ.2095-0837.22294 [5] 傅立国. 中国植物红皮书–第一册: 稀有濒危植物[M]. 北京: 科学出版社, 1991. [6] 国家林业和草原局, 农业农村部. 国家重点保护野生植物名录[EB/OL]. 2021[2024-11-27]. https://www.gov.cn/zhengce/zhengceku/2021-09/09/content_5636409.htm. [7] Ly V, Nanthavong K, Pooma R, et al. Hopea hainanensis[EB/OL]. The IUCN Red List of Threatened Species, 2018[2024-11-27]. https://dx.doi.org/10.2305/IUCN.UK.2018-1.RLTS.T32357A2816074.en. [8] 胡玉佳. 海南岛龙脑香森林的群落特征及其类型[J]. 生态科学, 1983, 2: 16−24. [9] Lan Q Y, Luo Y L, Ma S M, et al. Development and storage of recalcitrant seeds of Hopea hainanensis [J]. Seed Science and Technology, 2012, 40(2): 200−208. https://doi.org/10.15258/sst.2012.40.2.05 doi: 10.15258/sst.2012.40.2.05 [10] 路兴慧, 臧润国, 丁易, 等. 极小种群野生植物坡垒的生境特征及其对幼苗多度的影响[J]. 生物多样性, 2020, 28(3): 289−295. https://doi.org/10.17520/biods.2019143 doi: 10.17520/biods.2019143 [11] Wang C, Ma X, Ren M X, et al. Genetic diversity and population structure in the endangered tree Hopea hainanensis (Dipterocarpaceae) on Hainan Island, China [J]. PLoS One, 2020, 15(11): e0241452. https://doi.org/10.1371/journal.pone.0241452 doi: 10.1371/journal.pone.0241452 [12] Tang L, Long J Q, Wang H Y, et al. Conservation genomic study of Hopea hainanensis (Dipterocarpaceae), an endangered tree with extremely small populations on Hainan Island, China [J]. Frontiers in Plant Science, 2024, 15: 1442807. https://doi.org/10.3389/fpls.2024.1442807 doi: 10.3389/fpls.2024.1442807 [13] Wang R, Liu C N, Segar S T, et al. Dipterocarpoidae genomics reveal their demography and adaptations to Asian rainforests [J]. Nature Communications, 2024, 15(1): 1683. https://doi.org/10.1038/s41467-024-45836-5 doi: 10.1038/s41467-024-45836-5 [14] Tam N M, Duy V D, Duc N M, et al. Genetic variation and outcrossing rates of the endangered tropical species Dipterocarpus dyeri [J]. Journal of Tropical Forest Science, 2019, 31(2): 259−267. https://doi.org/10.26525/jtfs2019.31.2.259267 doi: 10.26525/jtfs2019.31.2.259267 [15] 张大勇. 植物生活史进化与繁殖生态学[M]. 北京: 科学出版社, 2004: 102. [16] Baskaware S V, Deodhar M A. Apomixis and sexual systems in various species of Garcinia with special reference to Garcinia indica (thouars) Choisy [J]. International Journal of Fruit Science, 2023, 23(1): 25−33. https://doi.org/10.1080/15538362.2023.2165596 doi: 10.1080/15538362.2023.2165596 [17] Asker S E, Jerling L. Apomixis in plants[M]. Boca Raton: CRC, 1992. [18] Appanah S, Turnbull J W. A review of dipterocarps: taxonomy, ecology and silviculture[M]. Bogor: Center for International Forestry Research, 1998: 48. [19] Dias A C C, Serra A C, Sampaio D S, et al. Unexpectedly high genetic diversity and divergence among populations of the apomictic Neotropical tree Miconia albicans [J]. Plant Biology, 2018, 20(2): 244−251. https://doi.org/10.1111/plb.12654 doi: 10.1111/plb.12654 [20] Sailer C, Stöcklin J, Grossniklaus U. Dynamics of apomictic and sexual reproduction during primary succession on a glacier forefield in the Swiss Alps [J]. Scientific Reports, 2020, 10(1): 8269. https://doi.org/10.1038/s41598-020-64367-9 doi: 10.1038/s41598-020-64367-9 [21] Xu Y, Zang R G. Conservation of rare and endangered plant species in China [J]. iScience, 2023, 26(2): 106008. https://doi.org/10.1016/j.isci.2023.106008 doi: 10.1016/j.isci.2023.106008 [22] Kaur A, Jong K, Sands V E, et al. Cytoembryology of some Malaysian dipterocarps, with some evidence of apomixis [J]. Botanical Journal of the Linnean Society, 1986, 92(2): 75−88. https://doi.org/10.1111/j.1095-8339.1986.tb01824.x doi: 10.1111/j.1095-8339.1986.tb01824.x [23] Ng K K S, Lee S L, Koh C L. Spatial structure and genetic diversity of two tropical tree species with contrasting breeding systems and different ploidy levels [J]. Molecular Ecology, 2004, 13(3): 657−669. https://doi.org/10.1046/j.1365-294X.2004.02094.x doi: 10.1046/j.1365-294X.2004.02094.x [24] Vignal A, Milan D, Sancristobal M, et al. A review on SNP and other types of molecular markers and their use in animal genetics [J]. Genetics Selection Evolution, 2002, 34(3): 275−305. https://doi.org/10.1186/1297-9686-34-3-275 doi: 10.1186/1297-9686-34-3-275 [25] Freeland J R. Kirk H, Petersen S D. 分子生态学[M]. 戎俊, 杨小强, 耿宇鹏, 等, 译. 2版. 北京: 高等教育出版社, 2015: 24. [26] Al-Samarai F R, Al-Kazaz A A. Molecular markers: an introduction and applications [J]. European Journal of Molecular Biotechnology, 2015, 9(3): 118−130. https://doi.org/10.13187/ejmb.2015.9.118 doi: 10.13187/ejmb.2015.9.118 [27] Zhao Y, Wang K, Wang W L, et al. A high-throughput SNP discovery strategy for RNA-seq data [J]. BMC Genomics, 2019, 20(1): 160. https://doi.org/10.1186/s12864-019-5533-4 doi: 10.1186/s12864-019-5533-4 [28] Kleven O, Endrestøl A, Evju M, et al. SNP discovery in the northern dragonhead Dracocephalum ruyschiana [J]. Conservation Genetics Resources, 2019, 11(4): 431−435. https://doi.org/10.1007/s12686-018-1045-9 doi: 10.1007/s12686-018-1045-9 [29] Chen Y K, Zhang H L, Zhang L, et al. Genetic diversity assessment of Hopea hainanensis in Hainan Island [J]. Frontiers in Plant Science, 2022, 13: 1075102. https://doi.org/10.3389/fpls.2022.1075102 doi: 10.3389/fpls.2022.1075102 [30] Sousa T V, Caixeta E T, Alkimim E R, et al. Population structure and genetic diversity of coffee progenies derived from Catuaí and Híbrido de Timor revealed by genome-wide SNP marker [J]. Tree Genetics & Genomes, 2017, 13(6): 124. https://doi.org/10.1007/s11295-017-1208-y doi: 10.1007/s11295-017-1208-y [31] Wang C, Ma X, Tang L. Isolation and characterization of twelve polymorphic microsatellite markers in the endangered Hopea hainanensis (Dipterocarpaceae) [J]. Ecology and Evolution, 2021, 11(1): 4−10. https://doi.org/10.1002/ece3.7077 doi: 10.1002/ece3.7077 [32] Peterson B K, Weber J N, Kay E H, et al. Double digest RADseq: an inexpensive method for de novo SNP discovery and genotyping in model and non-model species [J]. PLoS One, 2012, 7(5): e37135. https://doi.org/10.1371/journal.pone.0037135 doi: 10.1371/journal.pone.0037135 [33] Rochette N C, Rivera-Colón A G, Catchen J M. Stacks 2: analytical methods for paired-end sequencing improve RADseq-based population genomics [J]. Molecular Ecology, 2019, 28(21): 4737−4754. https://doi.org/10.1111/mec.15253 doi: 10.1111/mec.15253 [34] Rochette N C, Catchen J M. Deriving genotypes from RAD-seq short-read data using Stacks [J]. Nature Protocols, 2017, 12(12): 2640−2659. https://doi.org/10.1038/nprot.2017.123 doi: 10.1038/nprot.2017.123 [35] Kaushal P, Dwivedi K K, Radhakrishna A, et al. Partitioning apomixis components to understand and utilize gametophytic apomixis [J]. Frontiers in Plant Science, 2019, 10: 256. https://doi.org/10.3389/fpls.2019.00256 doi: 10.3389/fpls.2019.00256 [36] 姚家玲. 龙须草无融合生殖机理研究及资源评价[D]. 武汉: 华中农业大学, 2005. [37] Kaur A, Ha C O, Jong K, et al. Apomixis may be widespread among trees of the climax rain forest [J]. Nature, 1978, 271(5644): 440−442. https://doi.org/10.1038/271440a0 doi: 10.1038/271440a0 [38] Singh A N, Thakur A. Polyembryony in Dipterocarpus retusus [J]. Journal of Tropical Forest Science, 2004, 16(4): 475−476. [39] Pangsuban S, Bamroongrugsa N, Kanchanapoom K, et al. Facultative apomixis in Garcinia atroviridis (Clusiaceae) and effects of different pollination regimes on reproductive success [J]. Tropical Life Sciences Research, 2009, 20(2): 89−108. [40] Majeský Ľ, Vašut R J, Kitner M. Genotypic diversity of apomictic microspecies of the Taraxacum scanicum group (Taraxacum sect. Erythrosperma) [J]. Plant Systematics and Evolution, 2015, 301(8): 2105−2124. https://doi.org/10.1007/s00606-015-1218-x doi: 10.1007/s00606-015-1218-x [41] Yu F Y, Xu W J, Xiao Y E, et al. Identifying apomixis in matroclinal progeny from an interspecific crossing between Iris domestica and three different colors of Iris dichotoma [J]. Euphytica, 2017, 213(12): 273. https://doi.org/10.1007/s10681-017-2065-3 doi: 10.1007/s10681-017-2065-3 [42] Koltunow A M, Grossniklaus U. Apomixis: a developmental perspective [J]. Annual Review of Plant Biology, 2003, 54: 547−574. https://doi.org/10.1146/annurev.arplant.54.110901.160842 doi: 10.1146/annurev.arplant.54.110901.160842 [43] Hörandl E, Paun O. Patterns and sources of genetic diversity in apomictic plants: implications for evolutionary potentials[M]//Hörandl E, Grossniklaus U, Van Dijk P, et al. Apomixis: evolution, mechanisms and perspectives. Liechtenstein: ARG-Gantner Ruggell, 2007: 169−174. [44] 刘超男. 龙脑香亚科适应热带冠层的分子基础与海南岛坡垒的种群历史动态[D]. 上海: 华东师范大学, 2022. [45] Sowa S, Kulik M, Koroluk A, et al. Genetic structure of Carlina acanthifolia subsp. utzka populations on the north-western margins of the species range [J]. Global Ecology and Conservation, 2020, 24: e01225. https://doi.org/10.1016/j.gecco.2020.e01225 doi: 10.1016/j.gecco.2020.e01225 -

 点击查看大图
点击查看大图
计量
- 文章访问数: 161
- HTML全文浏览量: 122
- 被引次数: 0



 下载:
下载: