-
自从青霉素的发现和临床使用以来,抗生素拯救了许多人的生命,使人免于细菌感染。作为抑制细菌生长或杀死细菌的化合物,抗生素在较低浓度时就可以抑制微生物的生长或增殖[1]。20世纪60年代末,由于人们认为细菌感染不再构成威胁,新抗生素的开发速度放缓,因此,新抗生素的开发减少。如今,细菌耐药性问题已经成为人类健康安全重大危机,耐药型超级细菌的出现,使得抗生素效力大打折扣,且新发现的抗生素也越来越少,促使研发针对新的作用靶点、具有新作用机制的抗菌药物成为当务之急[2-3]。丝状温度敏感蛋白Z(Filament temperature-sensitive protein Z,FtsZ)是一种原核细菌分裂中不可或缺的分裂蛋白,在金黄色葡萄球菌、大肠杆菌、结核杆菌等细菌中广泛存在[4-5],且在体外和体内都能以依赖GTP结合的方式水解GTP为自身聚合提供能量[5-7],现已有高分辨率蛋白质X射线结晶学提供了FtsZ的详细结构信息[8],此外,不同的体外实验也可用于鉴定FtsZ蛋白。在细菌细胞进行二分裂过程中,FtsZ是第一个定位到细菌分裂层面的蛋白质,聚合成Z环后,环体会招募下游蛋白,组装成被称为分裂蛋白复合物[9-10]的大分子复合体以完成后续分裂活动。扰乱FtsZ的聚合作用可以抑制细菌分裂期隔膜的形成,若在细胞质分裂期间隔膜不能正确形成,则会观察到丝状或形态异常的细胞[11],进而导致细菌死亡。虽然细胞质分裂过程中调节Z环的聚合−解聚速率的机制尚未可知,但FtsZ蛋白N-末端的GTP结合部位和C-末端的T7-环对Z-环的形成起重要作用,针对这2个区域的FtsZ抑制剂可以干扰FtsZ蛋白的聚合或GTPase活性,或两者兼而有之。作为微管蛋白同系物,尽管超过80%的FtsZ序列与微管蛋白的序列不同[7]且FtsZ和微管蛋白之间的C-末端结构域的氨基酸序列不太保守,但是从FtsZ和微管蛋白的三维结构中可以看出,FtsZ和微管蛋白的N端结构域和C端结构域非常相似,这表明FtsZ和微管蛋白在生物体内可能发挥着相似的功能。微管蛋白已作为抗癌药物、抗原生动物药剂靶点被广泛研究[12-13],说明FtsZ同样具有成为药物靶点的潜力,且因为FtsZ与微管蛋白的低同源性,靶向FtsZ的抑制剂将会对真核细胞具有良好的选择性和较低的细胞毒性,这使得FtsZ成为一个有吸引力的新型抗菌靶点。
近年来,已有许多关于FtsZ蛋白靶向药物的研究,它们通过干扰FtsZ的GTPase活性、Z-ring的动力学过程或者破坏FtsZ的结构而发挥作用,包括天然抑制剂,如姜黄素、白花丹素、桃拓酚、黄连素、香豆素类、肉桂醛、白藜芦醇、小檗碱、血根碱、白屈菜亦碱、陶塔酚、绿垂毒素、紫杉烷类等[14];合成抑制剂如3-MBA[15]、PC190723[16]、3-苯基-异喹啉(3-phenyl-isoquinoline)[17]、三取代苯并咪唑(trisubstituted benzimidazoles)[18-19]等。传统药物筛选虽然筛选效果直观,但十分耗费人力、物力,虚拟筛选技术可以避免这些弊端,极大提高化合物的有效命中率。
本研究利用Python语言编写ChemScript脚本,构建二肽、三肽、四肽及五肽化合物结构数据库,通过分子对接筛选得到多种与FtsZ结合的短肽化合物。利用分子动力学方法动态分析各短肽与FtsZ的结合状态,解决分子对接忽视时间维度以及未直接体现水分子溶剂效应的问题。在此基础上,通过抑菌试验评价其对金黄色葡萄球菌(Staphylococcus aureus)、枯草芽孢杆菌(Bacillus subtilis)的抑菌活性以及对FtsZ的GTP酶活性的影响,旨在探究靶向FtsZ的潜在肽类抑制剂和作用机制。
-
1)质粒、菌株 金黄色葡萄球菌FtsZ蛋白的表达菌:pET28a-FtsZ(BL21)由笔者所在的实验室构建、储存;金黄色葡萄球菌菌株Staphylococcus aureus 04-02981、Bacillus subtilis 168由笔者所在的实验室储存。2)试剂 Phosphate Assay Kit(Abnova,中国台湾)、GTP(上海生工)、多肽TE101、PE101、PE102、PE103、PE104(上海生工)。3) 仪器 酶标仪MR-96A(迈瑞)。
-
从蛋白质结构数据库(Protein Data Bank,PDB)下载金黄色葡萄球菌FtsZ蛋白的晶体结构(PDB#4DXD)[20]。利用MOE分子对接软件,选用Amber99力场,修正蛋白质结构数据中包含的错误后,利用构建好的5个数据库分别进行对接。对接模式为诱导契合法,利用GBVI/WSA ΔG方法对它们之间的结合自由能做最终评价[21]。GBVI/WSA ΔG的计算公式如下,当 ΔG数值越负,表明配体与受体之间的引力越强。
$$ \mathrm{\Delta }\mathrm{G}\approx \mathrm{c}+\mathrm{\alpha }\left[\frac{2}{3}\left({\mathrm{\Delta }\mathrm{E}}_{Coul}+{\mathrm{\Delta }\mathrm{E}}_{Sol}\right)+{\mathrm{\Delta }\mathrm{E}}_{vdW}+{\beta \mathrm{\Delta }SA}_{weighted}\right] \text{,} $$ (1) 式中:ΔECoul、ΔEsol、ΔEvdW分别是静电、溶剂及范德华作用,ΔSA是溶剂暴露面积。
-
对接完成后,挑选打分靠前的配体,保存其与蛋白质结合的PDB结构,利用安装在Ubuntu Linux(18.06)操作系统上的GROMACS(2020.03)探究蛋白质与配体之间的相互作用随时间的动态变化,分析过程简述如下。首先生成FtsZ与配体复合物的GROMACS格式的空间结构文件及拓扑文件。选用针对蛋白质从头计算加以优化的AMBER99SB全原子力场,以及TIP3P显式水分子模型。创建具有周期性边界的正十二面体计算单元,将蛋白质复合物放置在单元盒中央,蛋白质复合物与单元盒边界的距离为1.0 nm。以水分子填充单元盒,以Na+和Cl−随机取代水分子使系统呈电中性,NaCl的终浓度为0.1 mol·L−1。利用最大斜率下降法使系统能量最小化,终止阈值为1000 kJ·(mol·nm)−1。能量最小化后,系统在等温−等容条件下使用速度重新标度法,通过调整标准Berendsen恒温器以获得正确的动能分布。然后利用Parrinello-Rahman压力耦合算法使系统达到等温−等压状态。系统平衡后,进行50 000 000步分子动力学综合运算(Production)。在每个步骤中,利用兰纳−琼斯势能以及库伦势能方程计算系统中所有原子之间的作用力及运动状态;其中,远距离静电相互作用采用平滑粒子−网格Ewald总和法计算。利用基数样条插值法将电荷分配给网格,然后进行三维快速傅里叶变换。静电力由傅里叶空间中的相互作用力反推得到。
RMSD(Root Mean Square Deviation)值计算公式为:
$$ {{\rm{RMSD}}}_{{t}_{1},{t}_{2}}={\left[\frac{1}{M}{\sum} _{i=1}^{N}{m}_{i}\left|\right|{{r}}_{i}\left({t}_{1}\right)-{{r}}_{i}\left({t}_{2}\right){\left|\right|}^{2}\right]}^{\frac{1}{2}} \text{,} $$ (2) 式中:ri是原子i在t时间的位置,而M是原子质量(m)的总和。
相互作用能(Interaction energy)为兰纳−琼斯势能(3)与库伦势能(4)之和。
$$ {V}_{LJ}=\frac{{C}_{ij}^{\left(12\right)}}{{r}_{ij}^{12}}-\frac{{C}_{ij}^{\left(6\right)}}{{r}_{ij}^{6}} \text{,} $$ (3) $$ {V}_{C}=f\frac{{q}_{i}{q}_{j}}{{\varepsilon }_{r}{r}_{ij}} \text{,} $$ (4) 式中:r是位置矢量长度,q是基电荷等于1.602 176 565×10−19C,f是电转换因子,等于1/4πε0或138.935 458 kJ·mol−1·nm e−2。
用LB液体培养基稀释法测定化合物的抑菌活性,首先将适量的LB液体培养基在高压水蒸气灭菌锅灭菌后,放入超净台紫外灭菌备用,向无菌96孔板内加入LB培养基,将目标化合物溶液(0.01 mol·L−1)和对照组溶液,分别加入96孔板各列的第一个孔内,其次用二倍稀释法稀释到第10孔,最后接种配置好的菌液(最后一列除外)。在37 ℃恒温培养箱培养24 h后,用酶标仪测定各孔的OD600值,所得各孔的数据由公式(5)处理可得到各化合物的抑菌活性。为了观察化合物在细菌生长阶段的不同影响,本研究还使用酶标仪的动力学程序,振板培养化合物处理的细菌,并以1 h为时间间隔连续测量其OD600值,绘制其生长曲线,每个实验重复3次。
-
用LB液体培养基稀释法测定化合物的抑菌活性,首先将适量的LB液体培养基在高压水蒸气灭菌锅灭菌后,放入超净台紫外灭菌备用,向无菌96孔板内加入LB培养基,将目标化合物溶液(0.01 mol·L−1)和对照组溶液,分别加入96孔板各列的第一个孔内,其次用二倍稀释法稀释到第10孔,最后接种配置好的菌液(最后一列除外)。在37℃恒温培养箱培养24 h后,用酶标仪测定各孔的OD600值,所得各孔的数据由公式(5)处理可得到各化合物的抑菌活性。为了观察化合物在细菌生长阶段的不同影响,本研究还使用酶标仪的动力学程序,振板培养化合物处理的细菌,并以1 h为时间间隔连续测量其OD600值,绘制其生长曲线,每个实验重复3次。
$$ \mathrm{抑}\mathrm{制}\mathrm{率}=1-\frac{{{O}{D}}_{\mathrm{试}\mathrm{验}\mathrm{孔}}-{{O}{D}}_{\mathrm{背}\mathrm{景}\mathrm{孔}}}{{{O}{D}}_{\mathrm{空}\mathrm{白}\mathrm{孔}}-{{O}{D}}_{\mathrm{背}\mathrm{景}\mathrm{孔}}} 。 $$ (5) -
GTP酶活性的测定使用Phosphate Assay Kit(Abnova,Taiwan China)进行测定,该试剂盒测量GTP在水解释放的无机磷浓度,该测定方法基于孔雀石绿钼酸盐与游离正磷酸盐在酸性条件下形成络合物。在620~640 nm处测得的绿色磷钼酸络合物的形成与游离有机磷的浓度直接相关,该测定的应用包括定量蛋白质磷酸酶底物的磷酸化和磷酸盐释放[22-23]。
将纯化得到的重组金黄色葡萄球菌FtsZ(6 µmol·L−1)与系列稀释的化合物在室温下与50 mmol·L−1 MOPS缓冲液(pH 6.5)中于96孔板中孵育10 min,对照样品中含有1%的DMSO,然后向混合物中加入200 mmol·L−1氯化钾和5 mmol·L−1氯化镁,最后加入500 mmol·L−1 GTP,在37℃孵育30 min。孵育结束后,向各孔中加入5 μL MG Acidic Solution试剂在室温下反应10 min,最后再向各孔中加入15 μL MG Blue Solution试剂,室温下孵育20 min后用酶标仪测得各孔在620 nm处的吸收光强度值,根据磷酸根浓度吸光度标准曲线,计算得到混合物中的磷酸根浓度,用以表征酶活性,并通过与空白组相对比,计算得到多肽对于FtsZ酶活的抑制情况,每个实验重复3次。
-
函数(公式1)综合考虑了平动/转动熵、静电效应、溶剂效应、范德华效应、溶剂暴露面积等要素,计算结果相对准确,常用于药物虚拟筛选和构效关系探究。根据MOE的初步虚拟筛选,所有对接多肽库的结果根据评分进行从高到低排序,同时综合考虑它们与蛋白的成键数目后,命中了系列多肽化合物,其对接细节见表1。从表1可知:PE101具有最低的打分值,为−11.933 9,其E-refine值也最低,为−58.525 7,说明根据分子对接结果来看,PE101具有最好的结合稳定性。类似的,其他PE多肽的打分值也很低,但E-refine值的大小却不尽相同。相较于PE多肽而言,TE102也具有较低的打分值,但TE101具有更低的E-refine,为进一步验证短肽配体与蛋白质的结合稳定性,笔者进行了分子动力学模拟计算。
表 1 对接结果数据
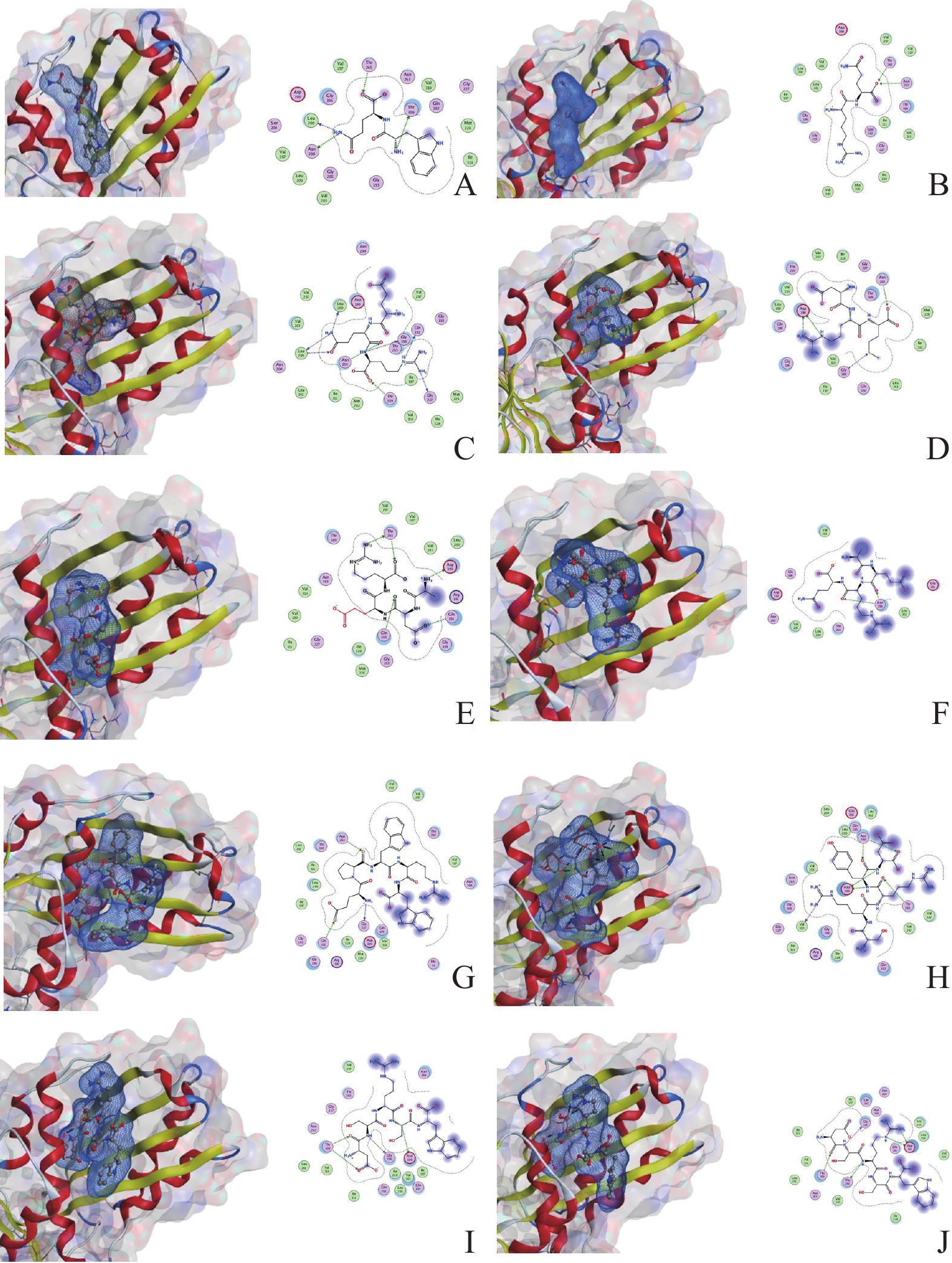
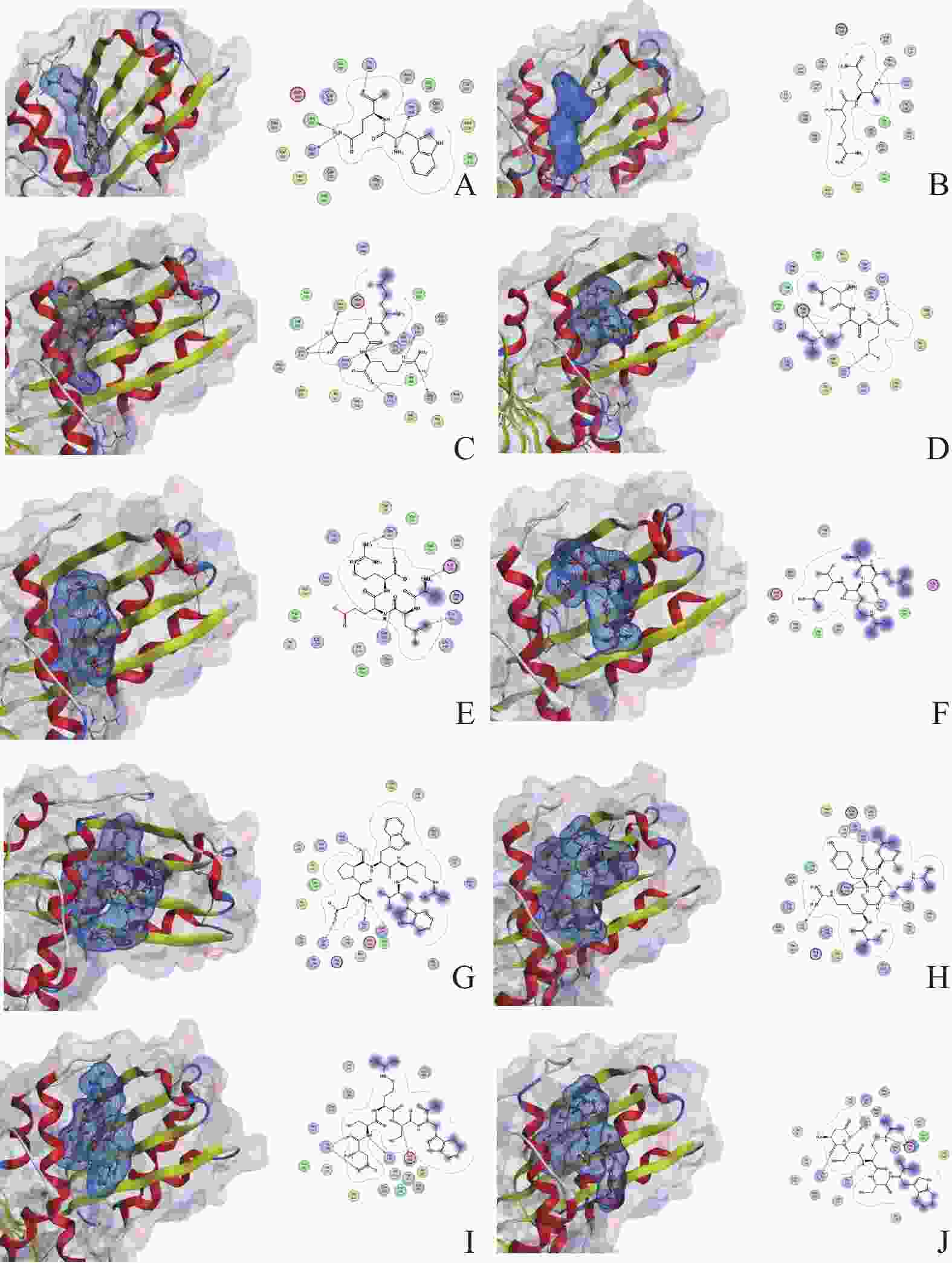
配体 S Rmsd-refine E-conf E-place E-score1 E-refine D101 −7.7623 1.9002 −430.7451 −62.9163 −7.1529 −36.6142 D102 −8.0550 1.5846 −237.1169 −70.7354 −9.6892 −33.6909 TR101 −9.4474 2.6825 −536.4507 −52.6038 −6.8441 −50.7704 TR102 −8.9825 1.8490 −371.1595 −72.7251 −6.7891 −46.7827 TE101 −9.3997 3.2524 −465.5232 −88.2899 −7.7524 −51.8134 TE102 −9.6111 1.7341 −498.0332 −87.8680 −6.6491 −39.1319 PE101 −11.9339 3.3185 −400.3169 −25.8219 −2.1784 −58.5257 PE102 −11.0793 2.8318 −572.0369 −43.4562 −0.8752 −49.9537 PE103 −10.0457 2.9602 −487.5392 −27.8190 −4.2072 −63.7616 PE104 −10.2516 2.6604 −373.5583 −42.5808 −5.0850 −61.7718 注:S值是对接的最终得分,Rmsd-refine表示对接之前的姿势与对接之后的姿势之间的均方根偏差细化,E-conf表示构象异构体的能量,默认情况下,此能量通过将溶剂化选项设置为Born来计算。E-place表示空间适配度的打分,E-score1表示第一次评分,E-refine是范德瓦尔斯静电和溶解能之和。 为了更清楚地阐述蛋白质与小分子配体的相互作用方式,图1展示了上述配体与FtsZ蛋白受体的对接空间位置结构示意图,并对不同配体与蛋白产生相互作用的氨基酸及作用形式进行汇总(表2),根据表2可知,所有的短肽大都倾向于和FtsZ蛋白的ASP199、LEU200、LEU209产生相互作用,且其形成的氢键的原子距离稳定在3 Å左右,表明形成的氢键稳定且可靠。
表 2 小分子和蛋白的氢键相互作用
配体 受体 相互作用 原子距离/Å 能量 /( kcal·moL-1) D101 N1 OG1 THR309 H-donor 2.91 −6.6 NE2 O LEU200 H-donor 2.86 −4.1 NE2 OD1 ASN208 H-donor 3.06 −4.0 OC1 OG1 THR265 H-acceptor 2.68 −1.8 D102 OC2 ND2 ASN263 H- acceptor 3.52 −0.5 OC2 OG1 THR265 H- acceptor 2.60 −2.5 TR101 NE2 O LEU200 H-donor 2.92 −3.1 NE2 O LEU209 H-donor 2.96 −0.5 N O GLY196 H-donor 3.21 −1.7 NE O GLN192 H-donor 3.29 −1.5 NH1 O GLY227 H-donor 2.87 −4.9 OE1 N LEU209 H-acceptor 3.17 −1.5 O ND2 ASN263 H-acceptor 2.72 −2.6 OC1 OG1 THR309 H-acceptor 2.54 −2.8 TR102 NE OD2 ASP199 H-donor 2.86 −8 NH2 OD2 ASP199 H-donor 2.69 −8.5 OD2 OG1 THR265 H-acceptor 2.6 −3.5 SD CA GLY193 H-acceptor 3.76 −1 NE OD2 ASP199 Ionic 2.86 −5.5 NH2 OD2 ASP199 Ionic 2.69 −7 TE101 N1 OD2 ASP199 H-donor 2.68 −8.0 OD1 CG GLN195 H- acceptor 2.80 −2.7 NH2 OG1 THR265 H-donor 2.80 −2.7 O2 CB GLN192 H-acceptor 2.82 −0.5 N1 OD2 ASP199 Ionic 2.79 −7.0 OC1 OG1 THR265 H-donor 2.67 −3.5 TE102 O2 ND2 ASN299 H-acceptor 2.87 −1.1 O5 ND2 ASN299 H-acceptor 3.15 −0.6 PE101 N1 O GLY227 H-donor 2.88 −5.1 OE1 NE2 GLN192 H-acceptor 2.92 −4.9 O3 ND2 ASN263 H-acceptor 2.73 −2.9 PE102 NH1 O VAL310 H-donor 3.06 −2.3 N6 OD2 ASP199 H-donor 2.82 −7.3 N8 OD2 ASP199 H-donor 3.34 −1.4 O6 OG1 THR265 H-acceptor 2.95 −2 O8 ND2 ASN299 H-acceptor 2.94 −2.3 OC1 ND2 ASN299 H-acceptor 2.74 −2.7 PE103 N1 OG LY196 H-donor 2.83 −4.5 CA5 OD2 ASP199 H-donor 3.4 −0.7 OD1 CB THR309 H-acceptor 3.32 −0.8 O1 OG1 THR309 H-acceptor 2.65 −1.4 PE104 N1 O GLY196 H-donor 2.96 −3.4 N2 OG1 THR265 H-donor 3.21 −1.3 NE3 OD1 ASP199 H-donor 3.02 −3.7 NH2 OD1 ASP199 H-donor 2.82 −9.8 O1 OG1 THR309 H-acceptor 2.55 −1.6 NE3 OD1 ASP199 Ionic 3.02 −4.3 NH2 OD1 ASP199 Ionic 2.82 −5.8 注:配体一列表示多肽小分子配体中与FtsZ蛋白形成氢键的具体原子;受体一列表示FtsZ蛋白中与小分子配体形成氢键的具体氨基酸及特定原子;相互作用表示其作用类型;原子距离表示形成氢键的两原子之间的距离长短;能量表示该氢键的能量高低。 -
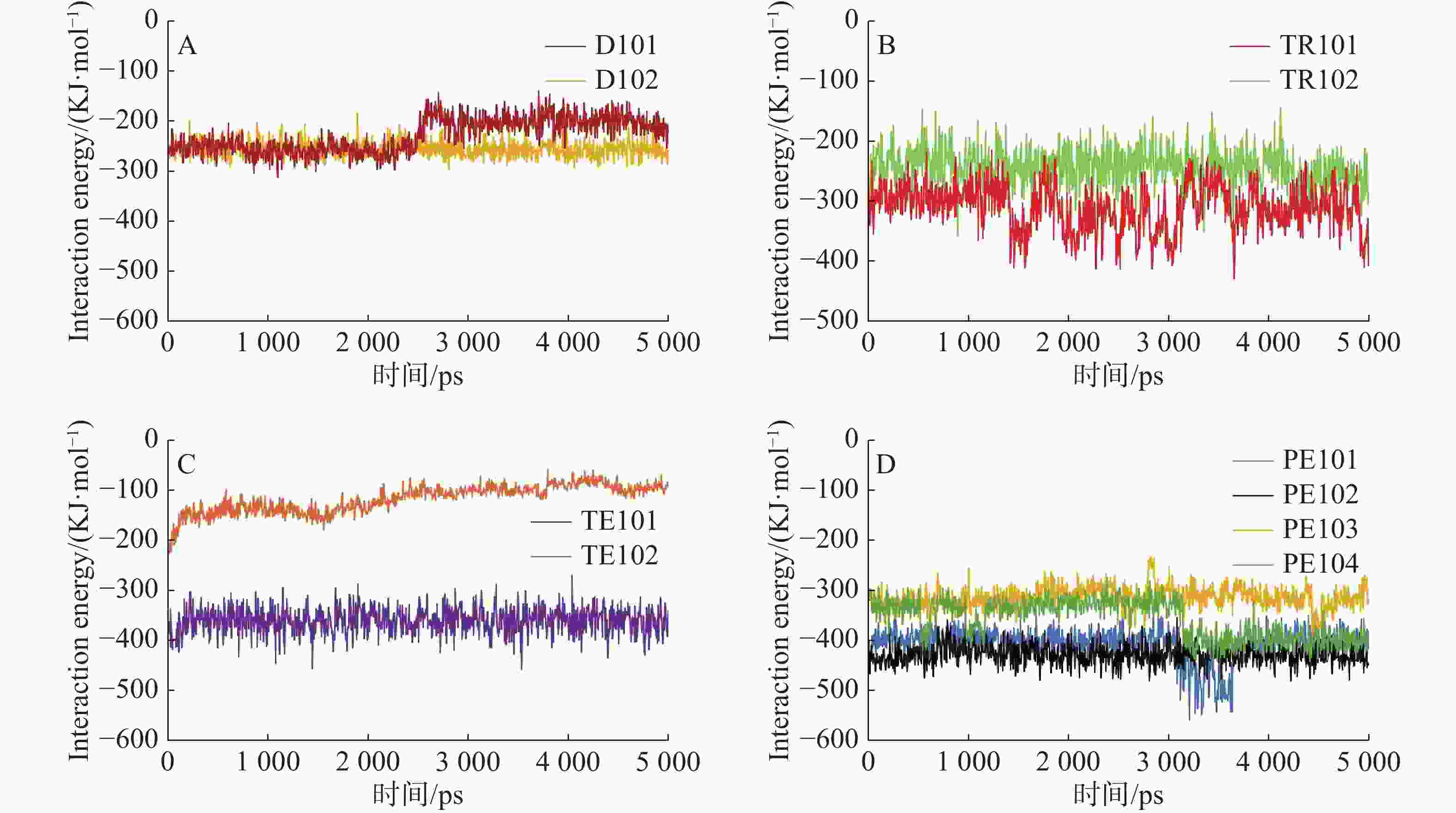
由于分子对接为静态分析,缺少对时间维度上原子运动以及水分子直接作用的分析,所以,本研究应用分子动力学方法动态分析在水溶液系统中所有原子之间的相互作用以及运动状态,从而验证小分子配体与FtsZ的相互作用强弱和结合稳定性。
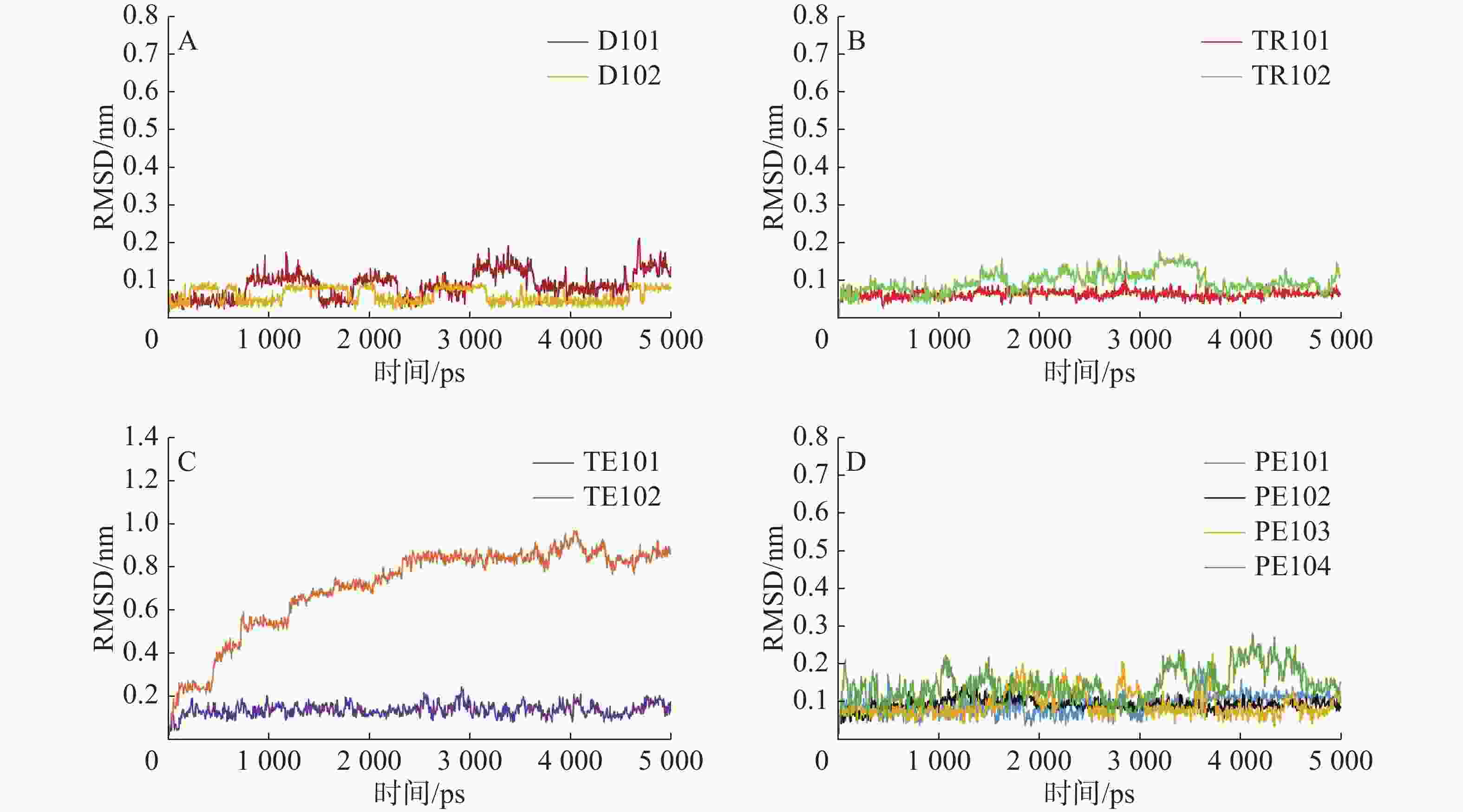
D101、D102与FtsZ分子动力学模拟过程中的RMSD值计算结果如图2-A所示,两个二肽均可以在FtsZ的PC口袋中稳定存在,二者存在周期性的RMSD值跳跃,其缘由可能是二肽体积较小,不能很好的在空间结构上契合蛋白口袋,这也是导致二态打分值较低的原因;三肽TR102也存在与二肽相同的情况,但其空间位置变化较为无序,与前者周期性重复不同,可能是因为结合亲和力较低,相反的,TR101能稳定地与FtsZ口袋结合,且RMSD值稳定在0.1以下(图2-B);四肽TE101无法在PC口袋中稳定存在,RMSD值随时间增加而持续增加,且在5 000 ps附近也不见稳定趋势,AERQ虽能在空间上大体保持位置不移动,但由图2-C能看出其稳定性也较差,波动范围较大;从图2-D可知,PE101、PE102五肽在口袋中的结合稳定性要优于PE103、PE104,其中PE104波动最为剧烈,但RMSD值始终稳定在0.1~0.2之间。
在蛋白质−配体相互作用的动力学分析中[24-25],相互作用能可用于表征2组相互作用分子之间的能量变化,由库仑势能和LJ势能组成,库仑势能表征分子之间的静电相互作用,LJ势能是2种物质在近距离内以相互排斥为主,而在长距离上则以相互吸引为主,表征了分子间范德华力的相互作用,相互作用能值越低,表明系统更稳定。相互作用能分析结果(图3)显示,PE101、PE102、PE103、PE104、TE101具有更低的相互作用能,说明其结合更为稳定。
-
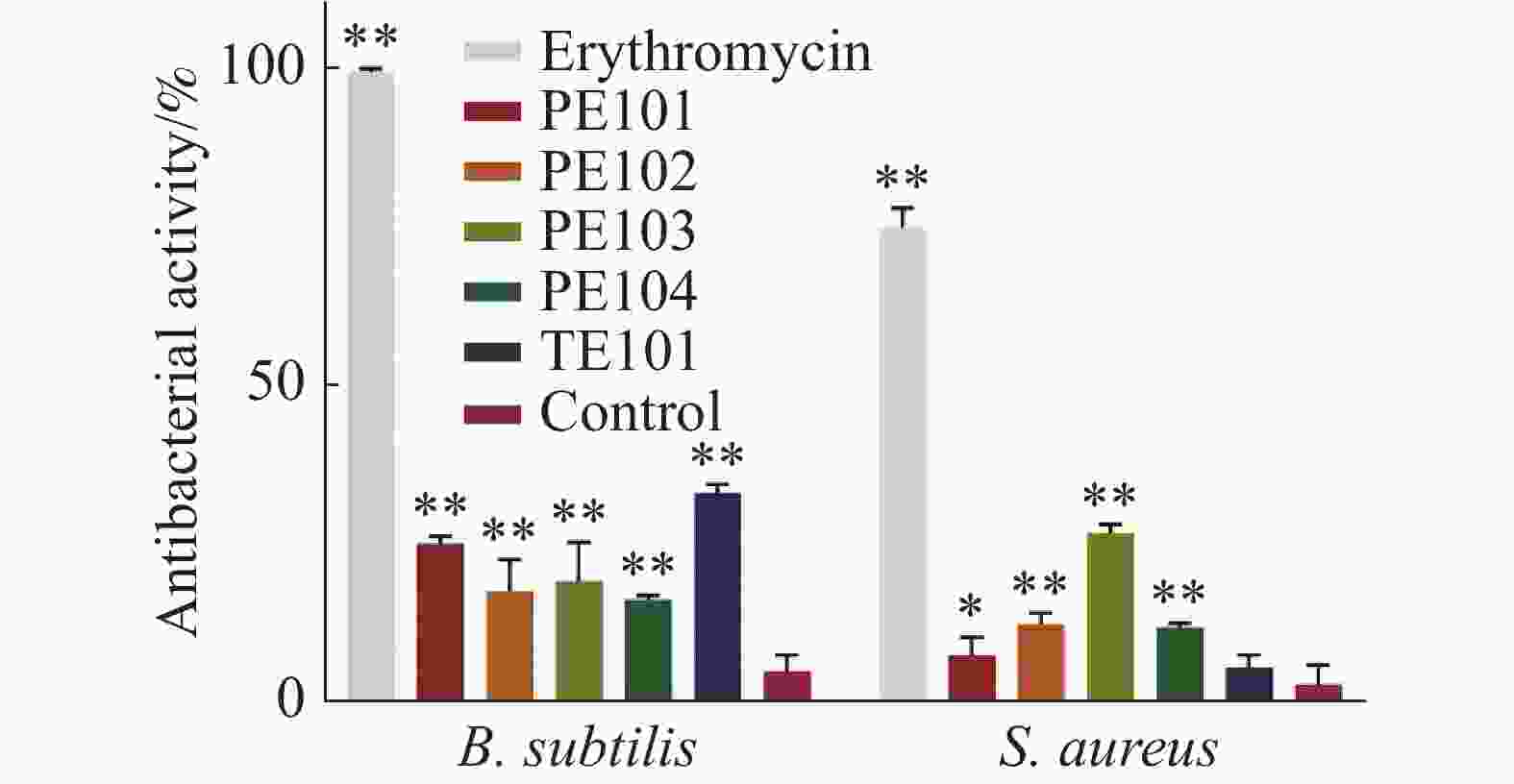
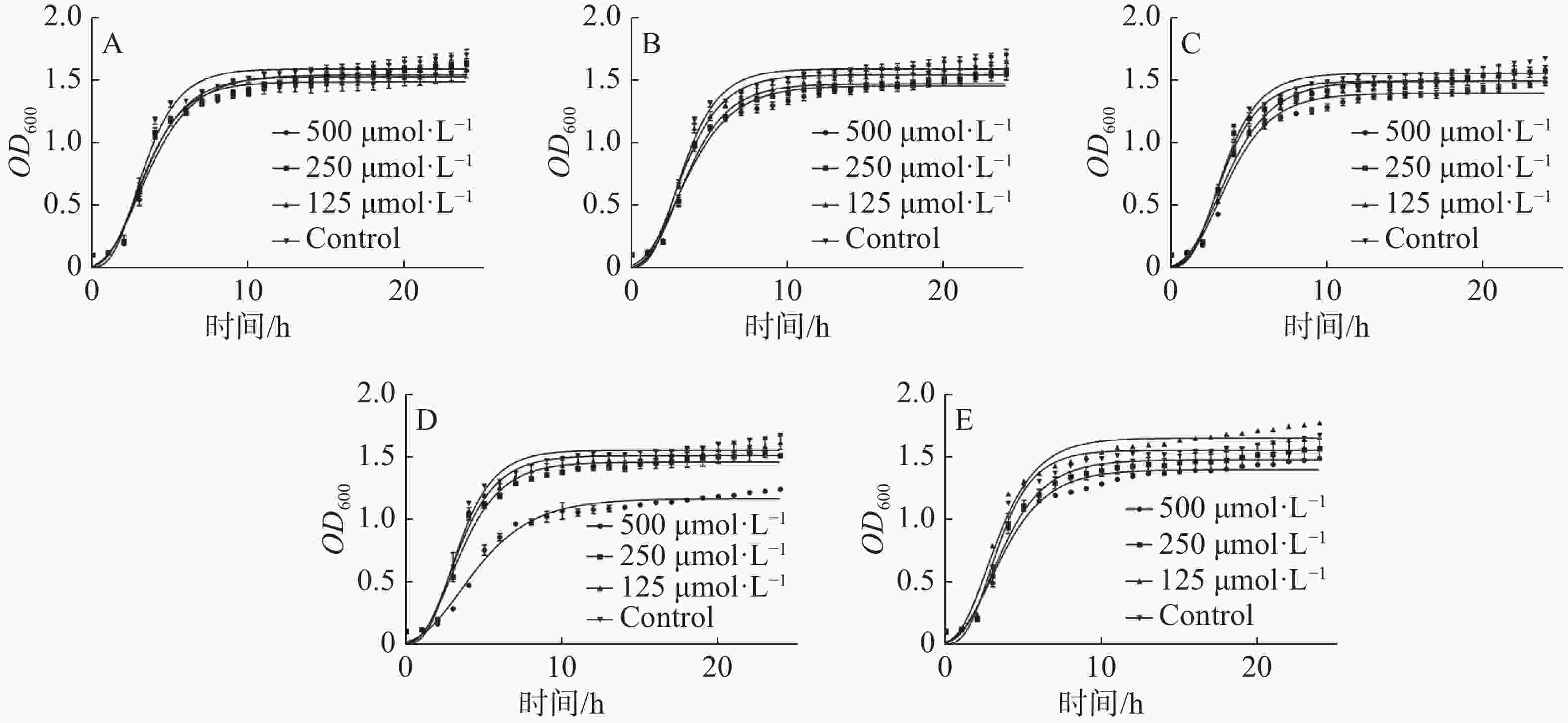
根据分子对接及分子动力学分析结果,选择PE101、PE102、PE103、PE104、TE101这5个多肽进行抑菌活性测定。本实验采用肉汤微量稀释法进行多肽对金黄色葡萄球菌及枯草芽孢杆菌的抑菌活性测试,实验根据CLSI(Clinical and Laboratory Standards Institute)标准,所得结果显示(图4),多肽在500 μmol·L−1的浓度下对金黄色葡萄球菌的抑制活性差距较大,PE103在其中效果最好,抑制率为25%左右,剩余多肽抑制率均在10%左右;各多肽对于枯草芽孢杆菌的作用略强于金黄色葡萄球菌,作用最强的TE101抑制率为25%,其他的五肽抑制率也达到了20%左右。另外本研究中测定了各浓度化合物处理后的细菌生长曲线,观察药物在各阶段对金黄色葡萄球菌生长的影响(图5),不难看出PE103(图5-D)能使细菌的对数生长期生长速率变缓,这可能是由于细菌细胞分裂受阻所导致。
-
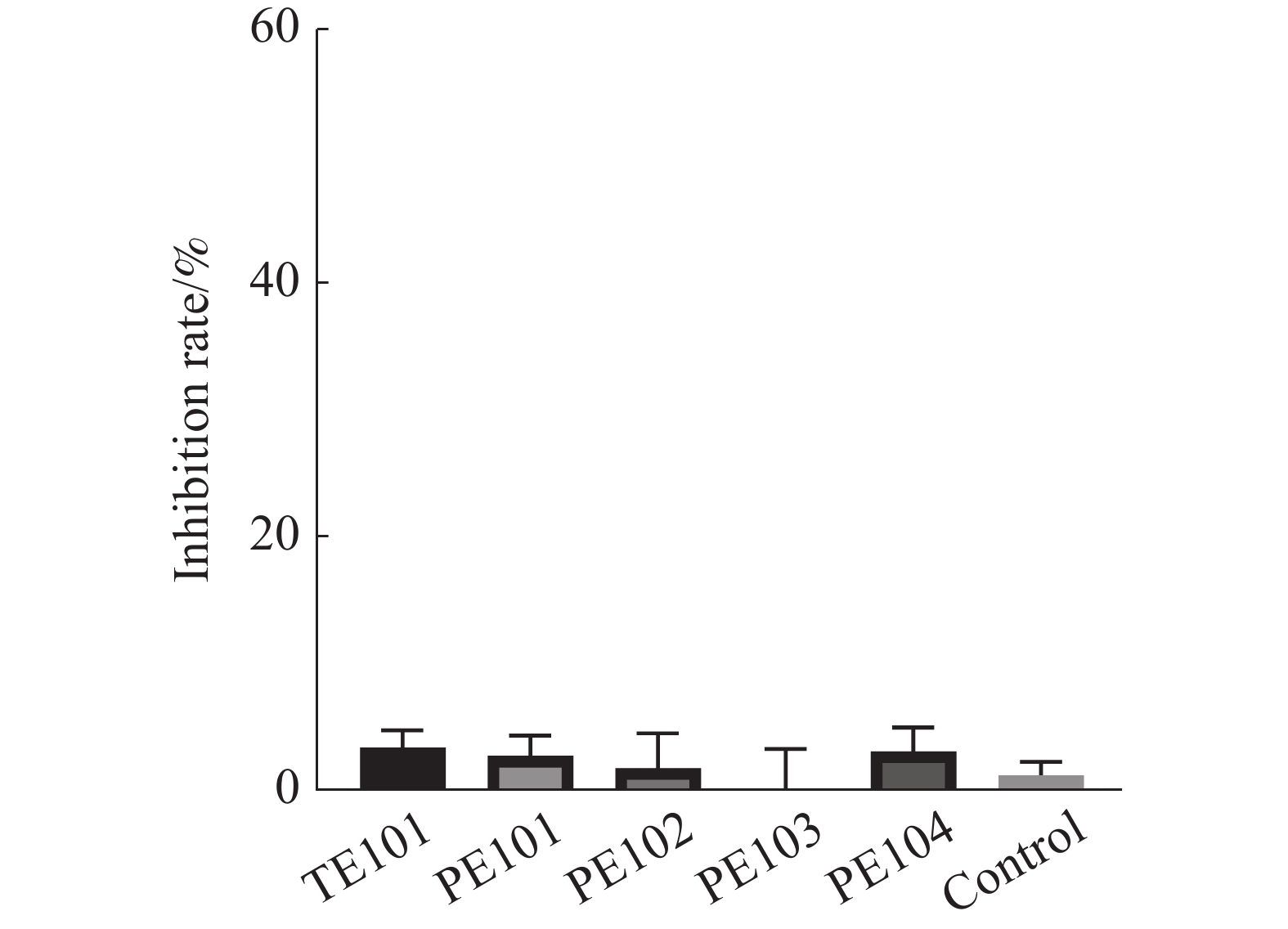
本研究通过测定多肽对于FtsZ的酶活性影响,对多肽的作用机制进行了初步探究,结果如图6所示,根据实验结果可知,多肽化合物对FtsZ的GTPase活性并没有影响。
-
在本研究中,针对金黄色葡萄球菌FtsZ蛋白的晶体结构(PDB Code:4DXD),优化处理后,以其PC口袋为结合位点,构建二肽、三肽、四肽、五肽库作为配体库,对数十万个多肽进行了虚拟筛选,根据对接打分表排序选择了分值靠前的10个多肽(二肽D101、D102,三肽TR101、TR102,四肽为TE101、TE102,五肽为PE101、PE102、PE103、PE104),在此基础上,进一步利用GROMAC程序进行分子动力学研究验证其对接结合可靠性和稳定性,通过计算比较它们在分子动力学过程中的配体RMSD值变化、配体−蛋白相互作用能的高低,进一步筛选出结合能力更好的多肽。RMSD结果表明,二肽、三肽不能很好的在PC口袋中稳定存在,相较而言,在模拟过程中四肽TE101及4个五肽PE101、PE102、PE103、PE104,RMSD值稳定在0.2以下,说明其结合稳定性更佳;相互作用能的计算结果显示,以上结合稳定的5个多肽具有更低的相互作用能。综合RMSD、Interaction energy的分析结果,本研究初步命中了TE101、PE101、PE102、PE103、PE104进行研究,抑菌活性结果显示,命中多肽对金黄色葡萄球菌及枯草芽孢杆菌的生长确实存在抑制。
另外,实验证明分子对接筛选的FtsZ抑制剂并不影响FtsZ的GTPase,这与之前研究报道的苯甲酰胺类衍生物的作用机制类似[26],已报道的多肽类抑制剂中,MciZ通过靶向枯草芽孢杆菌FtsZ蛋白抑制其组装过程发挥抑菌作用[27-29];ADEPs降解FtsZ蛋白来阻碍Z环的形成进而发挥抑菌作用[30-31];Kil通过抑制FtsZ蛋白的GTPase活性进而影响Z环的形成对大肠杆菌产生抑制作用[32-33],所以笔者推测,命中多肽的作用机制可能是由于其较大的分子体积占据了狭缝,导致FtsZ蛋白二级结构无法在R态和T态之间转换[34],完成FtsZ单体与Z环的动态交换过程,导致细菌分裂过程异常,且分子对接结果也显示,几乎所有的短肽配体都能与FtsZ狭缝中的氨基酸形成大量氢键、离子键,这可能也是稳定其蛋白结构的因素之一。
新靶点抗菌药物的开发需要庞大的先导化合物库,本研究以短肽作为配体进行分子对接筛选,并对结果进行分子动力学分析,对先导化合物库的丰富具有积极作用。
Molecular docking, and molecular dynamic and antibacterial analyses of polypeptide inhibitors targeting FtsZ protein
-
摘要: 为了得到靶向FtsZ的潜在肽类抑制剂和对其作用机制进行探究,采用分子对接方法筛选靶向金黄色葡萄球菌FtsZ蛋白的肽类化合物配体,利用分子动力学方法分析筛选到的配体与FtsZ蛋白结合状态的动态变化,计算配体重原子位置均方根偏差(RMSD)和相互作用能(Interaction energy)。在分子动力学分析基础上,分析以上短肽配体的抗菌活性,测定其对GTP酶活性的影响。结果表明,TE101、PE101、PE102、PE103和PE104五个多肽对于金黄色葡萄球菌的生长有抑制作用,且该作用与FtsZ蛋白GTPase活性无关。Abstract: The abuse of antibiotics has led to the emergence of drug-resistant bacteria, and new targets of antibiotics need to be developed urgently. FtsZ protein is a key protein in bacterial cell division, and inhibition of its dynamic process can lead to abnormal bacterial cell division and inhibition. In order to find bacterial inhibitors with new mechanisms of action, we conducted a virtual peptide screening by targeting the FtsZ protein of Staphylococcus aureus, and calculated the root mean square deviation (RMSD) and interaction energy of the screened peptides for evaluation. We investigated their binding stability, tested the antibacterial activity of the hit peptides, and measured their effects on GTPase to explore their mechanism of action. The screened five peptides, TE101, PE101, PE102, PE103 and PE104, were tested in combination with the results of molecular docking and molecular dynamics analysis. The results showed that these five peptides inhibited the growth of S. aureus, but did not act by affecting the GTPase of FtsZ protein.
-
Key words:
- FtsZ /
- molecular docking /
- molecular dynamic /
- antibacterial activities /
- GTPase
-
表 1 对接结果数据
配体 S Rmsd-refine E-conf E-place E-score1 E-refine D101 −7.7623 1.9002 −430.7451 −62.9163 −7.1529 −36.6142 D102 −8.0550 1.5846 −237.1169 −70.7354 −9.6892 −33.6909 TR101 −9.4474 2.6825 −536.4507 −52.6038 −6.8441 −50.7704 TR102 −8.9825 1.8490 −371.1595 −72.7251 −6.7891 −46.7827 TE101 −9.3997 3.2524 −465.5232 −88.2899 −7.7524 −51.8134 TE102 −9.6111 1.7341 −498.0332 −87.8680 −6.6491 −39.1319 PE101 −11.9339 3.3185 −400.3169 −25.8219 −2.1784 −58.5257 PE102 −11.0793 2.8318 −572.0369 −43.4562 −0.8752 −49.9537 PE103 −10.0457 2.9602 −487.5392 −27.8190 −4.2072 −63.7616 PE104 −10.2516 2.6604 −373.5583 −42.5808 −5.0850 −61.7718 注:S值是对接的最终得分,Rmsd-refine表示对接之前的姿势与对接之后的姿势之间的均方根偏差细化,E-conf表示构象异构体的能量,默认情况下,此能量通过将溶剂化选项设置为Born来计算。E-place表示空间适配度的打分,E-score1表示第一次评分,E-refine是范德瓦尔斯静电和溶解能之和。 表 2 小分子和蛋白的氢键相互作用
配体 受体 相互作用 原子距离/Å 能量 /( kcal·moL-1) D101 N1 OG1 THR309 H-donor 2.91 −6.6 NE2 O LEU200 H-donor 2.86 −4.1 NE2 OD1 ASN208 H-donor 3.06 −4.0 OC1 OG1 THR265 H-acceptor 2.68 −1.8 D102 OC2 ND2 ASN263 H- acceptor 3.52 −0.5 OC2 OG1 THR265 H- acceptor 2.60 −2.5 TR101 NE2 O LEU200 H-donor 2.92 −3.1 NE2 O LEU209 H-donor 2.96 −0.5 N O GLY196 H-donor 3.21 −1.7 NE O GLN192 H-donor 3.29 −1.5 NH1 O GLY227 H-donor 2.87 −4.9 OE1 N LEU209 H-acceptor 3.17 −1.5 O ND2 ASN263 H-acceptor 2.72 −2.6 OC1 OG1 THR309 H-acceptor 2.54 −2.8 TR102 NE OD2 ASP199 H-donor 2.86 −8 NH2 OD2 ASP199 H-donor 2.69 −8.5 OD2 OG1 THR265 H-acceptor 2.6 −3.5 SD CA GLY193 H-acceptor 3.76 −1 NE OD2 ASP199 Ionic 2.86 −5.5 NH2 OD2 ASP199 Ionic 2.69 −7 TE101 N1 OD2 ASP199 H-donor 2.68 −8.0 OD1 CG GLN195 H- acceptor 2.80 −2.7 NH2 OG1 THR265 H-donor 2.80 −2.7 O2 CB GLN192 H-acceptor 2.82 −0.5 N1 OD2 ASP199 Ionic 2.79 −7.0 OC1 OG1 THR265 H-donor 2.67 −3.5 TE102 O2 ND2 ASN299 H-acceptor 2.87 −1.1 O5 ND2 ASN299 H-acceptor 3.15 −0.6 PE101 N1 O GLY227 H-donor 2.88 −5.1 OE1 NE2 GLN192 H-acceptor 2.92 −4.9 O3 ND2 ASN263 H-acceptor 2.73 −2.9 PE102 NH1 O VAL310 H-donor 3.06 −2.3 N6 OD2 ASP199 H-donor 2.82 −7.3 N8 OD2 ASP199 H-donor 3.34 −1.4 O6 OG1 THR265 H-acceptor 2.95 −2 O8 ND2 ASN299 H-acceptor 2.94 −2.3 OC1 ND2 ASN299 H-acceptor 2.74 −2.7 PE103 N1 OG LY196 H-donor 2.83 −4.5 CA5 OD2 ASP199 H-donor 3.4 −0.7 OD1 CB THR309 H-acceptor 3.32 −0.8 O1 OG1 THR309 H-acceptor 2.65 −1.4 PE104 N1 O GLY196 H-donor 2.96 −3.4 N2 OG1 THR265 H-donor 3.21 −1.3 NE3 OD1 ASP199 H-donor 3.02 −3.7 NH2 OD1 ASP199 H-donor 2.82 −9.8 O1 OG1 THR309 H-acceptor 2.55 −1.6 NE3 OD1 ASP199 Ionic 3.02 −4.3 NH2 OD1 ASP199 Ionic 2.82 −5.8 注:配体一列表示多肽小分子配体中与FtsZ蛋白形成氢键的具体原子;受体一列表示FtsZ蛋白中与小分子配体形成氢键的具体氨基酸及特定原子;相互作用表示其作用类型;原子距离表示形成氢键的两原子之间的距离长短;能量表示该氢键的能量高低。 -
[1] 郑惊雷, 梁力建, 彭宝岗, 等. 3种常用抗生素在胆汁中对胆道致病菌的杀菌效果[J]. 中国现代医学杂志, 2007(4): 487 − 490. doi: 10.3969/j.issn.1005-8982.2007.04.030 [2] ARDAL C, BALASEGARAM M, LAXMINARAYAN R, et al. Antibiotic development - economic, regulatory and societal challenges [J]. Nature Reviews Microbiology, 2020, 18(5): 267 − 274. doi: 10.1038/s41579-019-0293-3 [3] SAMOILOVICH N N. Development of staphylococci resistant to antibiotics and its relation to the duration of antibiotic therapy [J]. Antibiotiki, 1961, 6(11): 270 − 276. [4] LOCK R L, HARRY E J. Cell-division inhibitors: new insights for future antibiotics [J]. Nature Reviews Drug Discovery, 2008, 7(4): 324 − 338. doi: 10.1038/nrd2510 [5] BRAMHILL D, THOMPSON C M. GTP-dependent polymerization of Escherichia coli FtsZ protein to form tubules [J]. Proc Natl Acad Sci U S A, 1994, 91(13): 5813 − 5817. doi: 10.1073/pnas.91.13.5813 [6] DE BOER P, CROSSLEY R, ROTHFIELD L. The essential bacterial cell-division protein FtsZ is a GTPase [J]. Nature, 1992, 359(11): 254 − 256. [7] MUKHERJEE A, DAI K, LUTKENHAUS J. Escherichia coli cell division protein FtsZ is a guanine nucleotide binding protein [J]. Proceedings of the National Academy of Sciences, 1993, 90(3): 1053 − 1057. doi: 10.1073/pnas.90.3.1053 [8] FUJITA J, SUGIYAMA S, TERAKADO H, et al. Dynamic assembly/disassembly of Staphylococcus aureus FtsZ visualized by high-speed atomic force microscopy [J]. Int J Mol Sci, 2021, 22(4): 1691 − 1697. doi: 10.3390/ijms22041691 [9] ADDINALL S G,LUTKENHAUS J. FtsA is localized to the septum in an FtsZ-dependent manner [J]. Bacteriol, 1996, 178: 7167 − 7172. doi: 10.1128/jb.178.24.7167-7172.1996 [10] GOEHRING N W, BECKWITH J. Diverse paths to midcell: assembly of the bacterial cell division machinery [J]. Curr Biol, 2005, 15(13): 514 − 526. doi: 10.1016/j.cub.2005.06.038 [11] MA S, MA S T. The Development of FtsZ inhibitors as potential antibacterial agents [J]. Chemmedchem, 2012, 7(7): 1161 − 1172. doi: 10.1002/cmdc.201200156 [12] SENGUPTA S, THOMAS S A. Drug target interaction of tubulin-binding drugs in cancer therapy [J]. Expert review of anticancer therapy, 2006, 6(10): 1433 − 1447. doi: 10.1586/14737140.6.10.1433 [13] WERBOVETZ K A. Tubulin as an antiprotozoal drug target [J]. Mini Reviews in Medicinal Chemistry, 2002, 2(6): 519 − 529. doi: 10.2174/1389557023405648 [14] TRIPATHY S, SAHU S K. FtsZ inhibitors as a new genera of antibacterial agents [J]. Bioorganic Chemistry, 2019, 91(24): 332 − 344. [15] OHASHI Y, CHIJIIWA Y, SUZUKI K, et al. The lethal effect of a benzamide derivative, 3-methoxybenzamide, can be suppressed by mutations within a cell division gene, FtsZ, in Bacillus subtilis [J]. Journal of Bacteriology, 1999, 181(4): 1348 − 1351. doi: 10.1128/JB.181.4.1348-1351.1999 [16] HAYDON D J, STOKES N R, URE R, et al. An inhibitor of FtsZ with potent and selective anti-staphylococcal activity [J]. Science, 2008, 321(5896): 1673 − 1675. doi: 10.1126/science.1159961 [17] MATHEW B, ROSS L, REYNOLDS R C. A novel quinoline derivative that inhibits mycobacterial FtsZ [J]. Tuberculosis, 2013, 93(4): 398 − 400. doi: 10.1016/j.tube.2013.04.002 [18] LI D, CHI B, WANG W W, et al. Exploring the possible binding mode of trisubstituted benzimidazoles analogues in silico for novel drug design targeting Mtb FtsZ [J]. Medicinal Chemistry Research, 2017, 26(1): 153 − 169. doi: 10.1007/s00044-016-1734-4 [19] PARK B, KUMAR K, AWASTHI D, et al. Synthesis and evaluation of novel trisubstituted benzimidazoles targeting FtsZ as antimicrobial agents [J]. Abstracts of Papers of the American Chemical Society, 2012, 243(55): 457 − 471. [20] TAN C M, THERIEN A G, LU J, et al. Restoring methicillin-resistant Staphylococcus aureus susceptibility to beta-lactam antibiotics [J]. Sci Transl Med, 2012, 4(126): 126 − 135. [21] SUN Y, WANG B C, PEI J L, et al. Molecular dynamic and pharmacological studies on protein-engineered hirudin variants of Hirudinaria manillensis and Hirudo medicinalis [J]. Brit J Pharmacol, 2022, 179(14): 3740 − 3753. doi: 10.1111/bph.15816 [22] D'ANGELO E, CRUTCHFIELD J, VANDIVIERE M. Rapid, sensitive, microscale determination of phosphate in water and soil [J]. Journal of Environmental Quality, 2001, 30(6): 2206 − 2209. doi: 10.2134/jeq2001.2206 [23] O'TOOLE M, LAU K T, SHEPHERD R, et al. Determination of phosphate using a highly sensitive paired emitter-detector diode photometric flow detector [J]. Analytica Chimica Acta, 2007, 597(2): 290 − 294. doi: 10.1016/j.aca.2007.06.048 [24] HALGREN T A, MURPHY R B, FRIESNER R A, et al. Glide: A new approach for rapid, accurate docking and scoring. 2. Enrichment factors in database screening [J]. Journal of Medicinal Chemistry, 2004(6): 1750 − 1759. [25] VORONTSOV I I, MIYASHITA O. Crystal molecular dynamics simulations to speed up MM/PB(GB)SA evaluation of binding free energies of di‐mannose deoxy analogs with P51G‐m4‐Cyanovirin‐N [J]. Journal of Computational Chemistry, 2015, 32(6): 1043 − 1053. [26] SUN N, DU R L, ZHENG Y Y, et al. Antibacterial activity of N-methylbenzofuro[3, 2-b]quinoline and N-methylbenzoindolo[3, 2-b]-quinoline derivatives and study of their mode of action [J]. European Journal of Medicinal Chemistry, 2017, 135(78): 1 − 11. [27] ARAUJO-BAZAN L, RUIZ-AVILA L B, ANDREU D, et al. Cytological profile of antibacterial FtsZ inhibitors and synthetic peptide MciZ [J]. Front Microbiol, 2016, 7: 1558 − 1575. [28] HANDLER A A, LIM J E, LOSICK R. Peptide inhibitor of cytokinesis during sporulation in Bacillus subtilis [J]. Mol Microbiol, 2008, 68(3): 588 − 599. doi: 10.1111/j.1365-2958.2008.06173.x [29] RAY S, KUMAR A, PANDA D. GTP regulates the interaction between MciZ and FtsZ: A possible role of MciZ in bacterial cell division [J]. Biochemistry-Us, 2013, 52(2): 392 − 401. doi: 10.1021/bi301237m [30] BROTZ-OESTERHELT H, BEYER D, KROLL H P, et al. Dysregulation of bacterial proteolytic machinery by a new class of antibiotics [J]. Nature medicine, 2005, 11(10): 1082 − 1087. doi: 10.1038/nm1306 [31] SASS P, JOSTEN M, FAMULLA K, et al. Antibiotic acyldepsipeptides activate ClpP peptidase to degrade the cell division protein FtsZ [J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(42): 17474 − 17479. doi: 10.1073/pnas.1110385108 [32] HAEUSSER D P, HOASHI M, WEAVER A, et al. The Kil peptide of bacteriophage lambda blocks Escherichia coli cytokinesis via ZipA-dependent inhibition of FtsZ assembly [J]. PLoS Genet, 2014, 10(3): 1004217 − 1004229. doi: 10.1371/journal.pgen.1004217 [33] CHEN Y, MILAM S L, ERICKSON H P. SulA inhibits assembly of FtsZ by a simple sequestration mechanism [J]. Biochemistry-Us, 2012, 51(14): 3100 − 3109. doi: 10.1021/bi201669d [34] WAGSTAFF J M, TSIM M, OLIVA M A, et al. A Polymerization-associated structural switch in FtsZ that enables treadmilling of model filaments [J]. Mbio, 2017, 8(3): 656 − 677. -




 下载:
下载: