-
文心兰(Oncidium hybridum)为多年生草本,其生长史从幼苗期发育到假鳞茎发育再到假鳞茎成熟萌发不定芽阶段,紧接抽苔阶段,由不定芽发育成花芽并分化成花序和各花器官直至开花阶段,待花衰败后,由基部的不定芽萌发阶段过渡到发育成营养芽阶段(图1)[1 − 2]。前3个阶段(i~iii)为营养生长阶段,最后阶段(vi)的营养芽会继续发育成另一个新植株。若在假鳞茎成熟阶段(iii)时不定芽分化出的不是花芽,而直接跳跃发育到营养芽阶段(vi),即为“跳芽现象”。
利用现代分子手段及生物信息分析方法研究关于兰花营养芽、花芽分化发育的基因表达调控途径越来越常见。在兰花快繁中,用转录测序初步确定与兰花芽分化相关的基因[3],蝴蝶兰(Phalaenopsis)腋芽增殖能力相关基因[4]。Li等[5]研究内源激素代谢和信号通路对蝴蝶兰腋芽发育至成花发育的分子调控机制。Fan等[6]认为杜鹃叶山茶(Camellia azalea)的花芽发育与植物激素途径和植物代谢等多种途径调控有关。在百子莲(Agapanthus praecox ssp.orientalis)的成花过程生长素信号发挥着重要调控作用[7],也有研究发现鳄梨(Persea americana) HFL 在芽中产生生长素信号,诱导花抑制因子 PaTFL1,从而抑制花序发育[8]。除了生长素、细胞分裂素对花芽分化过程的调控研究外[9],在兰花中,开花启动子相关基因能促进开花转变,而抑制开花启动的相关基因则以拮抗的形式抑制开花[10 − 11]。而在文心兰芽分化研究中,假鳞茎内糖类物质的变化对子代假鳞茎侧芽的萌发影响很大,假鳞茎成熟阶段淀粉会大量转化为供应芽萌发的能量[12]。
文心兰作为一种有着较高观赏价值的兰花切花和盆花,具有重要的市场价值和潜力。海南省气候资源优越,加上政策支持、技术加持等条件,为兰花产业的发展增添了强劲助力。在海南博大兰花科技有限公司东山兰花基地的文心兰切花生产过程中发现了文心兰的几个品种(‘博大一号’‘柠檬绿’)中均出现“跳芽”现象,该现象发生过多会导致一整个产花周期无花可产,总体切花产量下降,而在相同条件下其他品种的文心兰却少有“跳芽”现象。在触发新生营养芽和花芽分化过程中,因某些开花相关基因受到抑制或延迟[13],可能导致花芽分化失败,直接进入营养芽阶段;或营养生长相关基因被过度激活,而直接促进营养芽发育[14];又或者复杂的激素信号通路、基因网络调控也会打破营养生长与生殖生长的平衡[15],导致该现象的发生。目前,虽有前人指出文心兰“跳芽”的现象,但并未将“跳芽”现象进行概念化归纳,以及从生产实际中探索解决“跳芽”现象的方法仍很少,对于文心兰花芽/营养芽分化的分子机制少见报道。
本研究探究文心兰花芽及营养芽分化的分子机制,以期初步了解调控文心兰花芽和营养芽生长发育的关键因素,为减少文心兰切花生产过程中不定芽跳跃性发育现象提供理论基础,以提高文心兰生产效率和质量、优化文心兰栽培管理技术,对推动我国兰花育种、种苗生产和产业发展具有十分重要的现实意义。
-
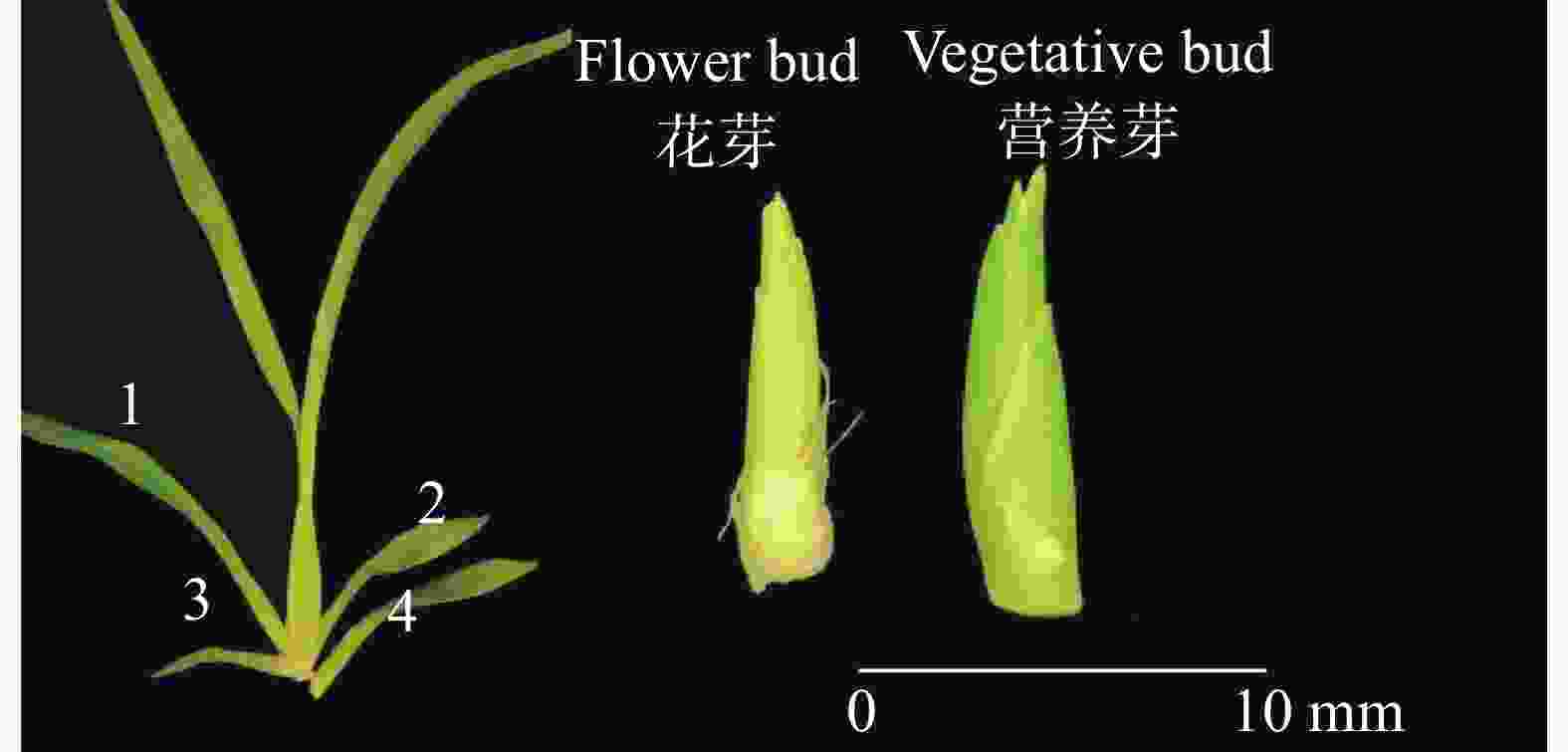
检测样品从基地同一批次生长健壮的文心兰‘博大一号’(Oncidium hybridum 'Boda NO1')植株中随机选择处于抽苔阶段的当年生假鳞茎基部第1或2子叶内长出约10 mm花芽的植株共3株;随机选择处于营养芽阶段的当年生假鳞茎基部第3、4子叶内长出约10 mm营养芽的植株共3株,取其花芽、营养芽(图2),剥去叶(鞘)片,称取约2~3 g,放入冻存管中,共计6个样,用于转录组测序实验。
-
首先,使用天根植物多糖多酚试剂盒提供的标准提取方法提取 RNA,然后对提取的 RNA 进行浓度、纯度、完整性检测,以确保其符合测序质量标准。其次,构建 cDNA 文库,并在 Illumina NovaSeq
6000 平台上对 cDNA 文库进行测序。最后,对转录组数据进行质量控制,以消除原始转录组数据中的冗余序列,从而获得高质量的纯净读数。 -
使用FASTP v0.19.3[16]对所有样本数据进行质量控制和优化评估(合格数据定义为 Q30 > 85%),而后将高质量序列与参考基因组进行序列比对,以营养芽为对照组,将差异倍数(Fold Change)≥2且PDR<0.05作为筛选标准。将筛选出的DEGs与GO、COG、KEGG等数据库进行序列比对,获得这些基因的注释信息。
-
提取转录组样本花芽、营养芽的RNA,检测合格的RNA用于后续实验。使用HiScript ll 1st Strand cDNA Synthesis Kit(+gDNA wiper)反转录试剂盒对RNA进行反转录,反转后的cDNA用于后续实验。使用Primer 6设计qRT-PCR特异性引物,引物序列见表1。使用并按照ChamQ SYBR qPCR Master Mix试剂盒说明书进行荧光定量PCR,选择Actin基因作为内参基因。数据结果分析使用2-^^CT法计算相对表达量。
基因
Genes正向引物(5′-3′)
Forward primers(5′-3′)反向引物(5′-3′)
Reverse primers(5′-3′)Actin GCAACATTGTTCTTAGCGGAGGCT TCTTCATGCTGCTTGGTGCAAGTG AUX/IAA22D AACAAAGCTCGACCTCCAAACG GTGTTGCGCTACAATGGTGTTG SAUR-6B ATGGGTTCATCGTCTCCGTCTC AACAGCACGATCCGCAGAACG MADS1 TGTGAGTGTGCTTGGTTGCTATC TTGATTGAAGACCCGAAAGATCCC PIF4 AGGAGACTATGAATCTGCCGATGG AAACTTGAACTTTGCTTACCCTTTCTG FLK GCAGAATCTACAGATGGAGCACAG GAGGCGGCACGTACTGAGG Table 1. The primer sequences of key genes.
-
纯化后的RNA经Agilent
2100 仪器检测,结果显示PIN值在8.4~9.5范围内,样品质量满足建库测序要求。通过清洗数据后,得到高质量序列127 452 717个,共37.36 Gb。各样品Q30均在94.62%以上,表明测序数据可靠,所构建文库符合要求,可用于后续分析。通过筛选得到7 671个DEGs,其中上调表达为3 695个,下调表达为3 976个。为更清晰地了解这些DEGs的情况,对其进行COG、GO、KEGG等数据库的功能注释。从表2中可知,共有7 420个DEGs被赋予注释信息,注释最多的数据库为eggNOG,为7 227个。注释较少的KEGG数据库注释到2 894个DEGs,COG数据库注释到2 662个。而一些未注释到的基因可能是未发现的新基因,也可能与参考基因组有关。
数据库
DatabaseDEGs注释总数
DEGs annotation totalCOG GO KEGG KOG NR Pfam Swiss-Prot eggNOG 数量
Number7 420 2 662 3 237 2 894 3 893 7 004 5 891 6 194 7 227 Table 2. Annotation and statistics of DEGs between vegetative buds and flower buds in Oncidium.
-
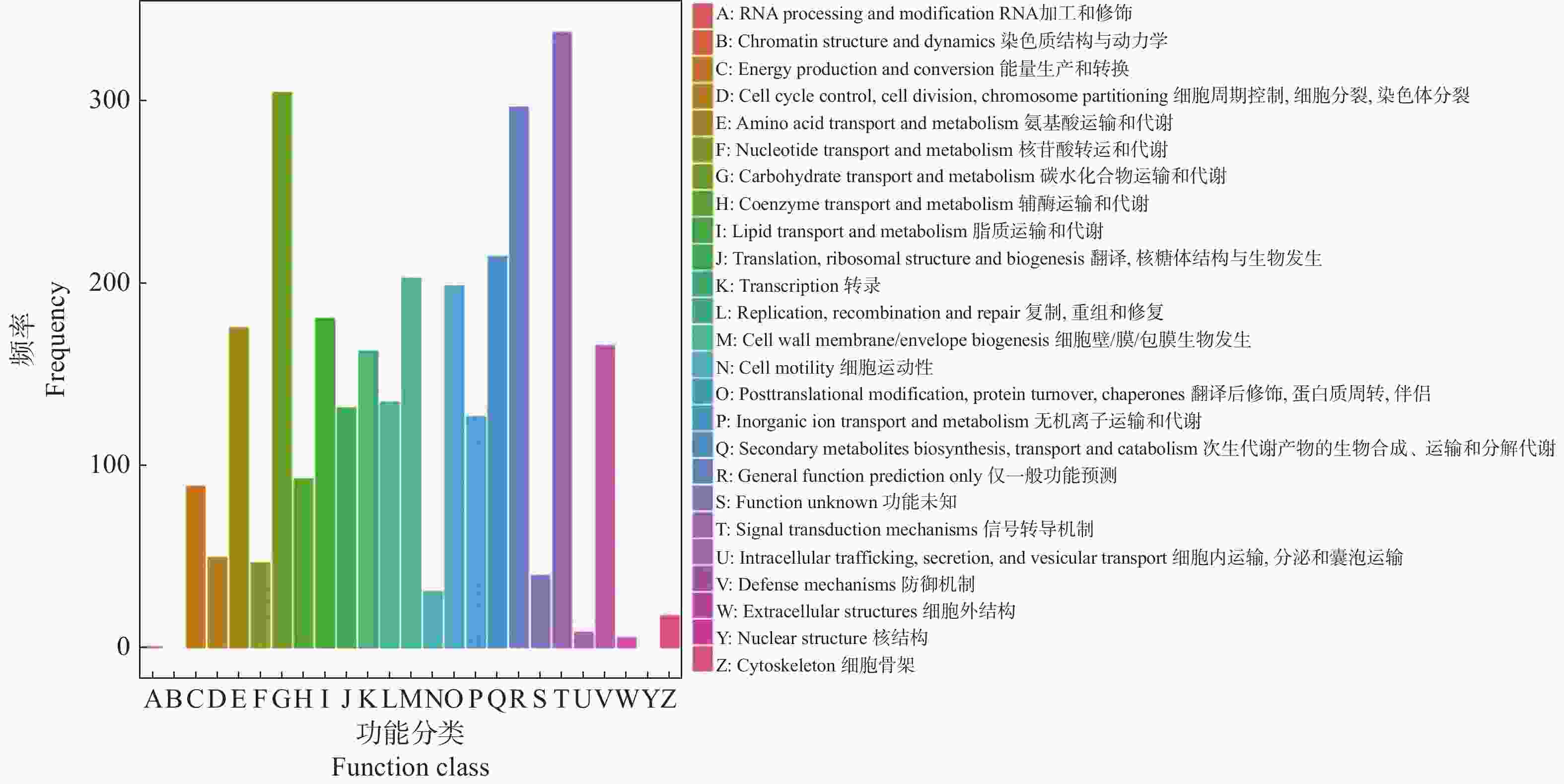
为进一步了解DEGs的直系同源信息,在COG数据库中(图3),DEGs分类到执行信号传导机制的数目最多,而后是碳水化合物运输和代谢,其次是一般功能预测和次生代谢产物的生物合成、运输和分解代谢。这些结果可以看出信号传导机制和碳水化合物是不定芽向花芽分化或营养芽分化的关键因素。
-
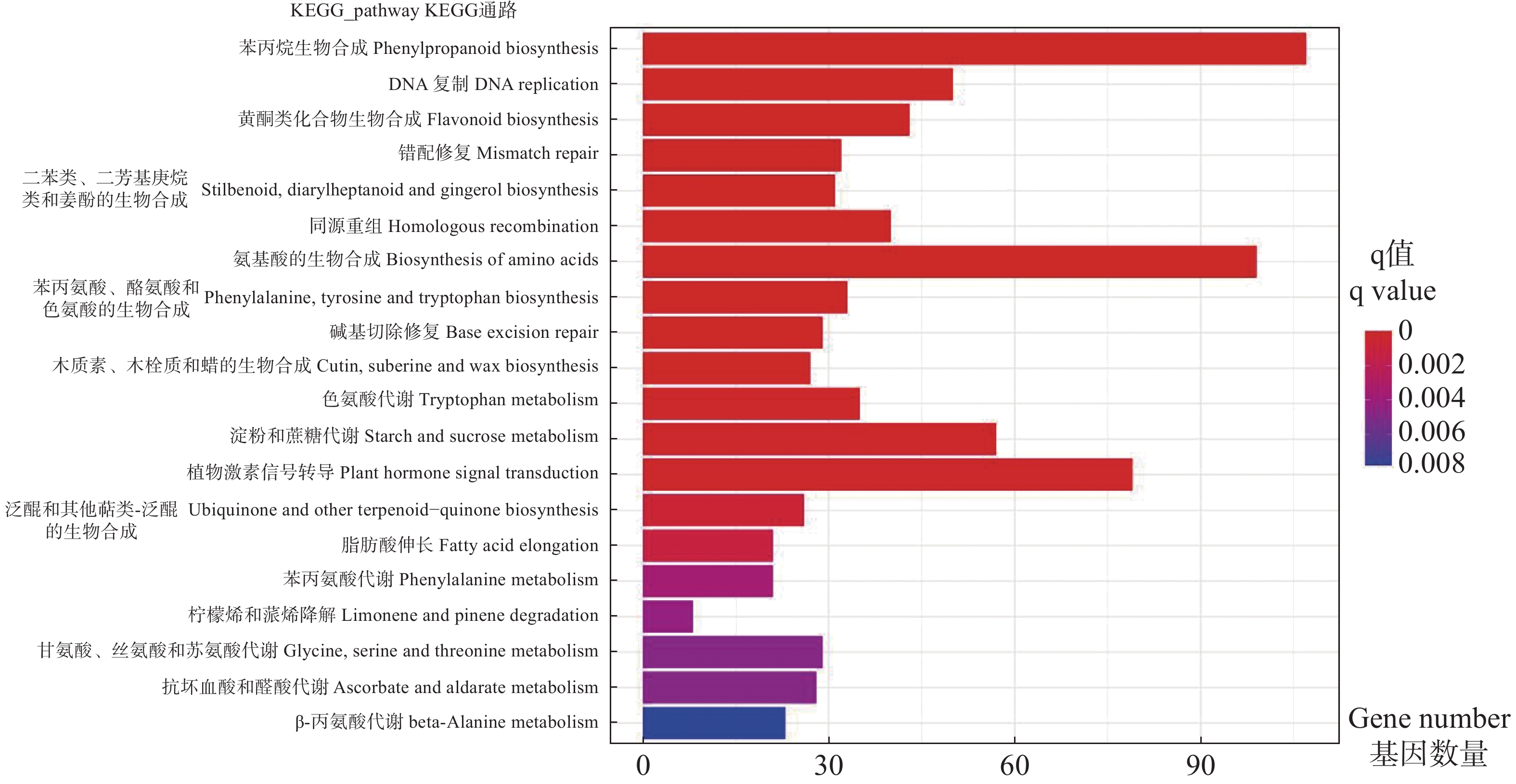
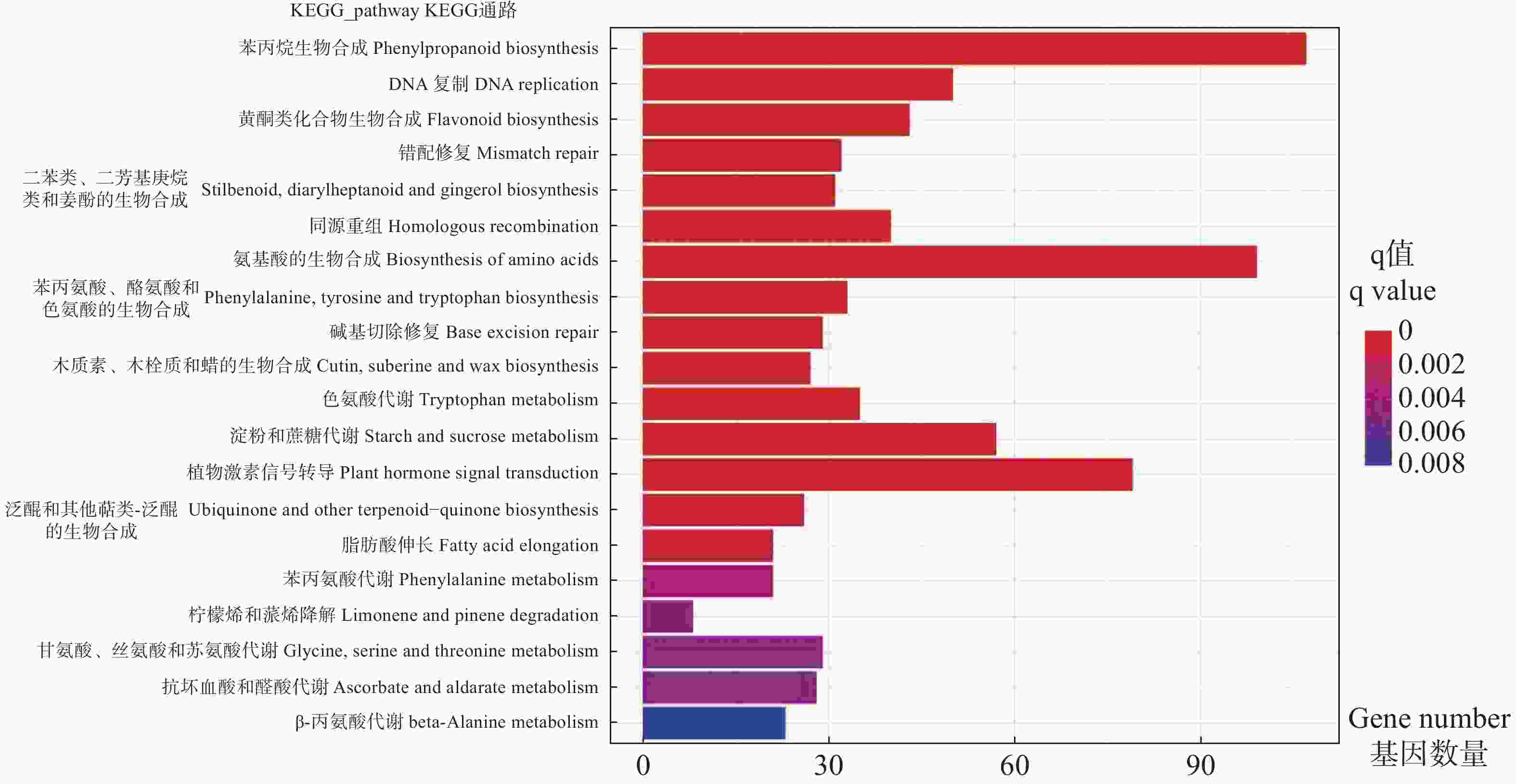
展示显著性q值最小的前20个通路,从图4可看出,DEGs显著富集在氨基酸生物合成途径、黄酮类化合物生物合成途径、苯丙烷等生物合成途径,淀粉和蔗糖、色氨酸等代谢途径和植物激素信号转导途径。其中,植物激素信号转导途径、氨基酸生物合成途径、苯丙烷生物合成途径富集的DEG数量最多。淀粉和蔗糖代谢、苯丙烷生物合成等途径参与植物次生代谢产物合成,色氨酸和植物激素信号转导途径等参与调控激素合成等植物生长发育代谢生理过程。
-
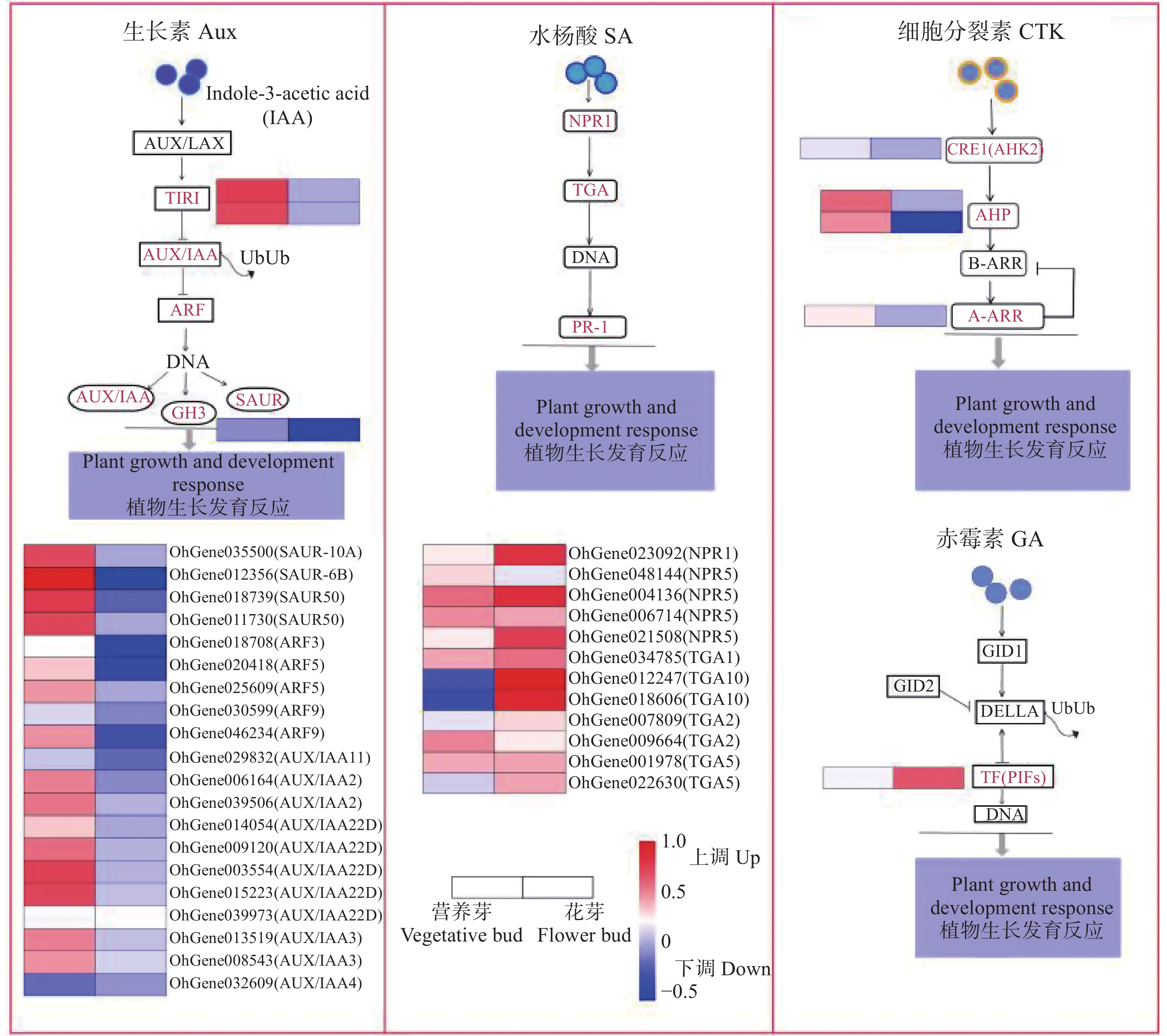
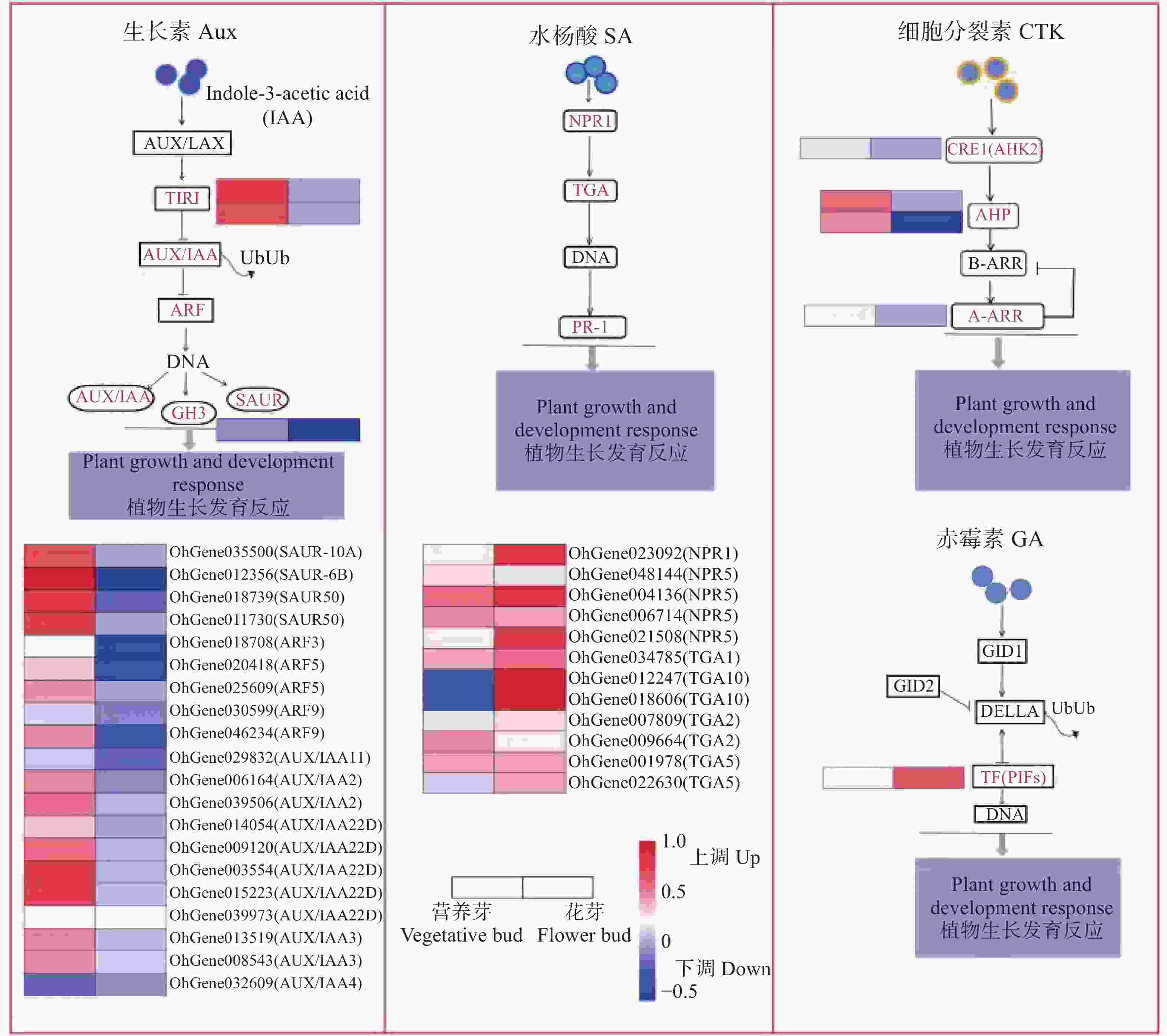
植物激素信号转导途径是环境信息处理功能中的重要途径,在植物激素信号转导途径中,共筛选出78个DEGs,其中上调表达有33个,涉及生长素(Aux)、赤霉素(GA)、油菜素甾醇(BR)、脱落酸(ABA)、水杨酸(SA)、茉莉酸(JA),而下调表达有45个,涉及Aux、GA、BR、ABA、SA、JA、细胞分裂素(CTK)。经分析,筛选出差异表达较为显著的4个信号通路(图5),在Aux信号转导途径中,TIR、AUX/IAA22D、SAUR基因及其同源基因表达量较高,其中后两者均上调表达,GH3基因也上调表达。在SA信号途径中,TGA10上调表达,NPRs和TGA2则上下调表达。在CTK信号转导途径中,AHP显著下调表达,赤霉素信号转导途径中仅有PIF4基因呈上调表达。
-
植物开花是一个多方因素参与调控的复杂过程,已有研究发现多种基因或转录因子与开花密切相关。本研究筛选出13个与之相关的基因及转录因子(表3),其中上调表达7个,下调表达6个。影响花发育的相关关键基因有APETALA2(AP2)、Flowering locus K homology domain isoform X1(FLK)同源基因均下调表达,Forever young flower protein及CAULIFLOWER/MADS-box、MADS1、MADS2等6个MADS-box基因家族则上调表达。这些转录因子和基因的表达对文心兰芽分化起重要调控作用。
DEGs 注释缩写
DEGs annotation abbreviation表达趋势
Expression trendsDEGs 注释缩写
DEGs annotation abbreviation表达趋势
Expression trendsDEGs 注释缩写
DEGs annotation abbreviation表达趋势
Expression trendsForever young flower protein 上up AP2 下down MADS 16 下down FLK 下down AP2 下down MADS 2 上up CAULIFLOWER/
MADS-box上up AP2 下down MADS1 上up CAULIFLOWER/
MADS-box上up MADS 2 上up MADS1 上up AP2 下down Table 3. Statistics of differentially expressed transcription factors and genes related to flowering
-
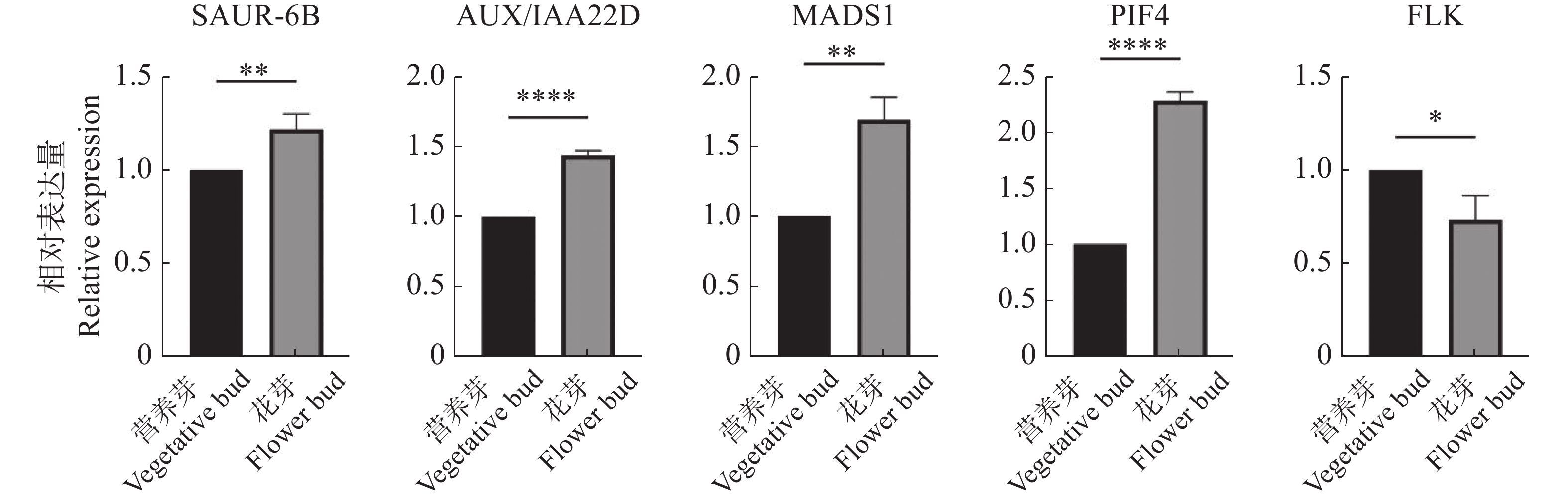
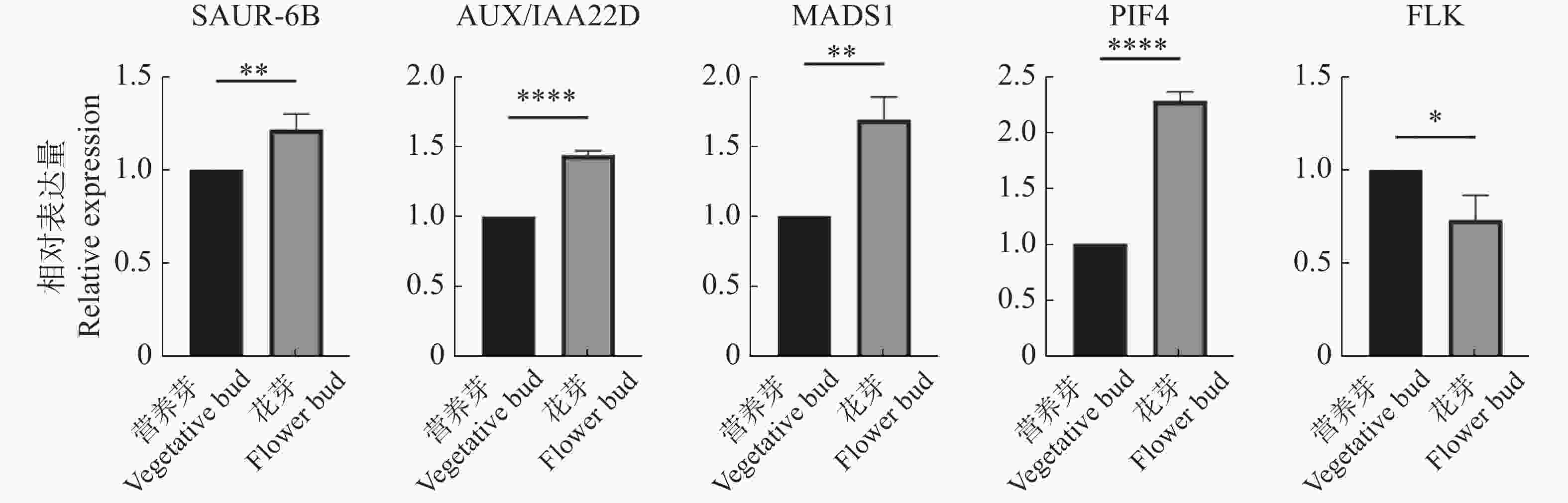
为进一步验证转录组数据的可用性,用qRT-PCR实验对五个关键基因OhGene009120(AUX/IAA22D)、OhGene011730(SAUR-6B)、OhGene004080(MADS1)、OhGene001497(PIF4)、OhGene015200(FLK)进行定量检测(图6),其变化趋势与转录组数据趋势一致。
-
芽分化是在环境条件和自身遗传等多种因素相互调控、共同作用下完成的。有研究表明,碳水化合物代谢和植物内源激素调控影响着花芽分化过程[17],在花魔芋(Amorphophallus konjac)的这一阶段过程中,糖代谢及内源激素扮演着至关重要的角色[18]。百子莲芽的KEGG通路富集在植物激素信号转导、淀粉和蔗糖代谢、苯丙烷生物合成途径上[7]。苯丙烷生物、黄酮类化合物生物是促进植物开花的次生活性代谢物质,这些合成途径在植物生长中发挥重要作用[19 − 20],本研究中均显著富集到这些途径,也与魔芋(Amorphophallus bulbifer)的花芽和叶芽KEGG富集研究结果一致[21]。花芽和营养芽分化在植物激素信号转导以及一些碳水化合物等代谢上有所差异,这与石长双[22]认为腋芽萌发与植物激素信号转导途径、糖类生物合成代谢途径有关的研究结果相近。
-
植物感受外部环境变化信号,通过植物激素信号转导途径调控多种激素的协同或拮抗作用以调整自身的生长和分化进程[23]。从营养生长到开花的过程,植物内源激素尤其重要。根据激素调节假说,Aux、CTK、GA等是营养生长转向生殖生长的重要调控因子[24]。AUX/IAA22D、SAUR、GH3同源基因均上调表达,而ARFs则多数下调表达,两者的相互作用可能影响生长素信号对芽分化的调控,推测花芽分化受其影响较大。TIR1与生长素分子结合,并通过调控生长素反应蛋白AUX/IAA、生长素反应因子ARF等的表达来调控植物生长,其信号的表达又受ARF-AUX/IAA的相互作用,在下游基因GH3/SAUR作用下与氨基酸结合,利于Aux向芽点运输,促进了生长素的转导和运输及其在芽中的响应作用,这些基因的表达水平可能更有利于茎尖分生组织内花芽分化的调节[25 − 27]。此外,本研究中的所有CTK信号均下调表达,说明CTK在对营养芽分化有正向调节作用,而在花芽分化中受限。CTK与Aux也能相互作用于植物生长,特别是芽的发育和分化,CTK信号促进生长素转运,从而引发自身的降解或糖基化[28],CTK调节分生组织活性,并在生殖阶段限制发育器官中的细胞形成[29]。赤霉素在芽发育过程中起着重要作用,于开花诱导的作用已经被前人在拟南芥上深入研究过。在本研究中,PIF4在花芽中高表达,PIF4可能是花芽分化的关键诱导基因,与PIF4可能是直接响应赤霉素促进蝴蝶兰成花的关键基因的结果一致[30]。TGA10为上调表达,在花芽分化中起重要作用。TGA1、TGA2、TGA5广泛参与植株的抗病反应,并与SA信号途径中关键因子NPR1相互作用,而TGA10对植物花器官发育起关键作用[31]。此外,ABA、JA等一些激素会通过生物钟和光周期相关的开花途径参与开花调节,以响应非生物胁迫[32 − 33]。
-
本研究中FLK、AP2基因均下调表达,推测可能是这些基因在文心兰花期抑制着花芽分化,而导致不定芽发育为营养芽。在拟南芥中,FLK影响FLC等开花基因的表达,延迟花芽分化[34]。AP2的过表达有时候会抑制开花[35],有时会直接参与侧芽的调控[36]。MADS1和MADS2在花芽中均高表达,对花芽发育分化起关键作用。MADS-box基因家族在花发育过程中起重要作用[37],MADS1能使转基因文心兰更早开花[38],来自兰花 Oncidium Gower Ramsey的MADS2能使转基因拟南芥提前或中度提前开花[39]。此外,还有Forever young flower protein、CAULIFLOWER/MADS-box也在花芽中高表达,这两种基因或许也促进花芽的发育,但仍需进一步实验验证。
-
本研究对文心兰‘博大一号’的花芽和营养芽转录测序分析,结果在一定程度上探析了生长素、细胞分裂素、水杨酸等激素及一些影响开花的相关基因MADS1、AP2、FLK等对文心兰花芽和营养芽分化的影响,为进一步深入研究文心兰生产过程中不定芽跳跃性发育现象形成的机制提供理论基础,便于提高文心兰生产效率和质量、优化文心兰栽培管理技术,对推动我国兰花产业发展具有十分重要的现实意义。
Transcriptome differences resolve the phenomenon of bud jumping in Oncidium and its regulation
DOI: 10.15886/j.cnki.rdswxb.20240197
- Received Date: 2024-12-24
- Accepted Date: 2025-03-21
- Rev Recd Date: 2025-03-03
-
Key words:
- Oncidium /
- transcription sequencing /
- flowering gene /
- flower bud differentiation /
- vegetative bud differentiation
Abstract: To address the decreased cut flower yield caused by the bud jumping development phenomenon, where adventitious buds interrupt the normal growth cycle of flower bud differentiation in Oncidium cut flower production, flower buds and vegetative buds of Oncidium hybridum 'Boda NO1' were selected for high-throughput transcriptome sequencing. A total of 127,452,717 high-quality sequences (37.36 Gb) were obtained, and 7,671 differentially expressed genes (DEGs) were identified. COG functional classification revealed primary enrichment in signal transduction pathways and carbohydrate transport/metabolism pathways. KEGG analysis showed that DEGs were significantly enriched in starch and sucrose metabolism, phytohormone signaling, and other pathways. Among these, 78 DEGs were identified in the phytohormone signaling pathway, with the most pronounced differences involving auxin, cytokinins, salicylic acid, and gibberellins. Thirteen transcription factors and flowering-related genes, including MADS1, AP2, and FLK, were also screened. These results partially elucidate the effects of auxin, cytokinins, and flowering-related genes on the differentiation of flower buds and vegetative buds in Oncidium. This study provides a theoretical foundation for further research on the mechanism underlying the bud jumping development phenomenon during Oncidium production. Additionally, it supports subsequent improvement in production efficiency, quality enhancement, and optimization of cultivation and management. These findings hold significant practical value for advancing orchid breeding, seedling production, and industry development in China.
| Citation: | LAN Hanqiao, LIU Le, LI Weishi, HAO Daicheng, CHEN Taizhen, LU Ling, TANG Minqiang, LING Peng. Transcriptome differences resolve the phenomenon of bud jumping in Oncidium and its regulation[J]. Journal of Tropical Biology. doi: 10.15886/j.cnki.rdswxb.20240197 |







 DownLoad:
DownLoad: